Python moduli koji se nalaze u direktorijumu /spark/jobs dizajnirani su kao Extract-Transform-Load/ETL analize. Dodatna podešavanja potrebna ovim analizama mogu se zadati iz datoteka pod /spark/config direktorijumom. Moduli koji su neophodni za izvršavanje analiza mogu se pronaći u /spark/dependencies direktorijumu. Ovi moduli obuhvataju postavke sesije, proširenja evidentiranja (engl. logging) kao i apstrakcije oko keras biblioteke.
Front-End koji se koristi za vizualizaciju analiza nalazu se u direktorijumu /visualization. Odvojeni Python moduli za pokretanje web-servera nalaze se pod /visualization/scripts i podeljeni su po izvorima podataka.
root/
|-- spark/
|---- configs/
| |-- cases_time_analysis.json
| |-- radiography_analysis.json
| |-- research_challenge_analysis.json
| |-- cases_clinical_spectrum_analysis.json
|---- dependencies/
| |-- spark.py
| |-- keras.py
| |-- logging.py
| |-- textblob.py
|---- jobs/
| |-- cases_time_analysis.py
| |-- radiography_analysis.py
| |-- research_challenge_analysis.py
| |-- cases_clinical_spectrum_analysis.py
| build_dependencies.sh
| packages.zip
| Pipfile
| Pipfile.lock
| .env
|-- visualization/
|---- meta/
|---- dependencies/
|---- scripts/Arhitektura projekta bazirana je na takozvanim Extract-Transform-Load (ETL) poslovima. Zbog lakšeg izvršavanja i testiranja, korak transformacije izolovan je od ekstrakcije i učitavanja. Ulazni podaci prihvataju se i pakuju u jedinstveni DataFrame. Zatim, kod koji obuhvata transformacije bavi se izvlačenjem podataka, daljim prosleđivanjem funkciji transformacije, kao i čuvanjem rezultata.
Generalizovano, funkcije transformacije bi trebalo dizajnirati kao idempotent funkcije. Drugim rečima, višestruko primenjianje funkcija transformacije ne bi trebalo da rezultuje promenama u izlazu, sve dok nema promena ulaznih podataka. Zbog ovakvog pristupa moguće je izvršavati analize sa ponavljanjima ukoliko je to potrebno (na primer, koristeći cron za poziv spark-submit komande, po pre-definisanom rasporedu poziva).
Transformacije predstavljaju pozive transformacionih funkcija Sparka gde svaki poziv rezultira formiranjem nove distribuirane strukture. Učitavanje, na kraju, svodi se na snimanje konačne strukture u celini.
Kako se ne bi slali argumenti sa komandne linije, efiaksnije rešenje je koristiti konfiguracione datoteke po potrebi - na primer, koristeći --files configs/job_name_config.json flag sa spark-submit komandom - flag koji će referencirati konfiguracionu datoteku. Ove datoteke mogu se koristiti u analizama u vidu rečnika, iliti dictionary ulaza kao json.loads(config_file_contents).
import json
config = json.loads("""{"field": "value"}""")Datoteka se učitava i parsuje funkcijom start_spark() iz pomoćne datoteke dependencies/spark.py koja pored parsovanja konfiguracije učitava Spark driver proces koji se pokreće na klasteru i alocira loger.
Deljene funkcije na koje se oslanjaju analize nalaze se u paketu dependencies i referenciraju module Spark-a koji su neophodni, na primer:
from dependencies import start_sparkOvaj paket, zajedno sa svim ostalim dependency-ma, mora biti kopiran na svaki Spark čvor. Postoji više načina za postizanje ovoga, izabrano je pakovanje svih zavisnosti u zip arhivu zajedno sa analizom koju treba izvršiti. Zatim se koristi --py-files naredba prilikom pokretanja analize. Pomoćna shell skripta build_dependencies.sh koristi se za pakovanje arhive. Ova skripta uzima u obzir graf zavisnosti okruženja i sve navedene zavisnosti u Pipfile datoteci.
Lokalno pokretanje klastera:
$ cd $SPARK_HOME && spark-shell --master localUkoliko $SPARK_HOME promenljiva okruženja ukazuje na instalaciju Spark-a, analiza pokreće se kao:
$SPARK_HOME/bin/spark-submit \
--master local \
--packages 'com.somesparkjar.dependency:1.0.0' \
--py-files packages.zip \
--files configs/job_name_config.json \
jobs/job_name.py \
hdfs://data/data_dir/--master local[*]- adresa Spark klastera. Ovo može biti lokalni klaster ili klaster u cloud-u koji se zadaje adresomspark://adresa_klastera:7077;--packages 'com.somesparkjar.dependency:1.0.0,...'- Maven dependency lista;--files configs/job_name_config.json- putanja do konfiguracione datoteke;--py-files packages.zip- prethodno pomenuta arhiva sa dependency-ma;jobs/job_name.py- Python modul sa kodom analize.hdfs://data/data_dir/- putanja do ulaznog dataseta na HDFS-u.
Početna tačka svake Spark analize je otvaranje sesije. Ovo je driver proces koji održava sve relevantne informacije i odgovoran je za distribuiranje i zakazivanje rada nad svim executor procesima.
def start_spark(app_name='my_spark_app', master='local[*]',
jar_packages=[], files=[], spark_config={}):
flag_repl = not (hasattr(__main__, '__file__'))
flag_debug = False
if not (flag_repl or flag_debug):
spark_builder = (
SparkSession
.builder
.appName(app_name))
else:
spark_builder = (
SparkSession
.builder
.master(master)
.appName(app_name))Pregled ETL obrade:
def main():
spark, sql_context, log, config = start_spark(
app_name='cases_time_analysis',
files=['configs/cases_time_analysis_config.json'])
log.warn('Running cases_time analysis...')
# extracting and transforming the dataset
data = extract_data(spark)
data_transformed = transform_data(data, sql_context)
# confirmed cases and deaths globally
data_transformed = transform_confirmed_cases(data_transformed)
load_data(data_transformed, "confirmed_cases_and_deaths_globally")Analize se mogu izvršavati na klasteru računara koji je realizovan Docker kontejnerima.
Korišćeni su bde-spark imidži - više o njima može se videti ovde. Više o postavci kontejnera i klasteru je u direktorijumu /bde-cluster. Klaster se može simulirati lokalno gde se pokreće infrastruktura čvorova nad HDFS-om. Tehnički, poziva se spark-submit komanda i spark-master čvoru šalju analize za izvršenje. Procedura slanja analize je pritom uopštena docker kontejnerom u kome se izvršava.
Infrastruktura je opisana odvojenom komozicijom i sadrži više servisa, kao na primer namenode, datanote i historyserver Hadoop deamone. Potrebno je podići infrastrukturu, a zatim postaviti izvore podataka na HDFS. Nakon ovoga, može se pokrenuti kontejner za izdavanje poslova koji će spark-submit naredbom proslediti Python modul.
Neophodno je da svi definisani infrastructure/submit kontejneri imaju pristup deljenoj mreži, u ovom slučaju mreža je bde.
$ docker network create bde$ cd infrastructure && ./infra_start.sh && ./infra_upload_to_hdfs.sh$ cd .. && ./analysis_start.sh
Infrastruktura sadrži potrebne image, ovo podrazumeva neophodne Hadoop deamone, kao i master/worker čvorove.
services:
pyspark-master:
image: bde2020/spark-master:3.1.1-hadoop3.2
container_name: pyspark-master
...
pyspark-worker-1:
image: bde2020/spark-worker:3.1.1-hadoop3.2
container_name: pyspark-worker-1
...
namenode:
image: bde2020/hadoop-namenode:2.0.0-hadoop3.2.1-java8
container_name: namenode
...
datanode:
image: bde2020/hadoop-datanode:2.0.0-hadoop3.2.1-java8
container_name: datanode
...
resourcemanager:
image: bde2020/hadoop-resourcemanager:2.0.0-hadoop3.2.1-java8
container_name: resourcemanager
...
nodemanager1:
image: bde2020/hadoop-nodemanager:2.0.0-hadoop3.2.1-java8
container_name: nodemanager
...
historyserver:
image: bde2020/hadoop-historyserver:2.0.0-hadoop3.2.1-java8
container_name: historyserver
...
volumes:
hadoop_namenode:
hadoop_datanode:
hadoop_historyserver:
networks:
default:
external:
name: bdeOsnovni image je bde2020/spark-submit.
Odavde se poziva spark-submit komanda i Python izvršni modul šalje ka klasteru. Na početku je potrebno dodati zavisnosti i alate za kompajliranje .c slojeva biblioteka (poput gcc kompajlera). U ovom slučaju, analize se zbog algoritama mašinskog učenja oslanjaju na numpy biblioteku.
Zatim se na osnovu niza zavisnosti pribavljaju potrebni izvori i razrešavaju zavisnosti. Na kraju, poziva se submit naredba sa modulom /app/app.py. Ukoliko je neophodno mogu se definisati promenljive okruženja poput HDFS_ROOT i HDFS_DATASET_PATH.
FROM bde2020/spark-submit:3.1.1-hadoop3.2
# Add build dependencies for c-libraries (important for building numpy and other sci-libs)
RUN apk --no-cache add --virtual build-deps musl-dev linux-headers g++ gcc python3-dev
# Copy the requirements.txt first, for separate dependency resolving and downloading
COPY app/requirements.txt /app/
RUN cd /app \ && pip3 install -r requirements.txt
# Copy the source code
COPY app /app
ADD app/start.sh /
RUN chmod +x /start_app.sh
ENV SPARK_MASTER spark://spark-master:7077
ENV SPARK_APPLICATION_PYTHON_LOCATION app/app.py
ENV SPARK_SUBMIT_ARGS "--total-executor-cores 80 --executor-memory 16g --executor-cores 8"
ENV HDFS_ROOT hdfs://namenode:9000
ENV HDFS_DATASET_PATH /data/data/
CMD ["/bin/bash", "/start_app.sh"]Na osnovu prethodnog Docerfile-a podiže se kontejner za predavanje poslova.
version: "3"
services:
submit:
build: ./
image: spark-submit:latest
container_name: analysis
environment:
...
networks:
default:
external:
name: bdeU nastavku su ukratko opisani korišćeni izvori podataka, zatim će biti obrađena većina izvršenih analiza.
Odabrana su četiri različita izvora podataka o ovom virusu:
- COVID-19 Radiography Database @Kaggle
- COVID-19 Open Research Dataset Challenge @Kaggle
- COVID-19 Dataset @Kaggle
- Diagnosis of COVIDand its Clinical spectrum @Kaggle
- Napomena* sadržaji dataset-ova se ne nalaze na repozitorijumu i potrebno ih je preuzeti i snimiti u direktorijum /data. Razlog je težina od više desetina GB.
+ Napomena** Nakon svake analize, podaci se mogu vizualizovati odvojenim skriptama iz direktorijuma /visualization.Kako postoji ukupno četiri grupa ETL analiza, nakon izvršavanja istih, moguće je pristupiti rezultatima i prikazati ih. Za potrebe vizualizacije rezultata odabrano je rešenje Dash Front-End koje je bazirano Flasku i Reactu. Pokretanje jedne ovakve web-aplikacije zahteva kao ulazni argumenat putanju do rezultata analiza na HDFS-u.
Na primer, pokretanje servera za vizualizaciju rezultata četvrtog dataseta dobija se uz:
$ cd visualization && py radiography_analysis_visualization.py hdfs://../path/to/results/COVID-19 Radiography Database @Kaggle
Ovaj dataset sadrži rendgenske snimke pacijenata obolelih od virusa SARS-CoV-2, pacijenata koji su pogođeni "normalnom" pneumonijom, kao i snimke zdravih ljudi. Tim istraživača Univerziteta u Dohi u saradnji sa medicinskim stručnjacima sastavio je ovaj izvor podataka.
Počnimo od učitavanja (Extraction) i formiranja polaznog DataFrame-a. Za potrebe eksrakcije, koristi se već dostupan deo API-a Sparka za analizu slika. Kako će se kasnije raditi predikcije, dodaja se kolona label koja će se koristiti kao deskriptor klase kojoj primerak slike pripada.
def extract_data(spark):
...
dataframe_normal = spark.read.format("image").option("dropInvalid", True) \
.load(normal_image_dir).withColumn("label", func.lit(DESCRIPTOR_NORMAL))
dataframe_covid19 = spark.read.format("image").option("dropInvalid", True) \
.load(covid19_image_dir).withColumn("label", func.lit(DESCRIPTOR_COVID19))
dataframe_lung_opacity = spark.read.format("image").option("dropInvalid", True) \
.load(lung_opacity_image_dir).withColumn("label", func.lit(DESCRIPTOR_LUNG_OPACITY))
dataframe_viral_pneumonia = spark.read.format("image").option("dropInvalid", True) \
.load(viral_pneumonia_image_dir).withColumn("label", func.lit(DESCRIPTOR_VIRAL_PNEUMONIA))
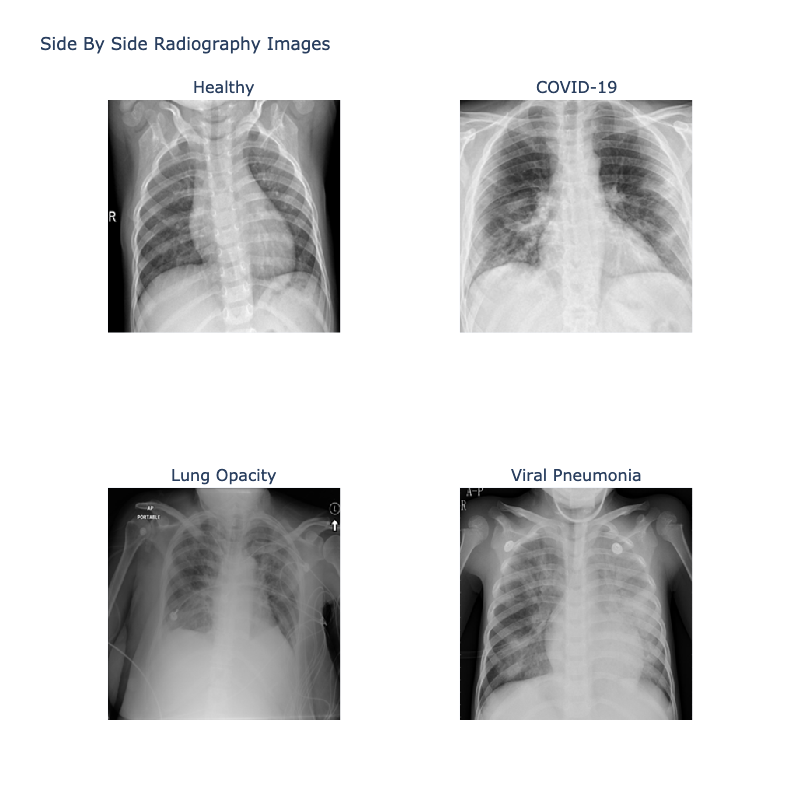
return [dataframe_normal, dataframe_covid19, dataframe_lung_opacity, dataframe_viral_pneumonia]Zatim se uzima četiri nasumičnih snimaka (po jedan iz svake kategorije). Polazi se od pregleda snimaka i traži postojanje šablona između slike i grupe kojoj pripada. Na RGB slici, svaki piksel predstavljen je sa tri 8-bitna broja koji predstavljaju vrednosti crvene, zelene i plave boje. Ovi brojevi imaju vrednosti u opsegu od 0 do 255 za svaki kanal boja.
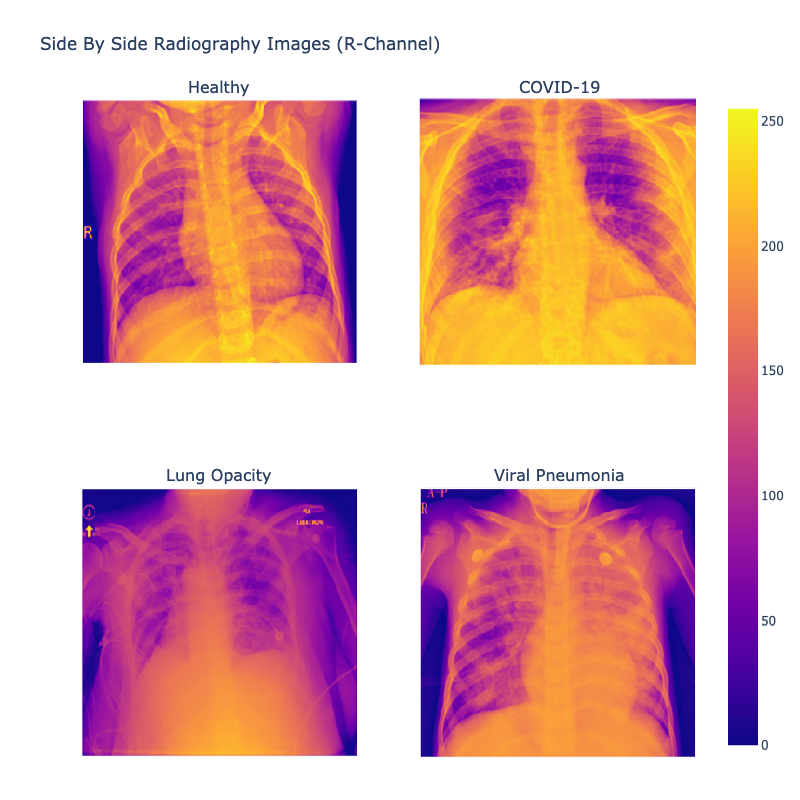
Iako su slike dataseta u crno-beloj boji, sačinjene su od tri kanala, međutim vrednosti svakog kanala su identične po pikselu. Zatim, na sledećoj slici dat je pregled istih slika sa izolovanim kanalom crvene boje.
Iz svega do sada izloženog, može se zaključiti da su rendgenski snimci iz ugla obrade prosti nizovi brojeva oblika [Height, Width, Channel].
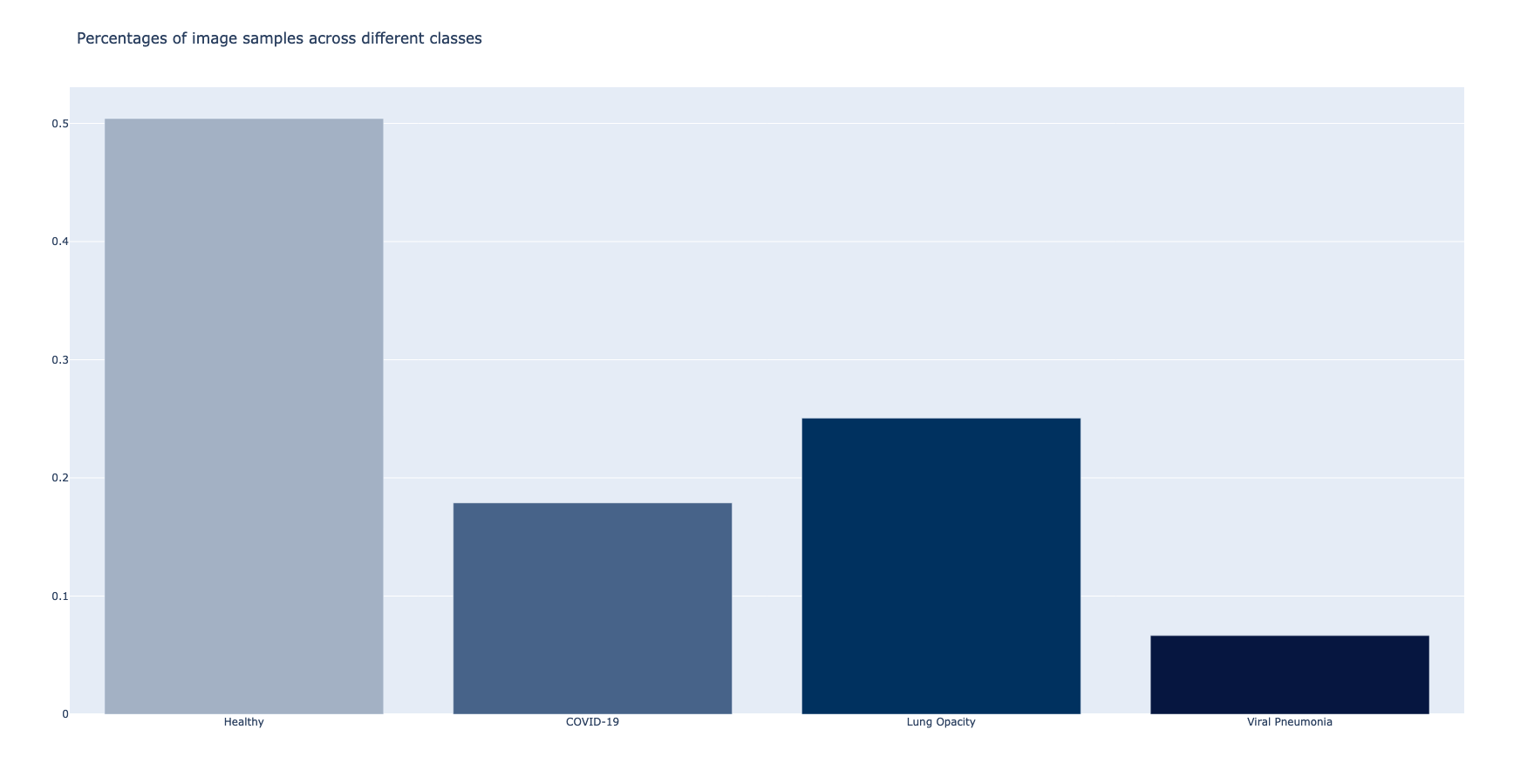
Sada je dobar trenutak razmotriti zastupnost svih klasa slika u datasetu. Grupisanjem po prethodno pomenutoj opisnoj koloni, a zatim i primenom agregacije, dobija se pregled zastupljenosti.
def transform_percentage_of_samples(dataframe):
df_percentages = dataframe.groupby('label') \
.agg((func.count('image')) \
.alias('count'), (func.count('image') / dataframe.count()) \
.alias('percentage')) \
.orderBy(func.col("label").asc())
return df_percentagesMože se videti da se klasa slika sa najvećim brojem primeraka odnosi na snimke zdravih pacijenata.
Ova analiza bi trebala da da uvid u moguće šablone koji postoje između boja snimaka i klasa kojima pripadaju. Traže se najmanje, najveće, srednje, kao i vrednosti standardne devijacije (engl. standard deviation) boja/osvetljenja.
def transform_colour_distribution(dataframe):
udf_function_min = udf(min_value, FloatType())
udf_function_max = udf(max_value, FloatType())
udf_function_mean = udf(mean_value, FloatType())
udf_function_standard_deviation = udf(standard_deviation_value, FloatType())
sample_size = 1000
df_normal = dataframe \
.filter(dataframe.label == DESCRIPTOR_NORMAL) \
.limit(sample_size)
df_covid19 = dataframe \
.filter(dataframe.label == DESCRIPTOR_COVID19) \
.limit(sample_size)
df_lung_opacity = dataframe \
.filter(dataframe.label == DESCRIPTOR_LUNG_OPACITY) \
.limit(sample_size)
df_viral_pneumonia = dataframe \
.filter(dataframe.label == DESCRIPTOR_VIRAL_PNEUMONIA) \
.limit(sample_size)
df_merged = reduce(lambda first, second: first.union(second),
[df_normal, df_covid19, df_lung_opacity, df_viral_pneumonia])
df_colour_distribution = df_merged \
.withColumn("min", udf_function_min("image.data")) \
.withColumn("max", udf_function_max("image.data")) \
.withColumn("mean", udf_function_mean("image.data")) \
.withColumn("standard_deviation", udf_function_standard_deviation("image.data"))
return df_colour_distributionKako je ovo veoma skupa operacija, bira se nasumičnih hiljadu slika iz svake klase. Zatim, primenom udf funkcija nalaze se pomenute vrednosti za svaku od njih. Separacijom po klasama, možemo da utvrdimo i vizualizujemo distribucije pronađenih vrednosti.
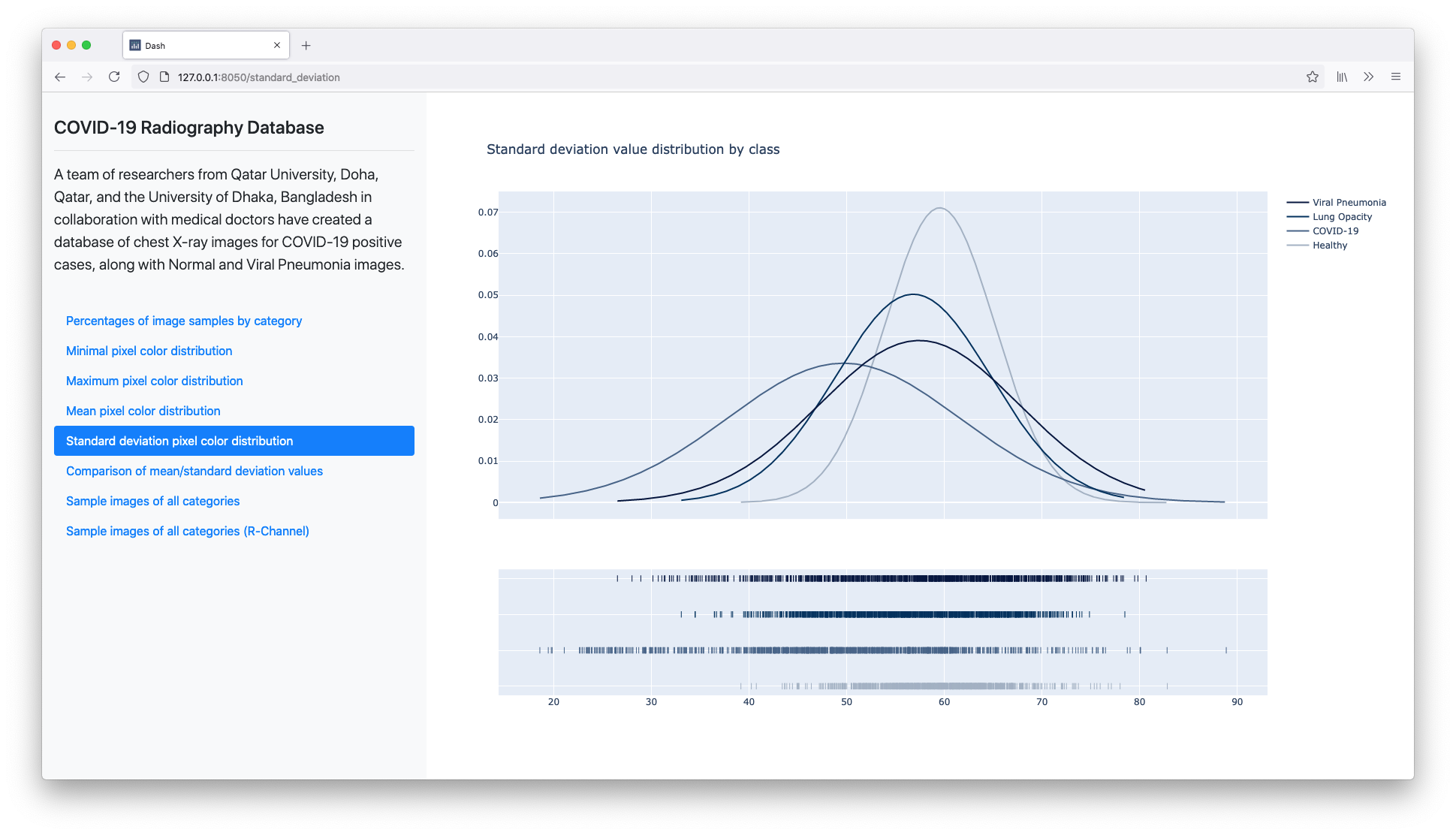
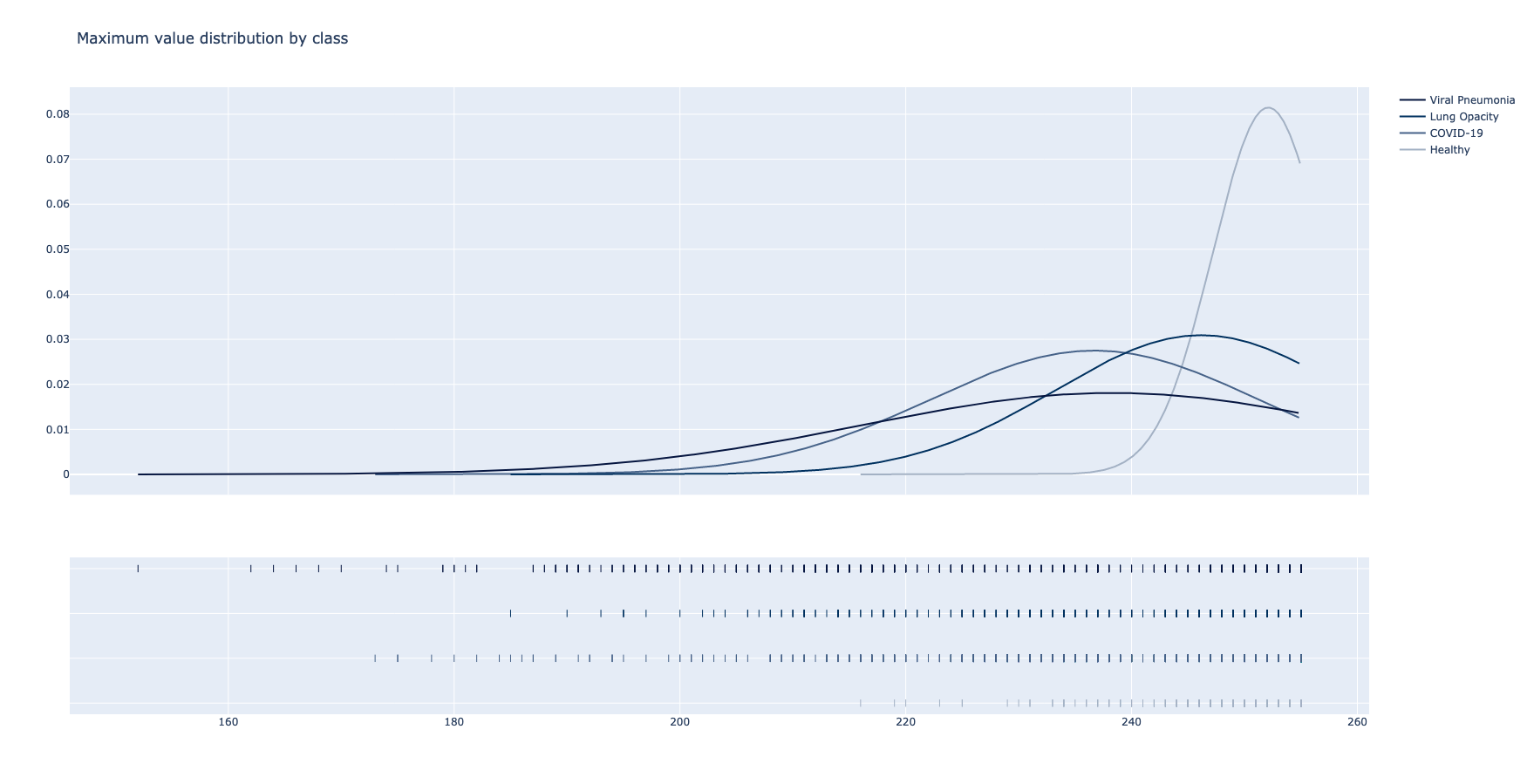
Klasa Viral Pneumonia se pokazala kao jedina koja približno oslikava "Normalnu" distribuciju @Wikipedia.
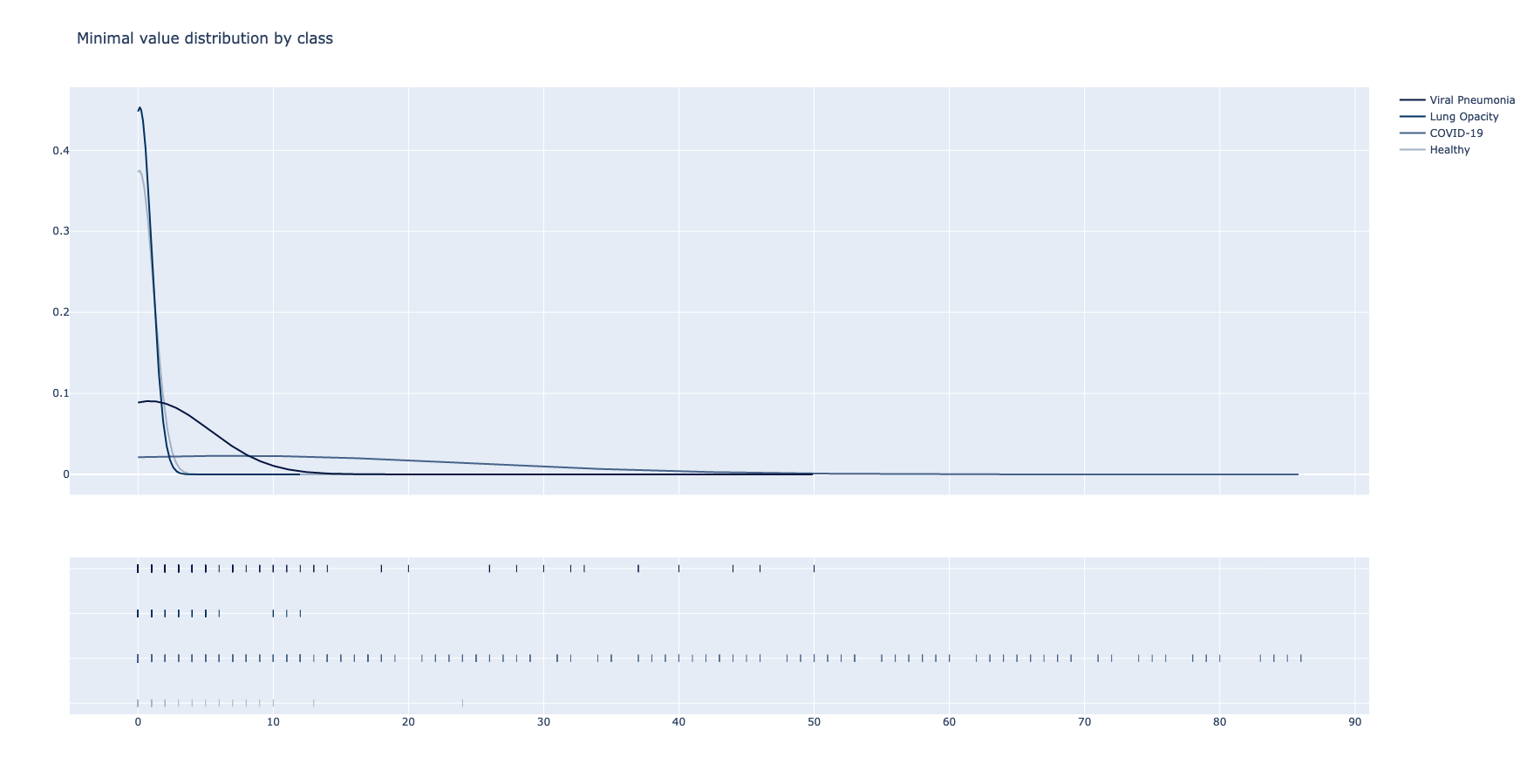
Najveća moguća vrednost osvetljenja je 255 i sve dostižu vrhove u ovom regionu, što se i očekuje. Viral Pneumonia, ponovo, je jedina klasa koja pokazuje niže vrednosti prilikom prikaza distribucije maksimuma. U slučaju maksimuma, Lung Opacity i COVID-19 imaju slične distribucije dok je primetno da snimci zdravih pacijenata imaju svetlije karakteristike.
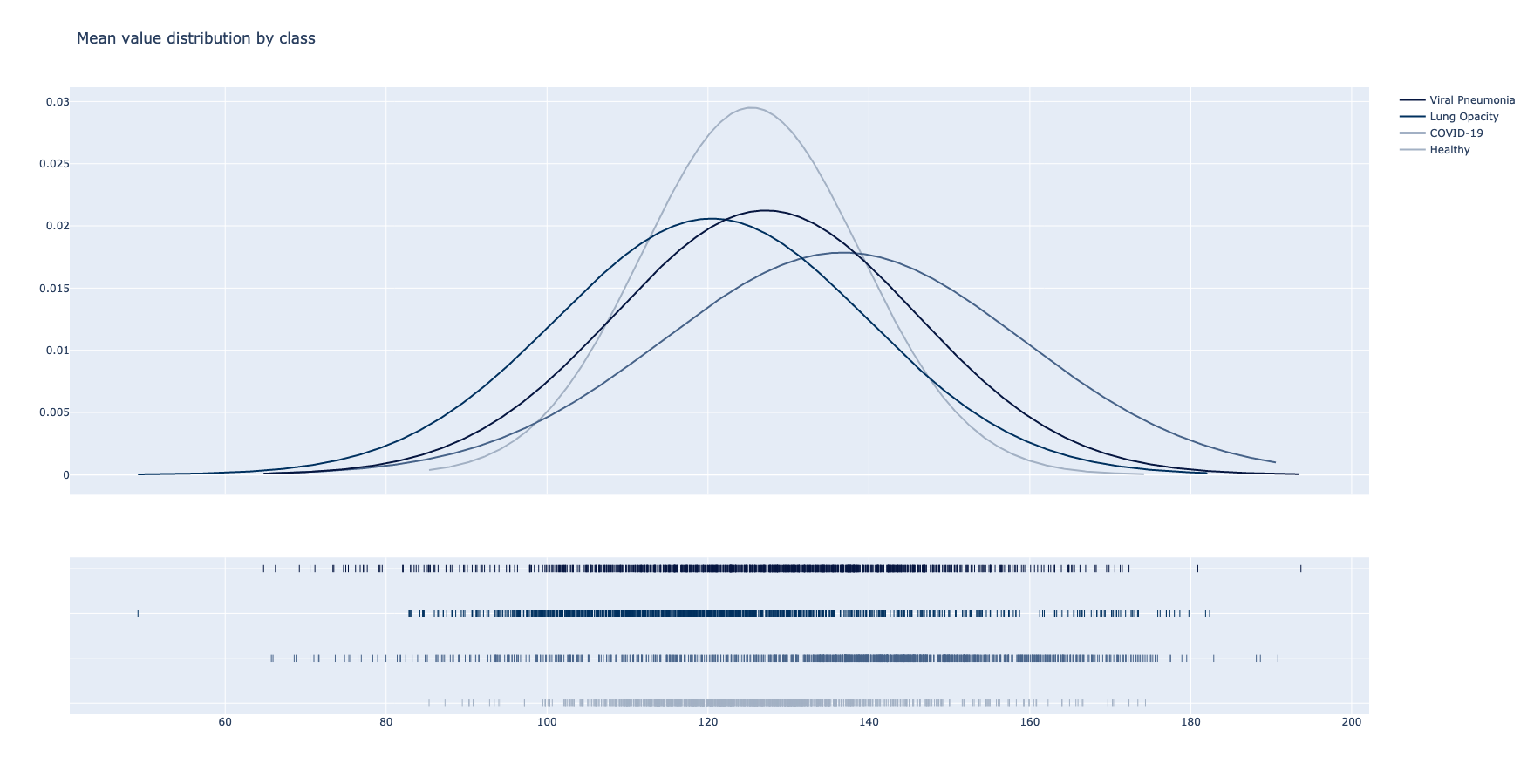
U slučaju srednjih vrednosti, klase Lung Opacity i Normal su predstavljene jako sličnom distribucijom. Kako se u svakom ciklusu analize uzima nasumičan podskup snimaka, može se zaključiti da postoji korelacija s obzirom da je u više navrata dobijen isti rezultat. Međutim, postoje jasne razlike u vrednostima dostignutih vrhova.
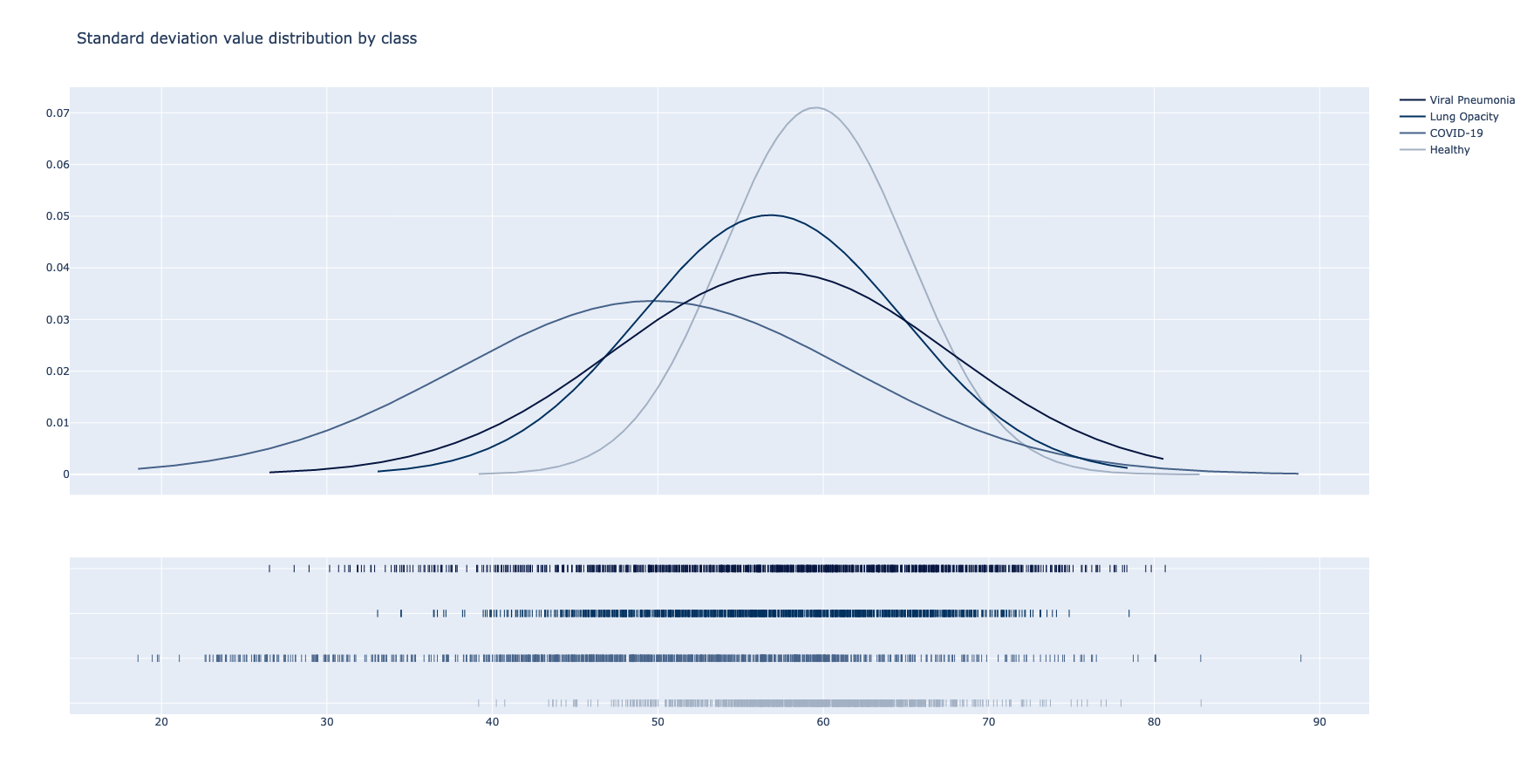
U slučaju standardne devijacije, odnosno mere odstupanja, većina primeraka poseduje vrednosti sličnih granica. Izuzetak predstavlja jedino klasa Normal sa primetno većim prosekom. Ova razlika iznosi približno 0.02.
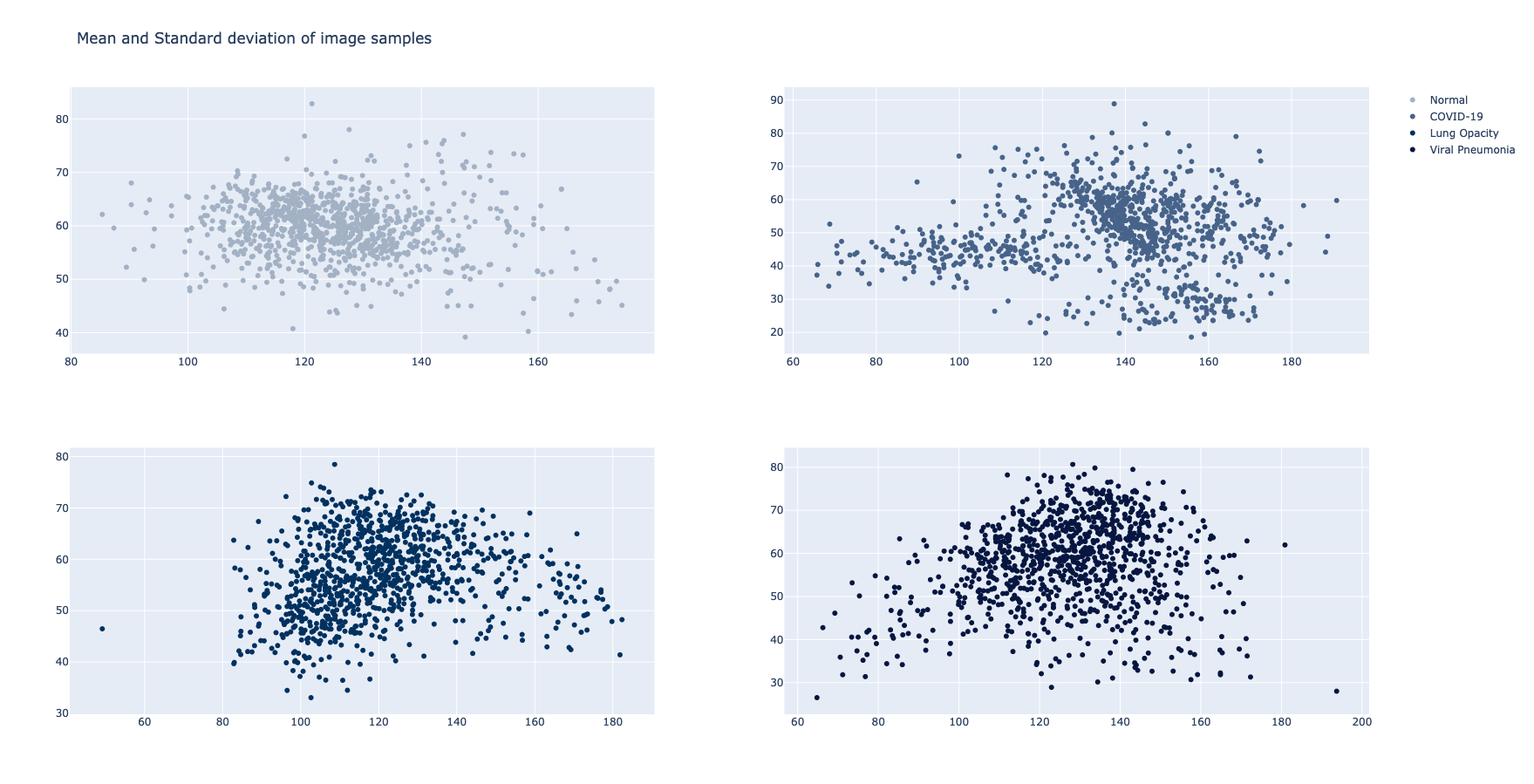
Na rezultatima x-osa predstavlja srednju vrednost, dok y-osa predstavlja unakrsne vrednosti standardne devijacije posmatranih snimaka. Na većini slika su grupacije prisutne u centralnom delu ravni što znači da nema velikog kontrasta između vrednosti piksela. Takođe, sve klase poseduju manji broj izolovanih uzoraka na periferiji spektra.
Klase COVID-19 i Viral Pneumonia su jedine koje poseduju manje rasute klastere u donjim-levim uglovima. Ovo su klasteri uzoraka sa malim srednjim vrednostima, kao i malim vrednostima devijacije.
Klasu Viral Pneumonia opisuje centralizovana veza, što znači da uzorci imaju više sličnosti među sobom, u poređenju sa ostalim klasama.
Klasu COVID-19, sa druge strane, opisuje suprotna rasuta veza. Odnosno, ima najviše rasutih i izolovanih uzoraka. Ovo može nagovestiti da rendgenski snimci ove grupe imaju više različitosti među sobom.
Sparkova mllib biblioteka poseduje paletu algoritama mašinskog učenja, jedan od ovih algoritama je Random Forest classifier. Takozvane šume sastoje se od stabala odluka, kombinuju veliki broj stabala kako bi se smanjio rizik overfitting-a. Ovaj algoritam se može koristi za binarnu i multiclass klasifikaciju.
U ovom slučaju, korišćena je multiclass klasifikacija za četiri grupe (prethodno pomenute grupe slika). Izdvajanjem opisnih parametara snimaka sastavlja se vektor karakteristika. Ovi parametri su min, max, mean i standard_deviation. Zatim se dataframe deli u odnosu 0.1/0.9 gde se veći deo koristi za treniranje ML modela. Nakon treniranja se vrši evaluacija modela na preostalim delom ulaza.
def transform_ml_classification(dataframe, spark):
...
df_vectorized = dataframe \
.withColumn("features_vector", udf_extract_features("image.data")) \
.withColumn("min", udf_function_min("image.data")) \
.withColumn("max", udf_function_max("image.data")) \
.withColumn("mean", udf_function_mean("image.data")) \
.withColumn("standard_deviation", udf_function_standard_deviation("image.data")) \
.select(["label", "min", "max", "mean", "standard_deviation"])
# Assembling features into a vector column
assembler = VectorAssembler(
inputCols=["min", "max", "mean", "standard_deviation"],
outputCol="features"
)
df_assembled = assembler \
.transform(df_vectorized) \
.select(['features', 'label'])
df_assembled.cache()
# Split the dataset into train/test subgroups
(training_data, test_data) = df_assembled.randomSplit([0.9, 0.1])
rf = RandomForestClassifier(labelCol='label', featuresCol='features', maxDepth=10)
rf_model = rf.fit(training_data)
rf_predictions = rf_model.transform(test_data)
multi_evaluator = MulticlassClassificationEvaluator(labelCol='label', metricName='accuracy')
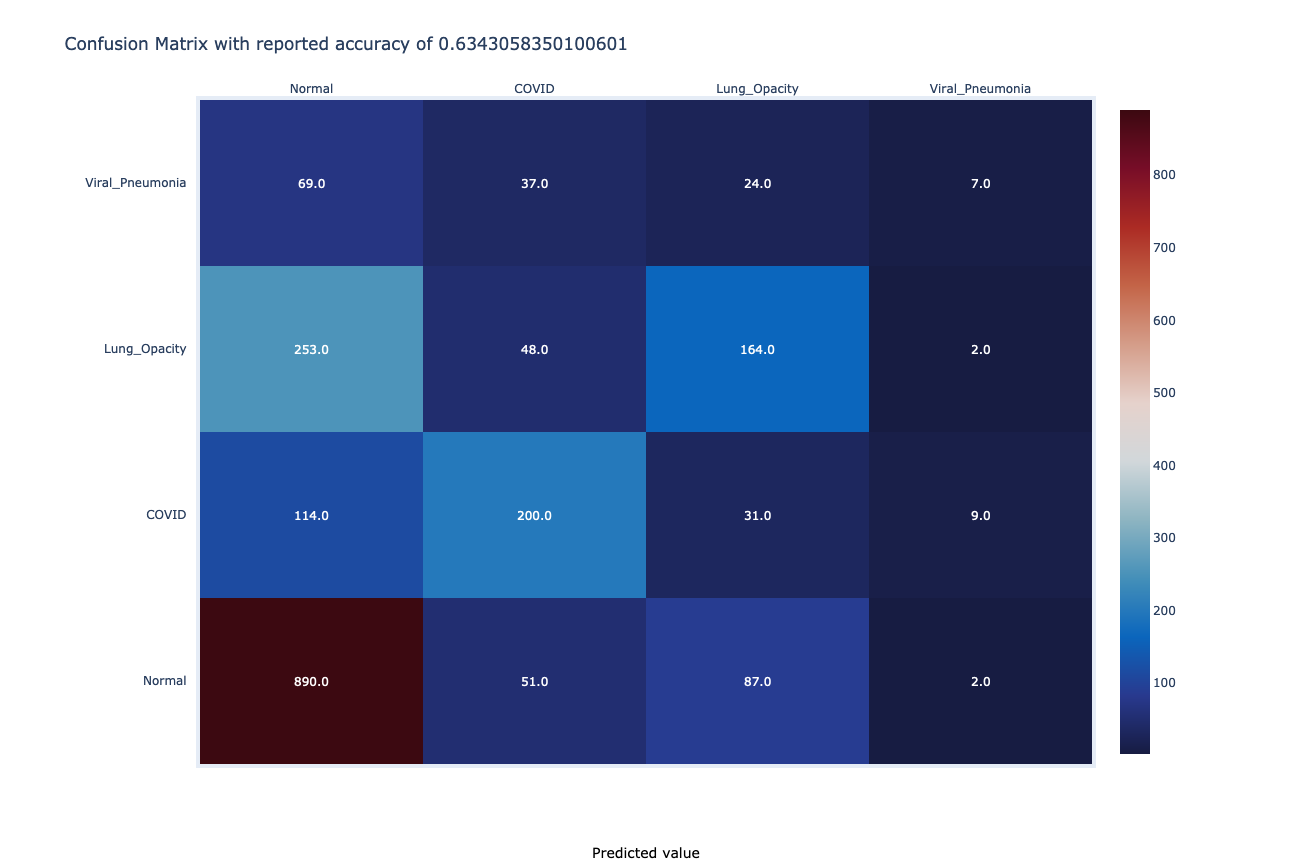
rf_accuracy = multi_evaluator.evaluate(rf_predictions)Evaluacijom je dobijena ukupna uspešnost od ~63%. Ovo je i očekivano s obzirom da je algoritam primenjen na neutemeljenoj metodi. Na sledećoj slici može se videti matrica pogodaka i promašaja modela. Model se pokazao kao najprecizniji za snimke klase Normal.
"Duboke" neuronske mreže su sofisticiranija metoda klasifikacije, pre svega u slučaju obrade vizualnih datasetova.
Međutim, podrška distribuiranog treniranja dubokih mreža u Pysparku nije na zavidnom nivou. Rešenja koja su dostupna neretko podržavaju samo starije verzije Pysparka ili nisu dovoljno stabilna. Iz ovog razloga, treniranje neuronske mreže nije distribuirano.
Odabrana je klasa CNN (Convolutional neural network) neuronskih mreža. Konvolucionalne mreže sastoje se od slojeva konvolucije čija je svrha detekcija šablona, praćenih slojevima pod-uzorkovanja. Pri tome, treniranje modela bazirano je na metodi poznatijoj kao Transfer Learning. Ovom metodom se znanje već treniranog, osnovnog modela, koristi za rešavanje drugog problema. Drugim rečima, eksploatiše se već naučeno iz jednog problema kako bi se generalizovao drugi.
Modeli trenirani nad slikovnim izvorima često koriste prve slojeve za detektovanje ivica, srednje za oblike, a kasnije slojeve za karakteristike. Transfer Learning metodom se slojevi prve dve grupe ostavljaju netaknutim, dok se slojevi za detekciju karakteristika prilagođavaju konkretnom problemu.
Za osnovni model odabran je DenseNet i sastoji se od 169 slojeva.
def transform_dl_classification(dataframe, spark):
classes = [CLASSNAME_NORMAL, CLASSNAME_COVID19, CLASSNAME_LUNG_OPACITY, CLASSNAME_VIRAL_PNEUMONIA]
batch_size = 16
epochs = 50
udf_function_get_hdfs_origin = udf(hdfs_origin, StringType())
udf_function_classify = udf(classify, StringType())
# Preparing the distributed dataframe
dataframe_keras = dataframe.withColumn("height", dataframe.image.height) \
.withColumn("width", dataframe.image.width) \
.withColumn("n_channels", dataframe.image.nChannels) \
.withColumn("class_name", udf_function_classify("label")) \
.withColumn("origin", udf_function_get_hdfs_origin("image"))
dataframe_keras = dataframe_keras.filter(func.col("class_name") != CLASSNAME_INVALID)
dataframe_keras = dataframe_keras.drop("image", "label")
dataframe_keras.cache()Samo "pripremanje" izvora za treniranje obavlja se distribuirano. Nakon oblikovanja ulaznog datafrema, isti se centralizuje u dirver procesu i počinje sa treniranjem mreže. Na kraju, potrebno je snimiti dobijeni model i iskoristiti ga za predikcije.
Ulazni dataframe deli se u razmeri gde se 80% koristi za treniranje modela, a preostalih 20% za validaciju. Validacija podrazumeva i izgrađivanje matrice preciznosti, uz pregled preciznosti po klasama za predikciju.
# Data generators
[train_datagen, train_gen] = train_generator_from_dataframe(dataframe_keras_master, batch_size, classes)
[test_datagen, test_gen] = test_generator_from_dataframe(dataframe_keras_master, batch_size, classes)
# Constructing the neural net
dense169 = DenseNet169(input_shape=(299, 299, 3), include_top=False, weights='imagenet')
dense169.trainable = False
model = Sequential(
[
dense169,
Flatten(),
Dense(units=256, activation='relu'),
Dense(units=256, activation='relu'),
Dense(units=4, activation='softmax')
]
)
model.compile(...)
model.fit(...)
model.save(...)
[conf_matrix, accuracy] = model_efficacy(model.predict(test_gen), test_gen, classes)
return [
spark.createDataFrame(conf_matrix),
spark.createDataFrame(accuracy)
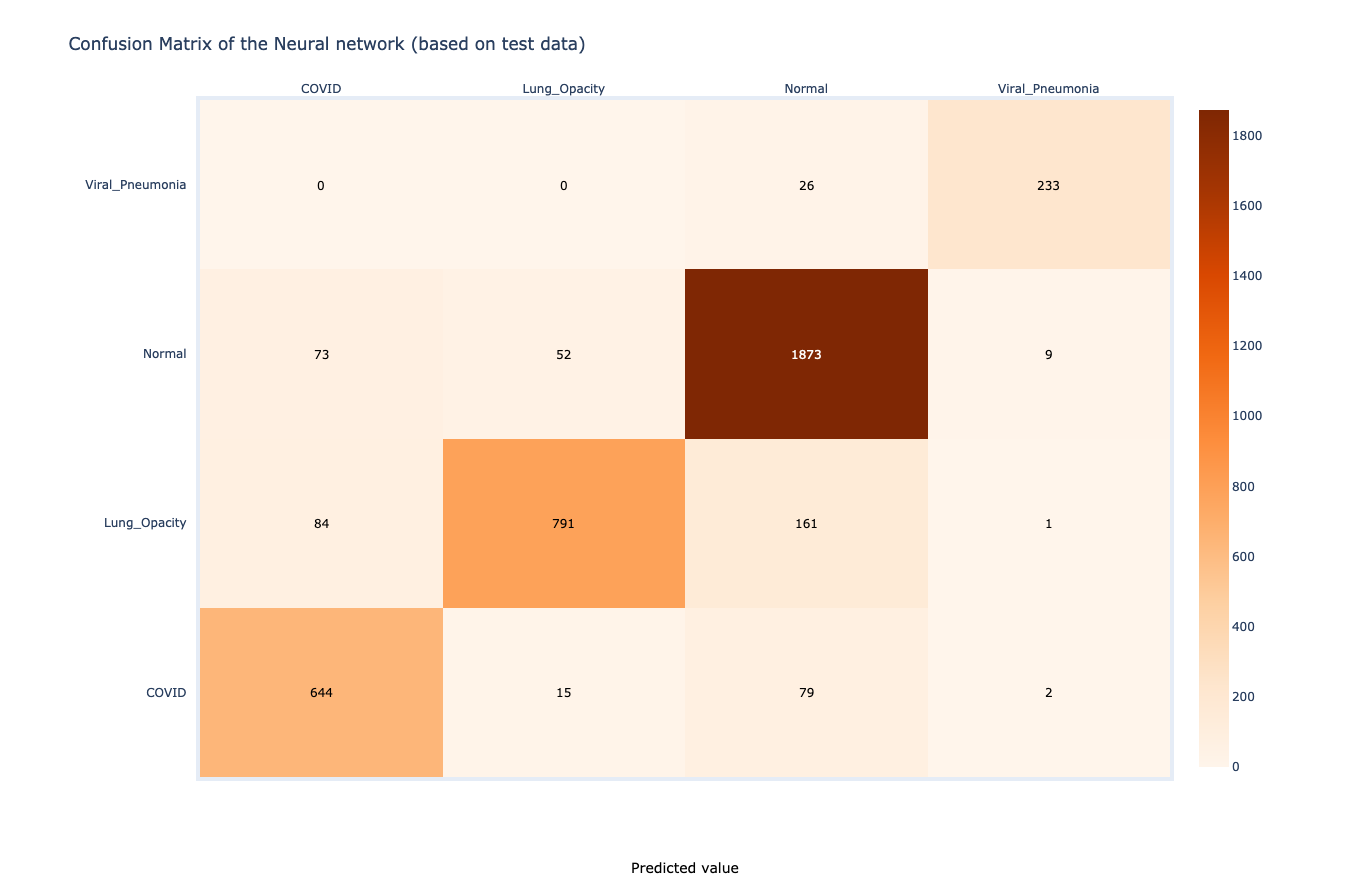
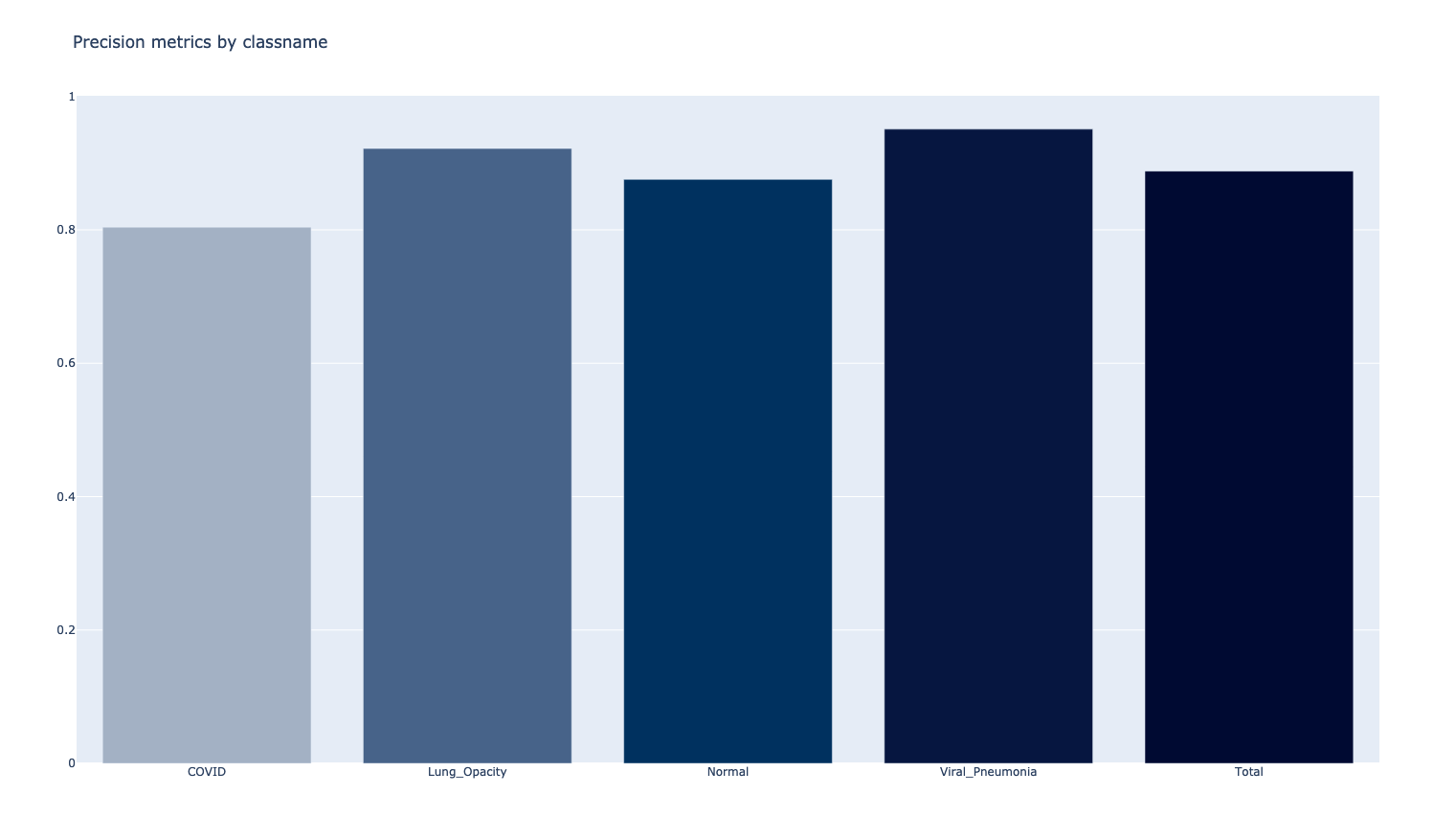
]Na slikama u nastavku mogu se videti matrica preciznosti, iliti "konfuzije", i distribucija preciznosti modela po klasama.
Matrica preciznosti daje pregled uspešnih klasifikacija, kao i promašaja. Najveći broj promašaja prisutan je u slučajevima klasifikacije COVID-19 pacijenata. Nasuprot, najveća preciznost ostvarena je u klasifikaciji primeraka viralne pneumonije.
Može se videti da je nakon treniranja od 50 epocha preciznost modela blizu 90%. Trenirani model nalazi se u direktorijumu /spark/keras-model i može se koristiti za distribuirano zaključivanje, koje je objašnjeno u sledećoj analizi.
Kako je model izgrađen na jednom čvoru, a zatim i snimljen, može se koristiti za distribuirane/paralelne obrade.
Metoda distribuiranog zaključivanja (engl. Model inference) je distribuirano izvršavanje modela nad delovima dataframea, paralelno i na različitim čvorovima. Model se učitava odvojeno na različitim čvorovima i primenjuje - time ostvarujući distribuirano zaključivanje.
Ova tehnika se veoma često koristi u slučaju obrada streaming podataka, ovde je primenjena nad delom ulaznog skupa podataka. Uzima se nasumičnih 100 primeraka i metodom inferencije se rade predikcije, oslanjajući se na prethodno trenirani model. Na ovaj način više ćvorova će paralelno obrađivati delove distribuirane strukture.
def transform_dl_model_inference(dataframe):
num_sample_images = 100
udf_function_get_hdfs_image = udf(hdfs_image_data, ArrayType(ByteType()))
# Take random 100 sample images
dataframe_pred = dataframe \
.withColumn("image_data", udf_function_get_hdfs_image("image")) \
.drop("image", "label") \
.limit(num_sample_images)
dataframe_pred.cache()
# Distributed model inference
# Predict batches across different partitions in parallel
dataframe_inference = dataframe_pred \
.select(predict_batch_udf(func.col("image_data")).alias("prediction"))
return dataframe_inference
@pandas_udf(ArrayType(FloatType()))
def predict_batch_udf(image_batch_iter):
batch_size = 64
model = load_model("./outputs/model/")
for image_batch in image_batch_iter:
images = np.vstack(image_batch)
ds_tensors = tf.data.Dataset.from_tensor_slices(images)
ds_tensors = ds_tensors.map(parse_image, num_parallel_calls=8) \
.prefetch(5000).batch(batch_size)
predictions = model.predict(ds_tensors)
yield pd.Series(list(predictions))predict_batch_udf je metoda koja će se izvršavati na svim čvorovima. Predikcije će se odnositi samo na delove dataframea koji se nalaze na konkretnom čvoru. Nakon paralelnih proračuna, sve predikcije se skupljaju u driver procesu.
COVID-19 Open Research Dataset Challenge @Kaggle
Ovaj dataset sadrži više od 400,000 medicinskih članaka o virusu COVID-19, SARS-CoV-2 i relevantnim temama.
Polazi se od definisanja šeme (shcema) dataseta, a zatim i izvlačenja abstract zapisa (sažetaka) svakog članka. Ovde se primenjuje par koraka normalizacije i "pročišćavanja".
def transform_papers_and_abstracts(dataframe):
df_abstracts = (
dataframe.select("paper_id", func.posexplode("abstract").alias("pos", "value"))
.select("paper_id", "pos", "value.text")
.withColumn("ordered_text", func.collect_list("text").over(Window.partitionBy("paper_id").orderBy("pos")))
.groupBy("paper_id")
.agg(func.max("ordered_text").alias("sentences"))
.select("paper_id", func.array_join("sentences", " ").alias("abstract"))
.withColumn("words", func.size(func.split("abstract", "\s+")))
)
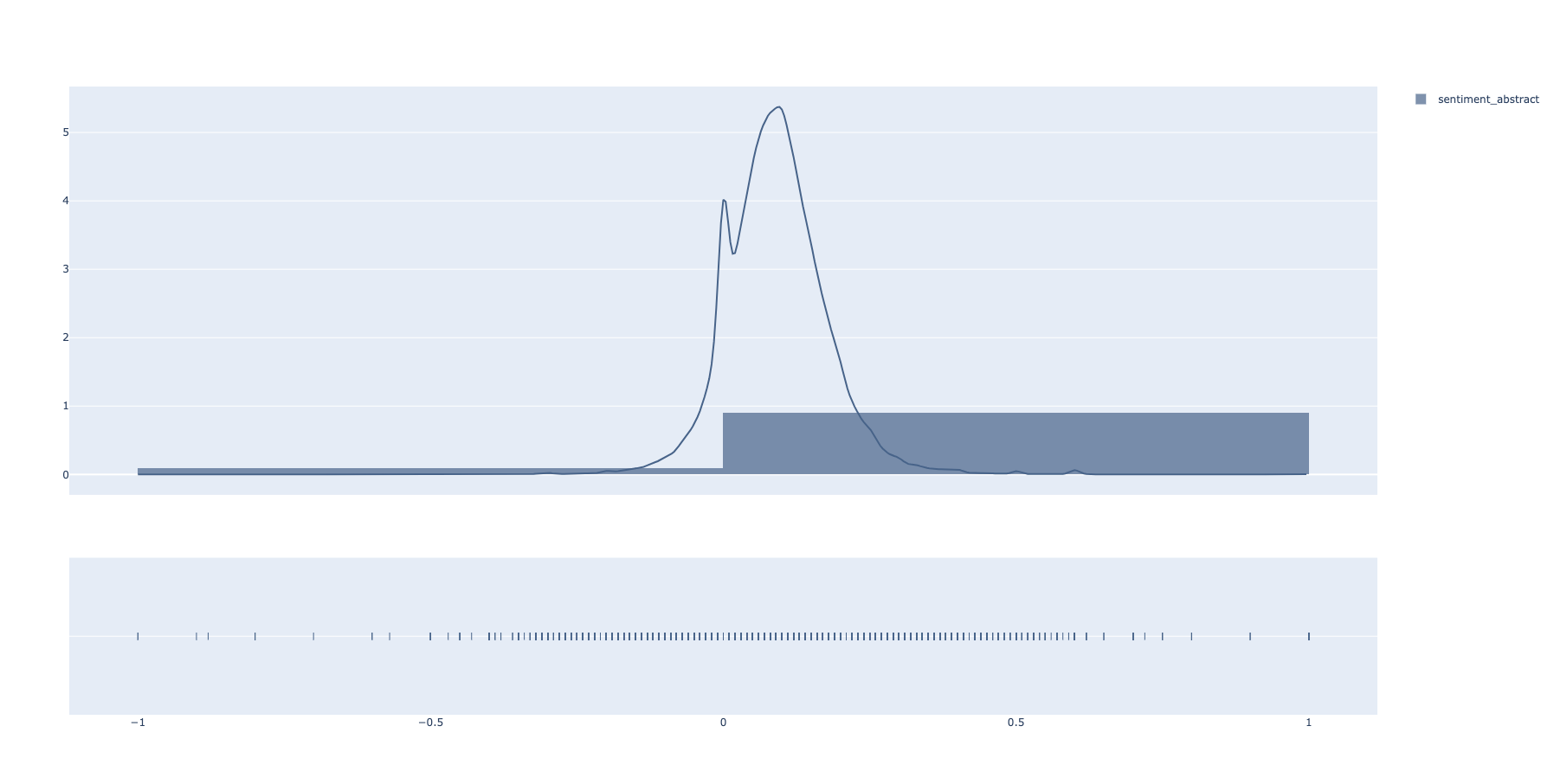
...Udf funkcijama paralelizovano se nalaze sentiment vrednosti sažetaka (abstract). Prethodno se sličnom analogijom vrši normalizacija svakog sažetka u vidu transformacije u mala slova i tokenizacije.
def transform_abstracts_words(dataframe):
udf_function_clean = udf(generate_cleaned_abstracts, StringType())
udf_function_sentiment = udf(generate_sentiment, DoubleType())
stopwords = set(STOPWORDS)
dataframe = dataframe.withColumn("clean_abstract", udf_function_clean("abstract"))
dataframe = dataframe.withColumn("sentiment_abstract", udf_function_sentiment("clean_abstract"))
...Oslanjajući se na prethodno dobijeni i normalizovani dataset, gradi se korpus reči i prikazuje 25 najčešće korišćenih.
COVID-19 Dataset @Kaggle
Ovaj dataset sadrži brojeve registorvanih, oporavljenih i preminulih pacijenata od virusa COVID-19 širom sveta, podeljeno po geografskim oblastima.
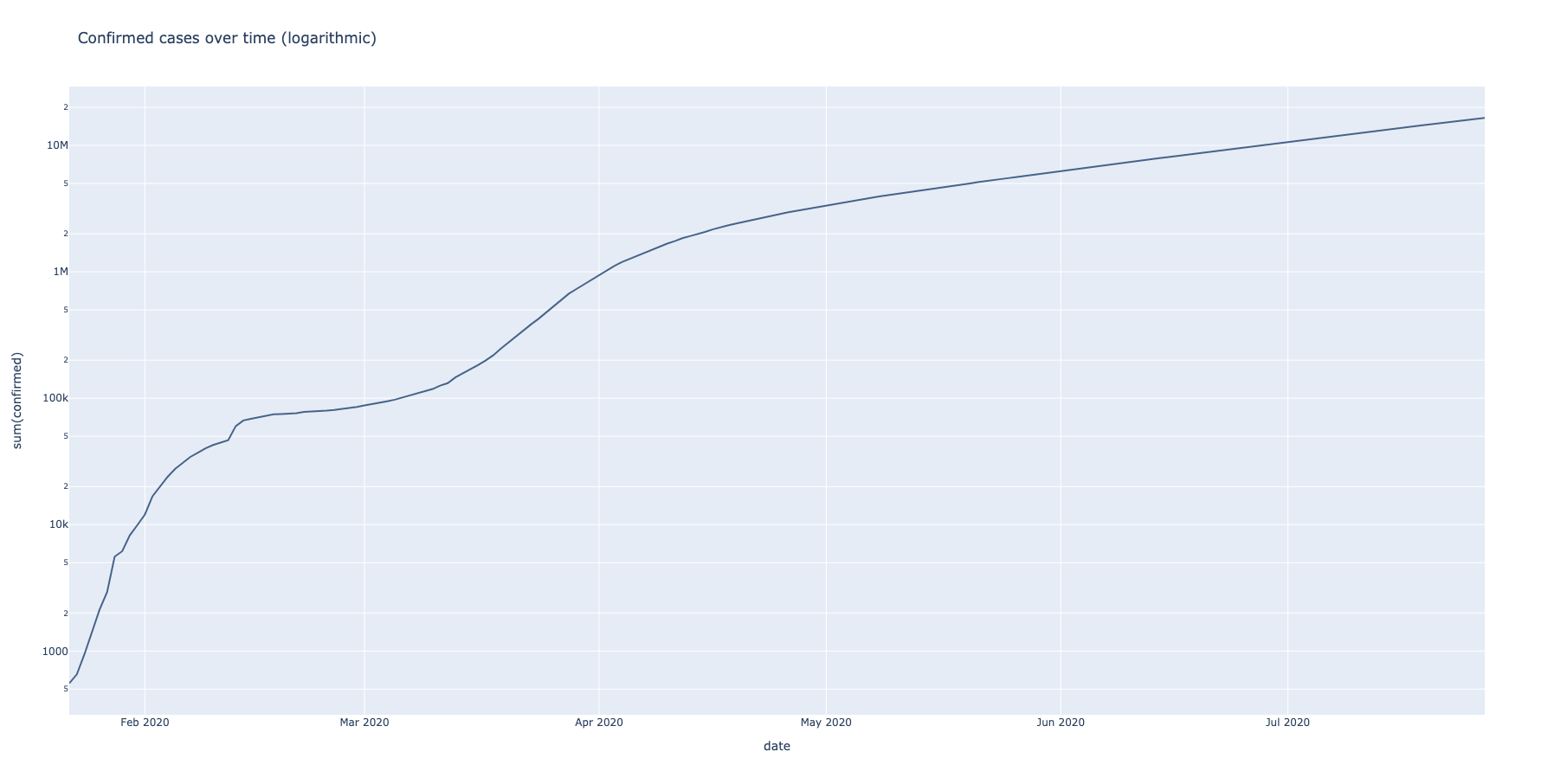
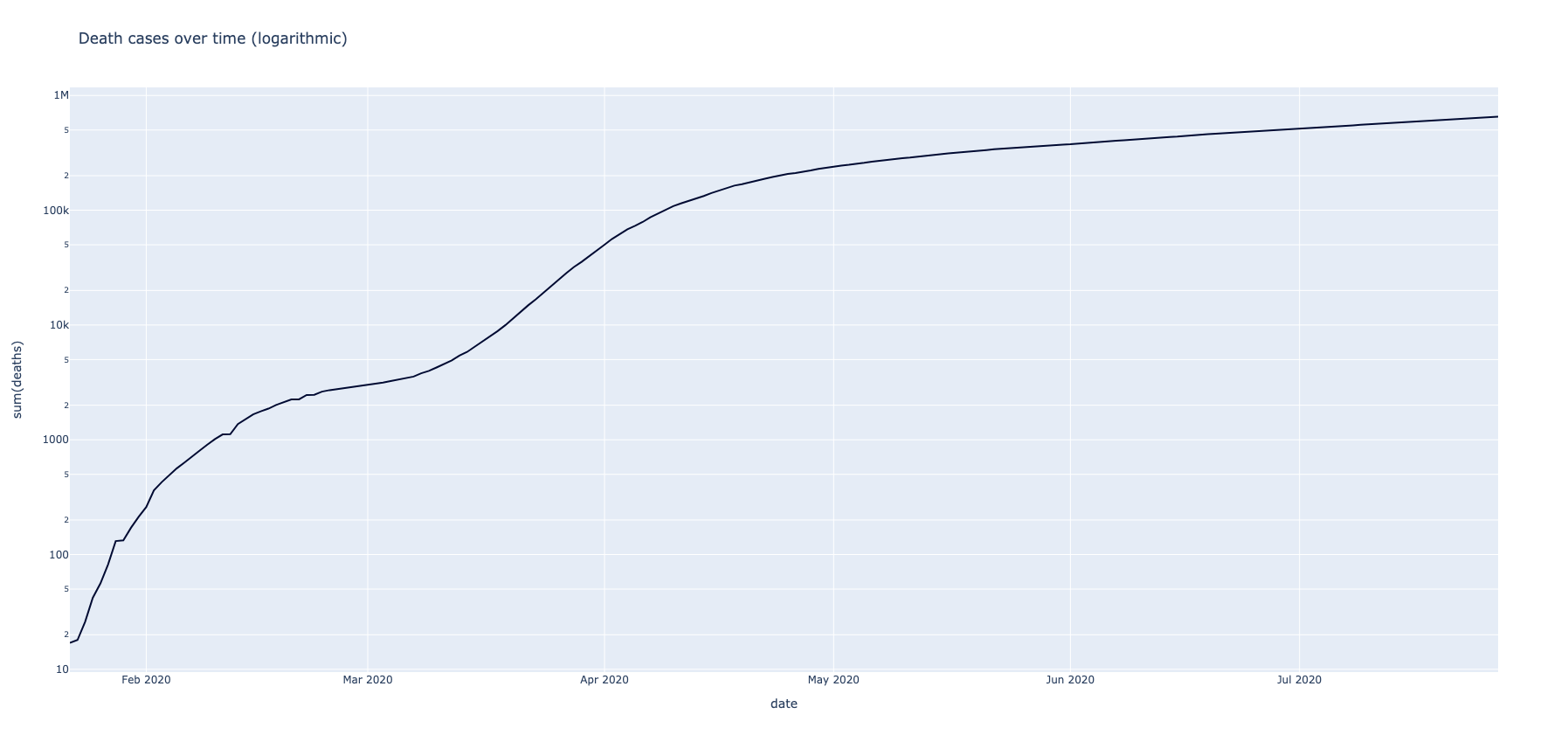
Nakon uvodnih transformacija i proširivanja dataseta novim kolonama, kao i preuređivanjem početnih, vrši se sumiranje gorepomenutih klasa slučajeva. Sumirani vremenski prikaz registrovanih i preminulih pacijenata prikazuje se po logaritamskoj skali.
def transform_papers_and_abstracts(dataframe):
dt_transformed = dataframe
dt_transformed = dt_transformed.fillna('', subset=['state'])
dt_transformed = dt_transformed.fillna(0, subset=['confirmed', 'deaths', 'recovered', 'active'])
dt_transformed = dt_transformed.withColumn("active", dt_transformed["confirmed"] - dt_transformed["deaths"] - dt_transformed["recovered"])
dt_transformed = dt_transformed.withColumn("country", func.regexp_replace('country', "Mainland China", "China"))
df_globally = dataframe.groupBy("date").sum("confirmed", "deaths").orderBy("date")
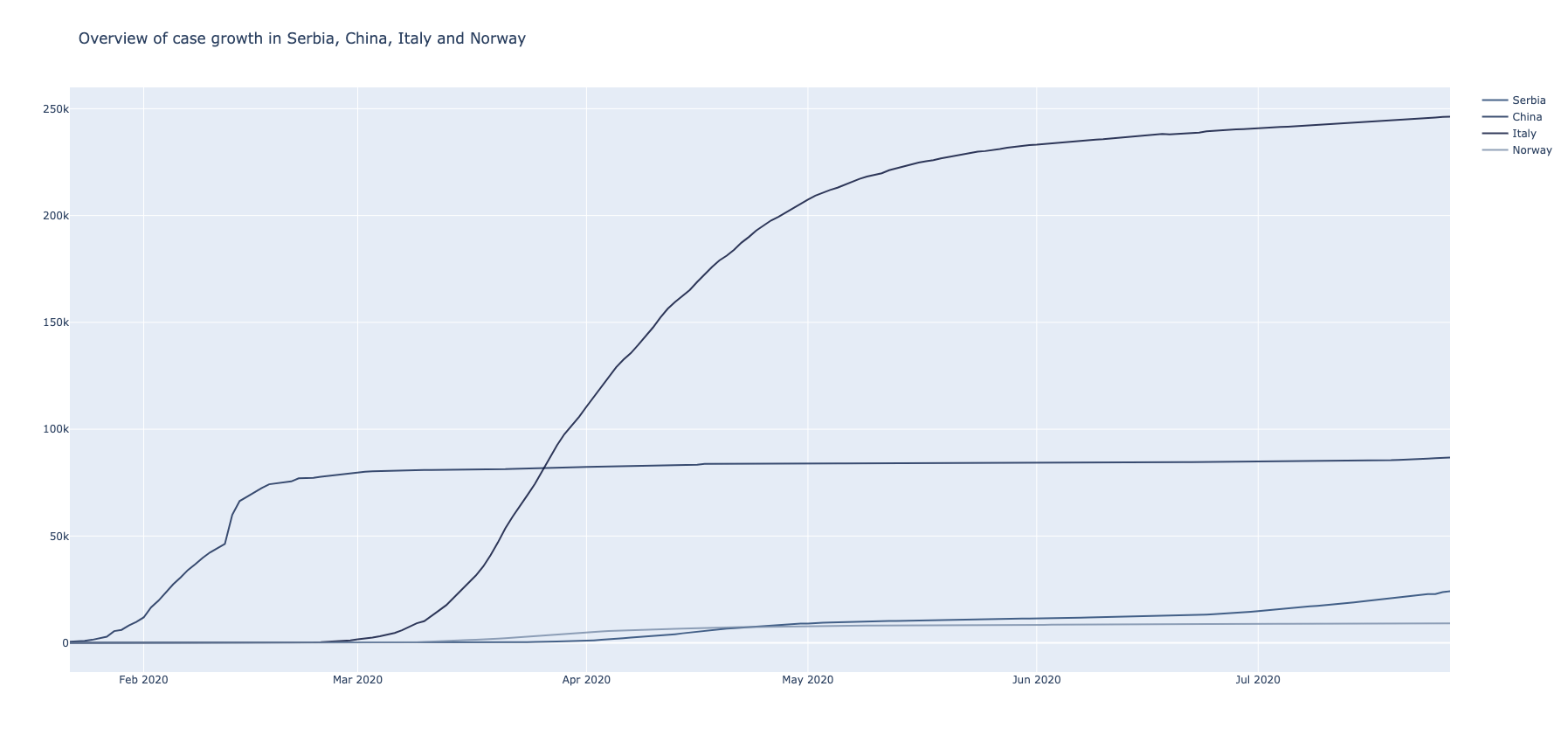
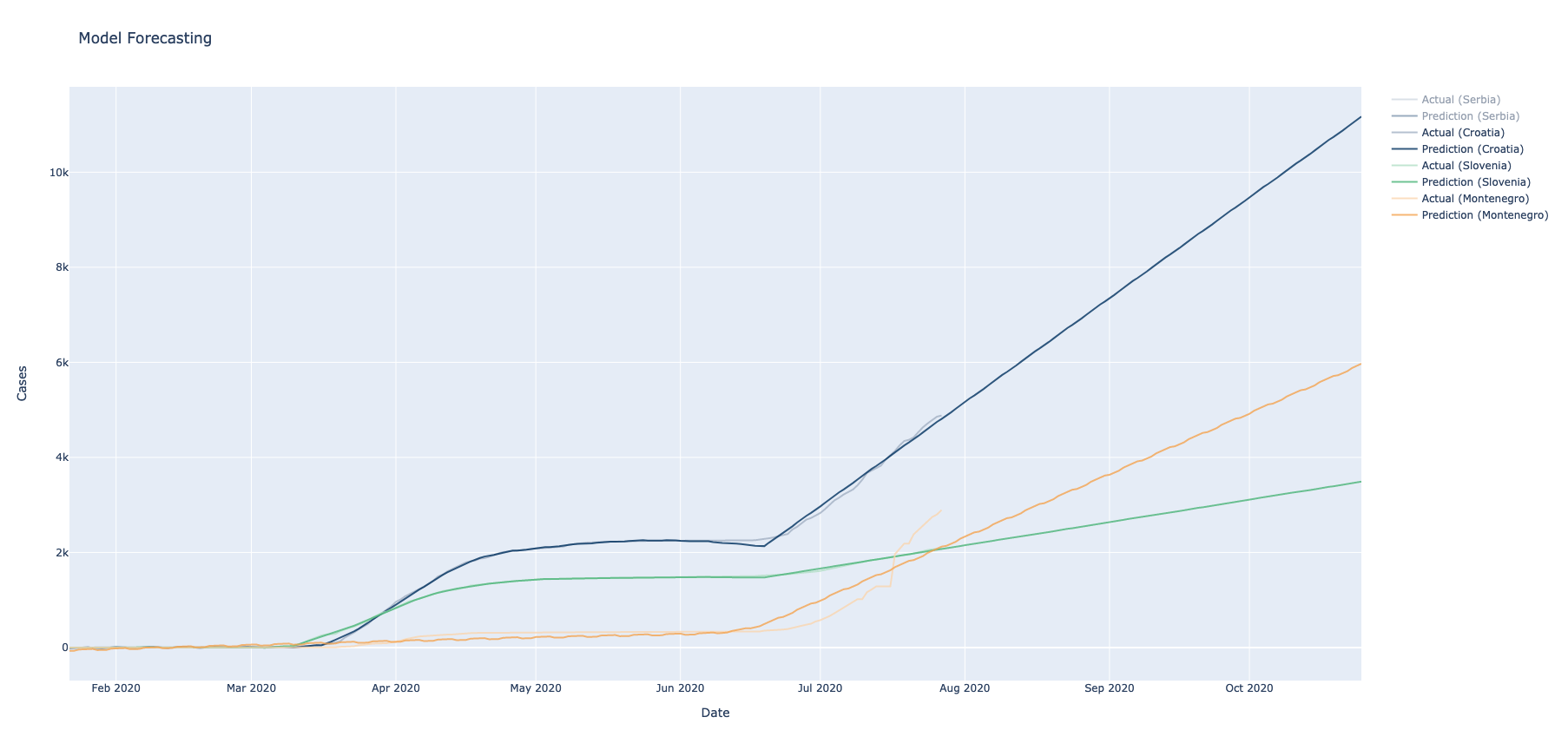
...Kao primer, izabrane su države - Srbija, Kina, Italija i Norveška. Filtriranjem i sumiranjem dobijaju se vremenski pregledi napredovanja virusa u njima.
def transform_confirmed_cases_countries(dataframe):
df_serbia = dataframe.filter(dataframe.country == "Serbia")
df_china = dataframe.filter(dataframe.country == "China")
df_italy = dataframe.filter(dataframe.country == "Italy")
df_norway = dataframe.filter(dataframe.country == "Norway")
df_serbia_grouped = df_serbia.groupBy("date").sum("confirmed").orderBy("date")
df_china_grouped = df_china.groupBy("date").sum("confirmed").orderBy("date")
df_italy_grouped = df_italy.groupBy("date").sum("confirmed").orderBy("date")
df_norway_grouped = df_norway.groupBy("date").sum("confirmed").orderBy("date")
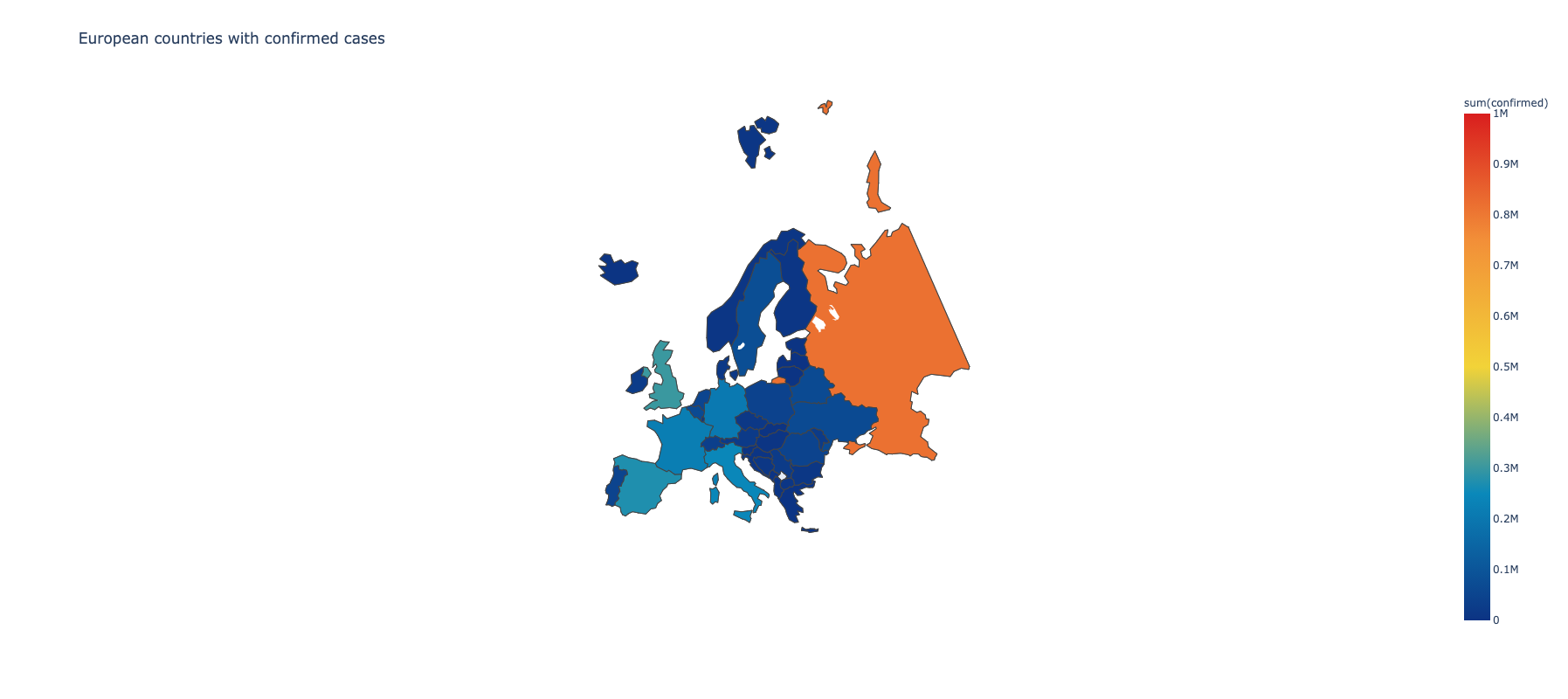
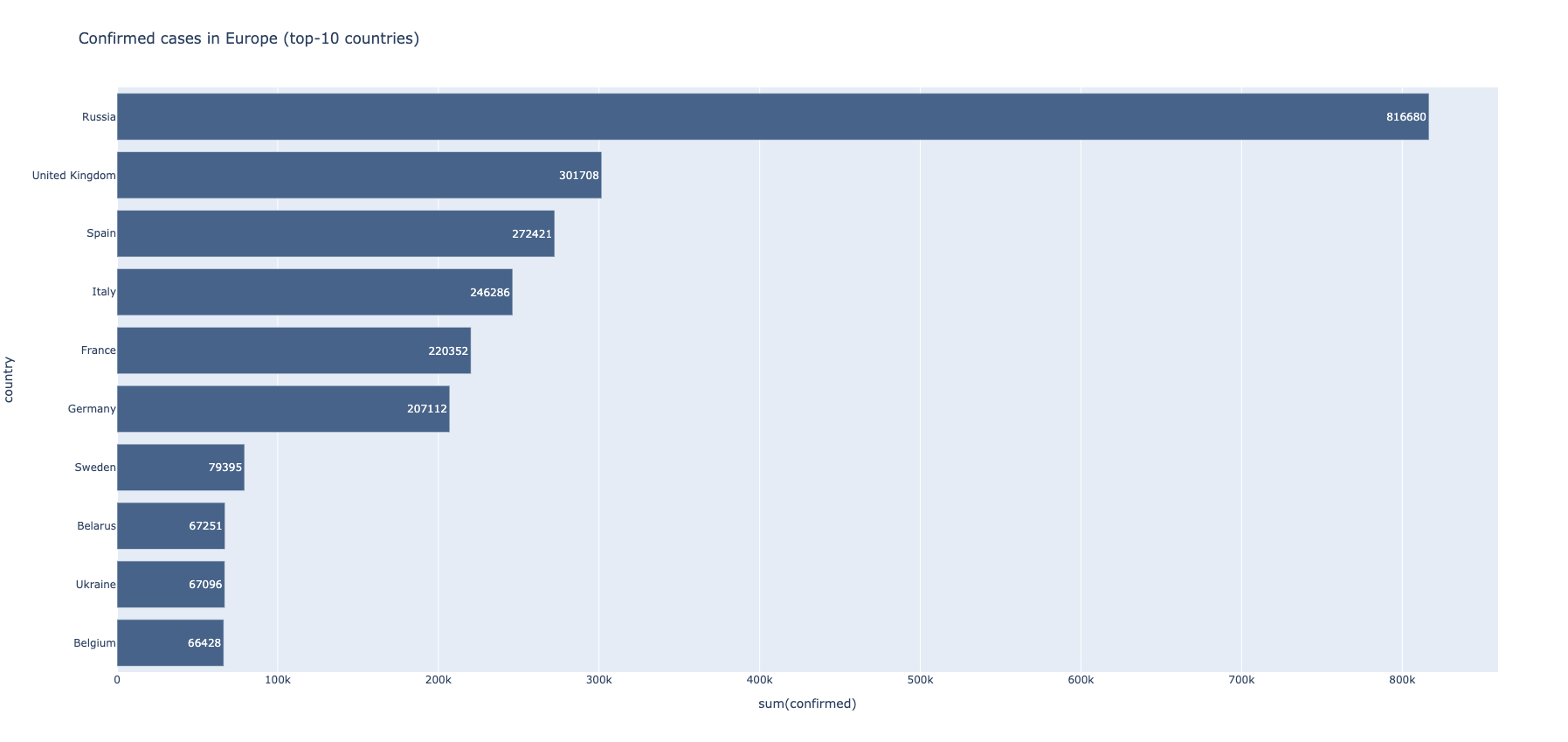
...Particionisanjem datafrema po zemlji, a zatim i filtiranjem sa grupacijom koja uzima u obzir samo države Evrope, dobija se presek trenutnog stanja registrovanih pacijenata na našem kontinentu.
def transform_confirmed_cases_europe(dataframe):
df_temp = dataframe.select([c for c in dataframe.columns if c not in {"state"}])
w = Window.partitionBy("country")
df_latest = df_temp.withColumn("maxDate", func.max("date").over(w)).where(func.col("date") == func.col("maxDate"))
df_latest_grouped = df_latest.groupby("country").sum("confirmed")
df_latest_grouped_europe = df_latest_grouped.filter(df_latest_grouped.country.isin(europe))
...Iz istog dataframe-a se lako izvlači opadajuća lista najgore pogođenih država.
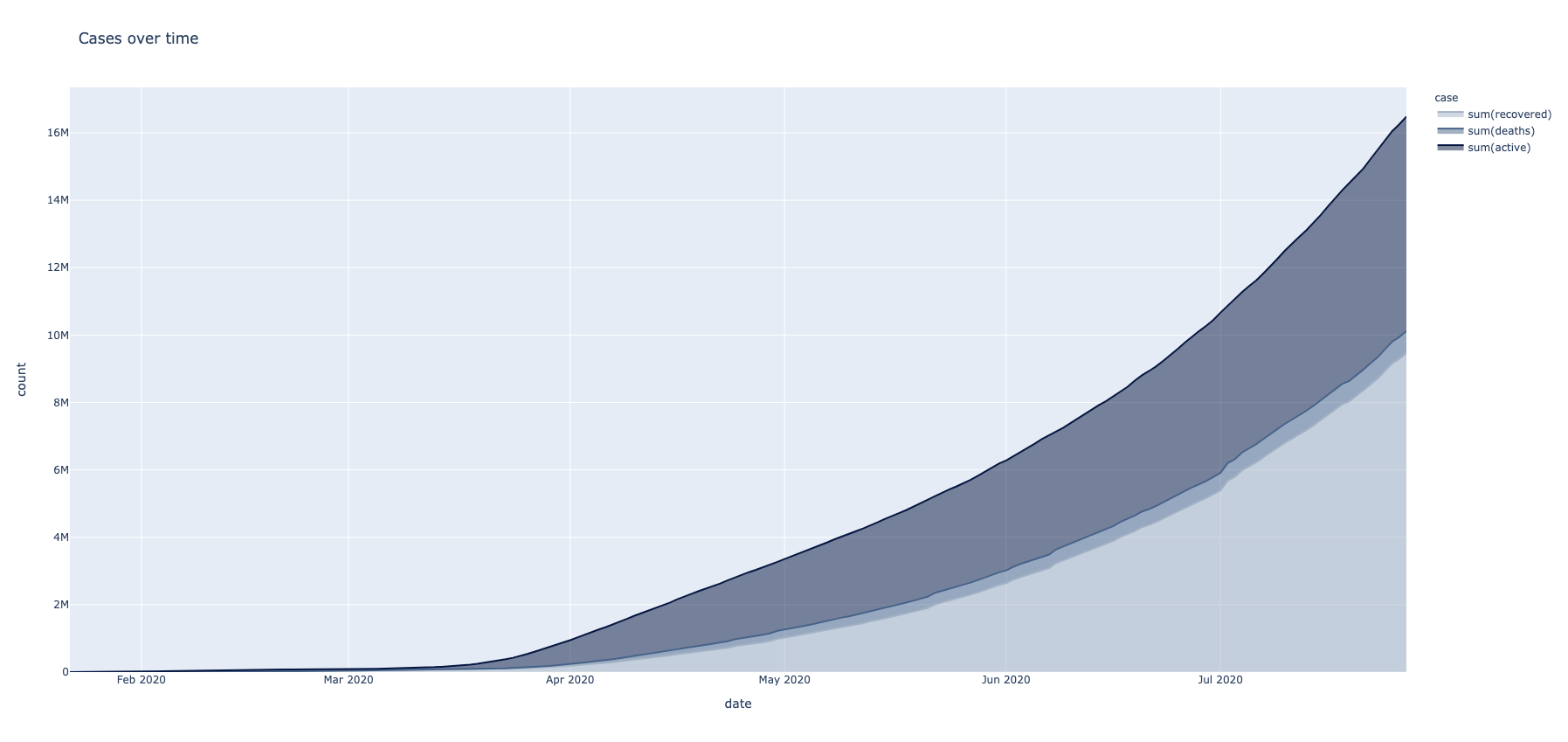
Prethodno obogaćeni dataframe active kolonom koja predstavlja razliku ostalih kolona se koristi kao osnova. Sa ovime na umu, veoma lako se vrši sumiranje traženih parametara.
def transform_confirmed_cases_comparison(dataframe):
df_grouped = dataframe.groupBy("date").sum("recovered", "deaths", "active").orderBy("date")
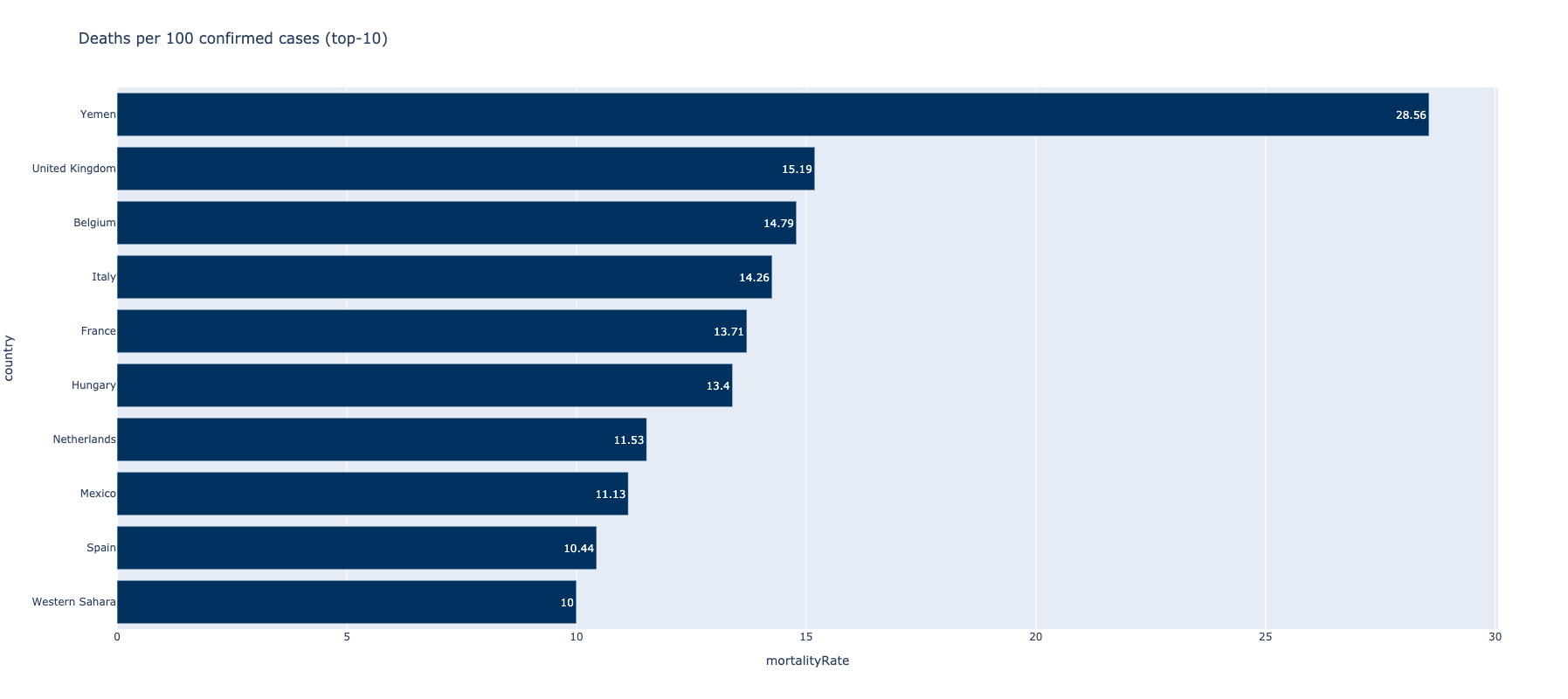
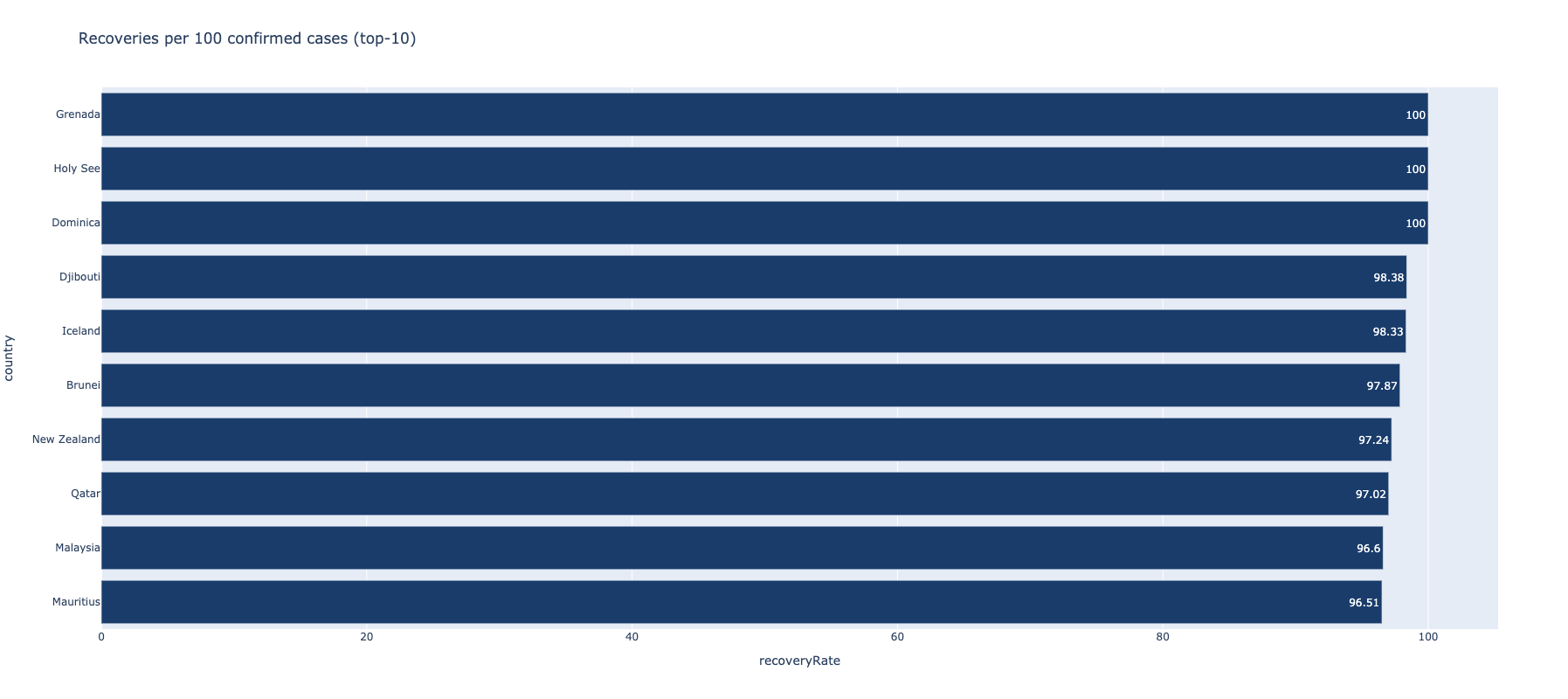
...Dodavanjem novih kolona i izvlačenjem informacija o odnosima oporavljenih i preminulih pacijenata u odnosu na ukupan broj, dobija se mera kvaliteta ophođenja država prema pandemiji. Zatim, na osnovu novododatih kolona, lako se izdvajaju države se najboljim ili najgorim koeficijentima.
def transform_confirmed_cases_comparison_countries(dataframe):
w = Window.partitionBy('country')
df_latest = dataframe.withColumn("maxDate", func.max("date").over(w)).where(func.col("date") == func.col("maxDate"))
df_latest_grouped = df_latest.groupby("country").sum("confirmed", "deaths", "recovered", "active")
df_latest_grouped_with_mortality_rate = df_latest_grouped.withColumn("mortalityRate", func.round(
df_latest_grouped["sum(deaths)"] / df_latest_grouped["sum(confirmed)"] * 100, 2)).orderBy("mortalityRate")
df_latest_grouped_with_recovery_rate = df_latest_grouped.withColumn("recoveryRate", func.round(
df_latest_grouped["sum(recovered)"] / df_latest_grouped["sum(confirmed)"] * 100, 2)).orderBy("recoveryRate")
...Analizom serijskih vremenskih podataka mogu se utvrditi trendovi i predviđanja u sklopu nekog domena. S obzirom da je dataset vremenski orijentisan, mogu se koristiti biblioteke za treniranje modela i predviđanje budućnosti. Odabrana biblioteka koja je korišćena u ovom slučaju naziva se prophet.
Počevši od filtriranja po državama, dobija se takozvani timeseries dataframe koji sadrži dnevne preseke četiri države.
Zatim, izvršenjem operacije groupBy po koloni države, iteratori koji sadrže podatke podeljene po državama biće raspoređeni na različitim čvorovima. Drugim rečima, jedan worker čvor će držati barem jednu grupu. Treniranje prophet modela može se odavde izvršavati distribuirano. Svaki od čvorova će vršiti potrebne proračune za formiranje modela nad svojom pod-grupom podataka (svi će se odnositi na istu državu). @pandas_udf(result_schema, PandasUDFType.GROUPED_MAP) metode će biti zadužene za distribuirano treniranje i formiranje predikcija. Na kraju, delovi predikcija biće sumirani u driver procesu.
def transform_time_series_forecasting(dataframe, spark):
df_time_series_data = dataframe.filter(dataframe.country.isin(["Serbia", "Croatia", "Slovenia", "Montenegro"]))
df_time_series_data = df_time_series_data.select(["date", "confirmed", "country"]) \
.drop_duplicates() \
.orderBy("date")
df_time_series_data = df_time_series_data.withColumnRenamed("date", "ds")
df_time_series_data = df_time_series_data.withColumnRenamed("confirmed", "y")
df_forecast = (
df_time_series_data
.groupBy('country')
.apply(distributed_model_prediction)
)
return df_forecast
@pandas_udf(result_schema, PandasUDFType.GROUPED_MAP)
def distributed_model_prediction(history_pd):
history_pd['ds'] = pd.to_datetime(history_pd['ds'])
# make the model
prophet_model = Prophet()
prophet_model.fit(history_pd)
future_pd = prophet_model.make_future_dataframe(
periods=90,
freq='d',
include_history=True
)
# make predictions
results_pd = prophet_model.predict(future_pd)
results_pd["country"] = history_pd["country"].iloc[0]
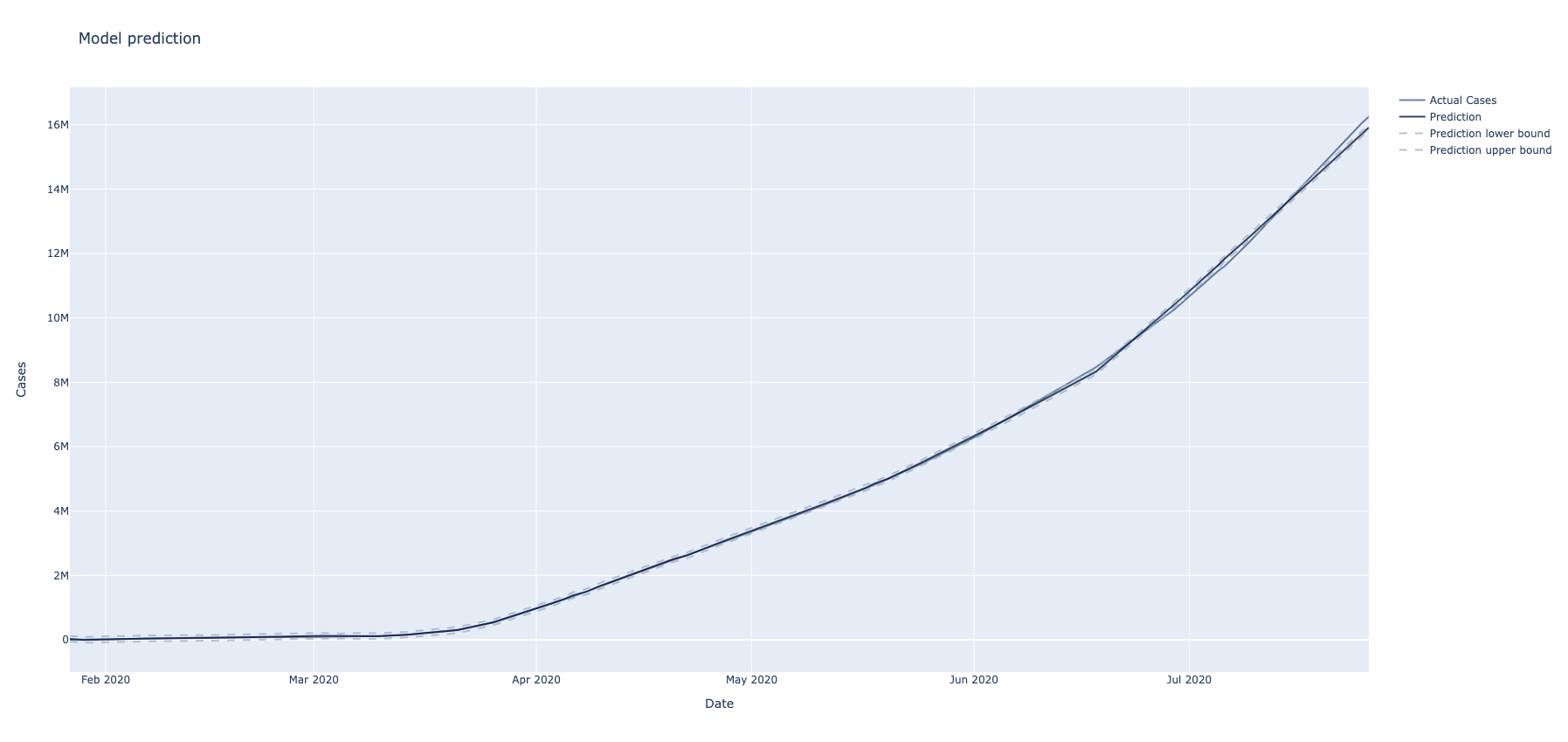
return pd.DataFrame(results_pd, columns=result_schema.fieldNames())Nakon definisanja podataka za treniranje modela, potrebno je odraditi i samo treniranje koje je distribuirano (po državama). Primenom doobijenih modela dobija se gornja i donja granica predikcije, kao i kriva koja predstavlja predviđeni napredak pandemije.
Diagnosis of COVID-19 and its clinical spectrum @Kaggle
Ovaj dataset sadrži anonimne laboratorijske nalaze pacijenata bolnice iz São Paula. Laboratirijski nalazi su širokog spektra i opisuju izmerene nivoe različitih parametara u krvi. Pored laboratorijskih nalaza prisutne su i informacije o pozitivnom/negativnom rezultatu testiranja pacijenata.
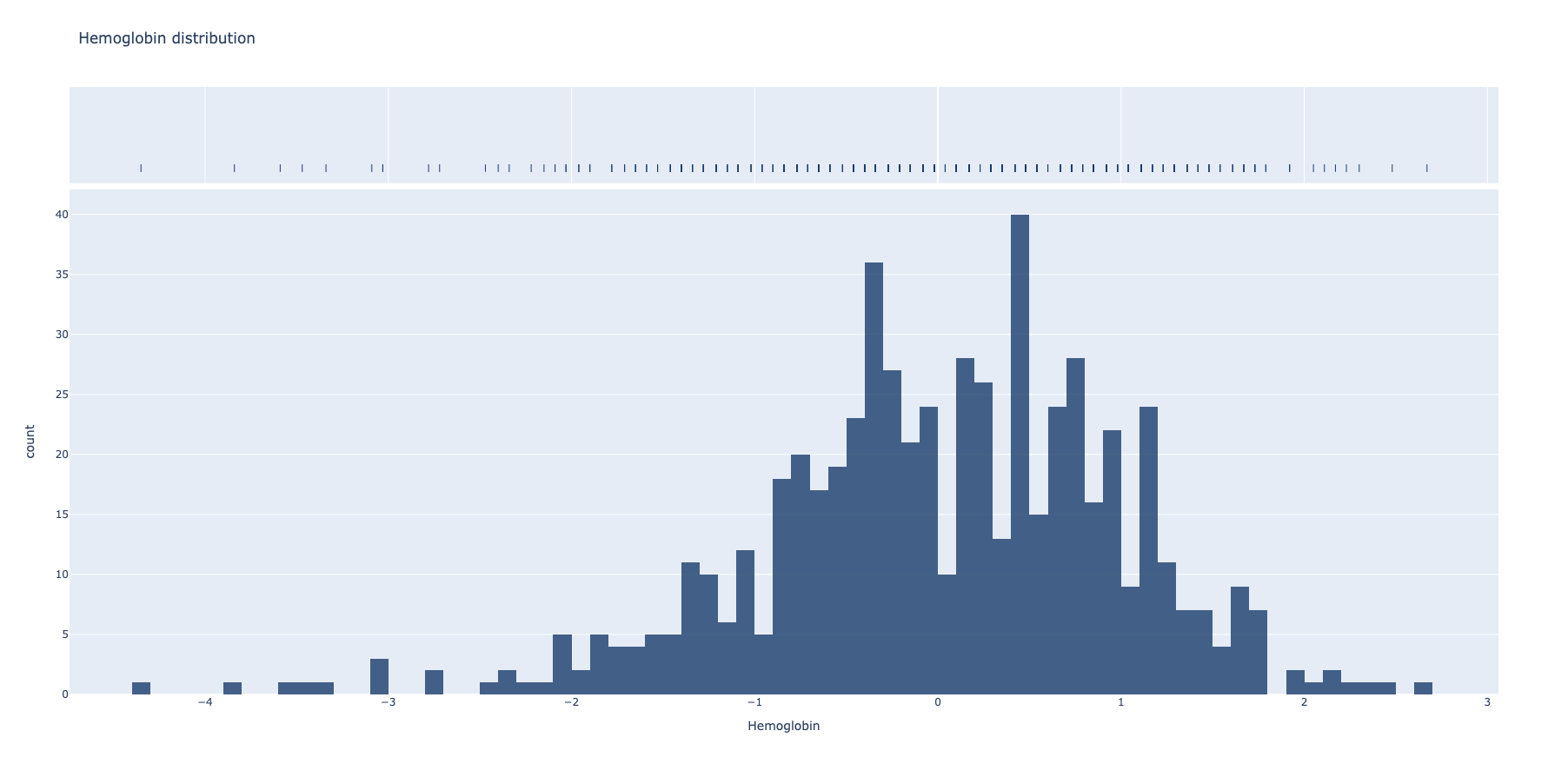
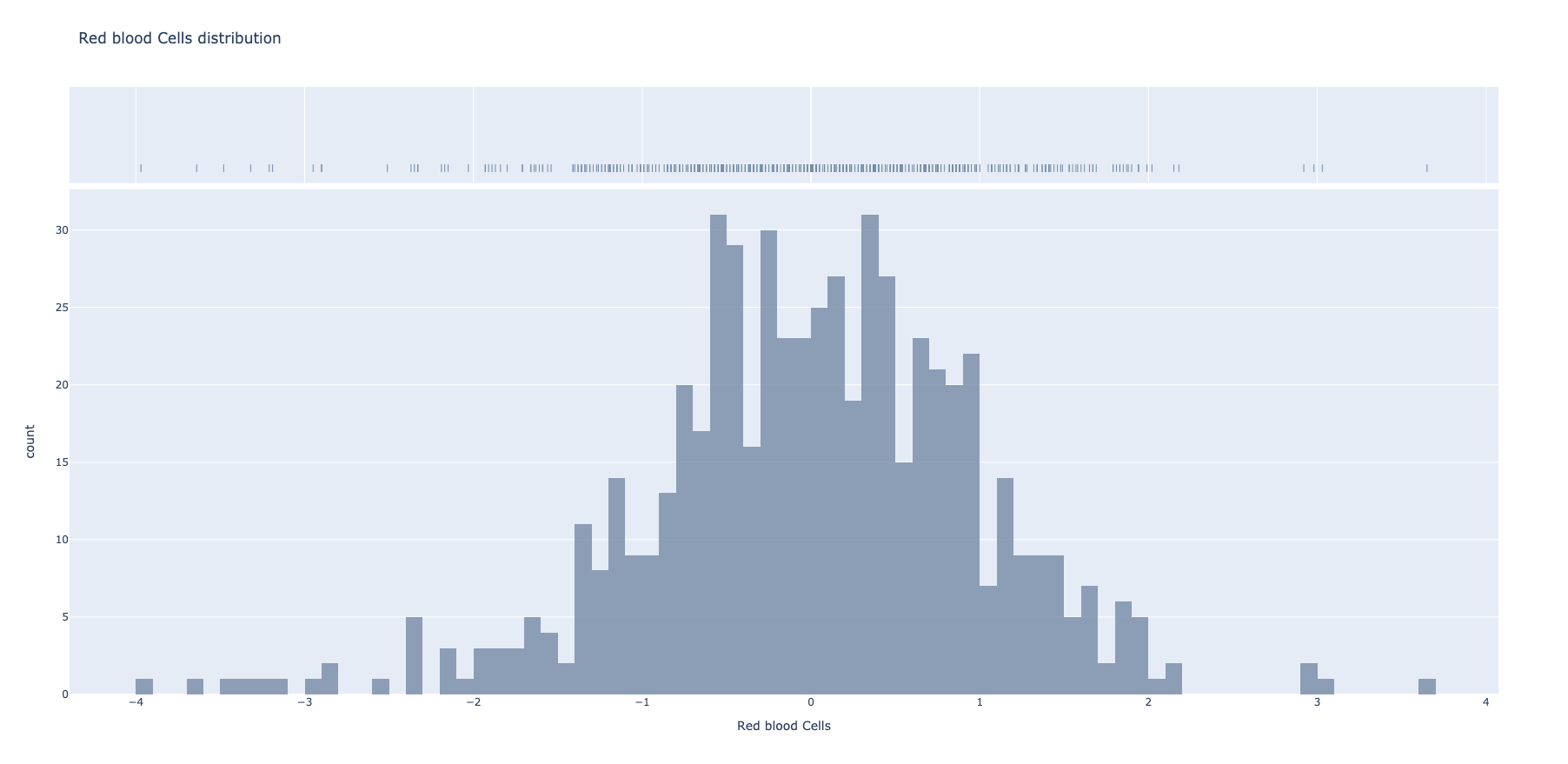
Nakon uvodnih transformacija i normalizacije dataseta, poput popunjavanja nepoznatih vrednosti, selekcijom i tranformacijom može se doći do distribucije pomenutih parametara.
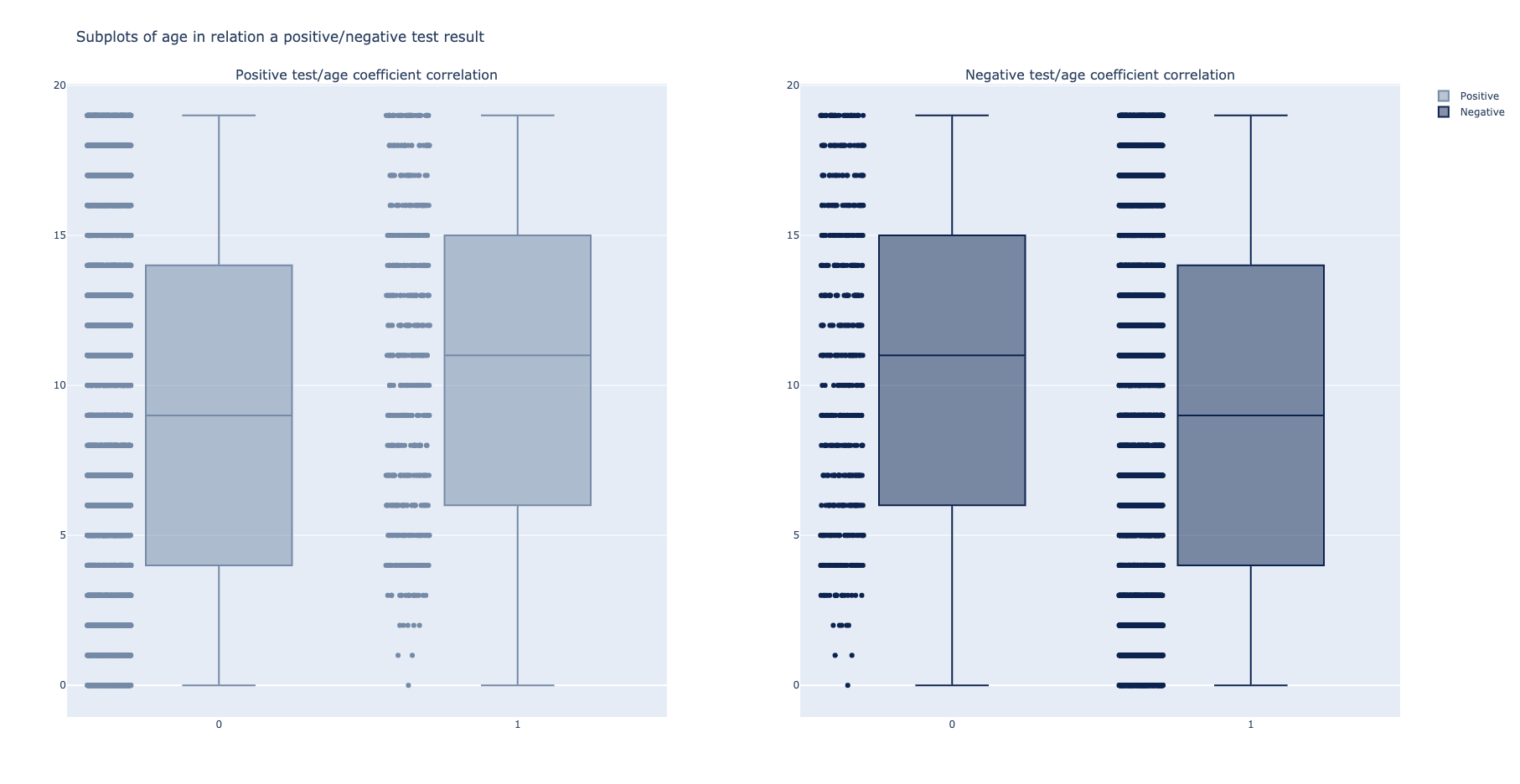
def transform_hemoglobin_red_blood_cells_values(dataframe):
dataframe = dataframe.fillna(0)
dataframe = dataframe.replace("nan", "0")
dataframe = dataframe.withColumn("Hemoglobin", dataframe["Hemoglobin"].cast(IntegerType()))
df_hemoglobin = dataframe.select("Hemoglobin")
df_red_bloof_cells = dataframe.select("Red blood Cells").withColumn("Red blood Cells", func.round(dataframe["Red blood Cells"], 2))
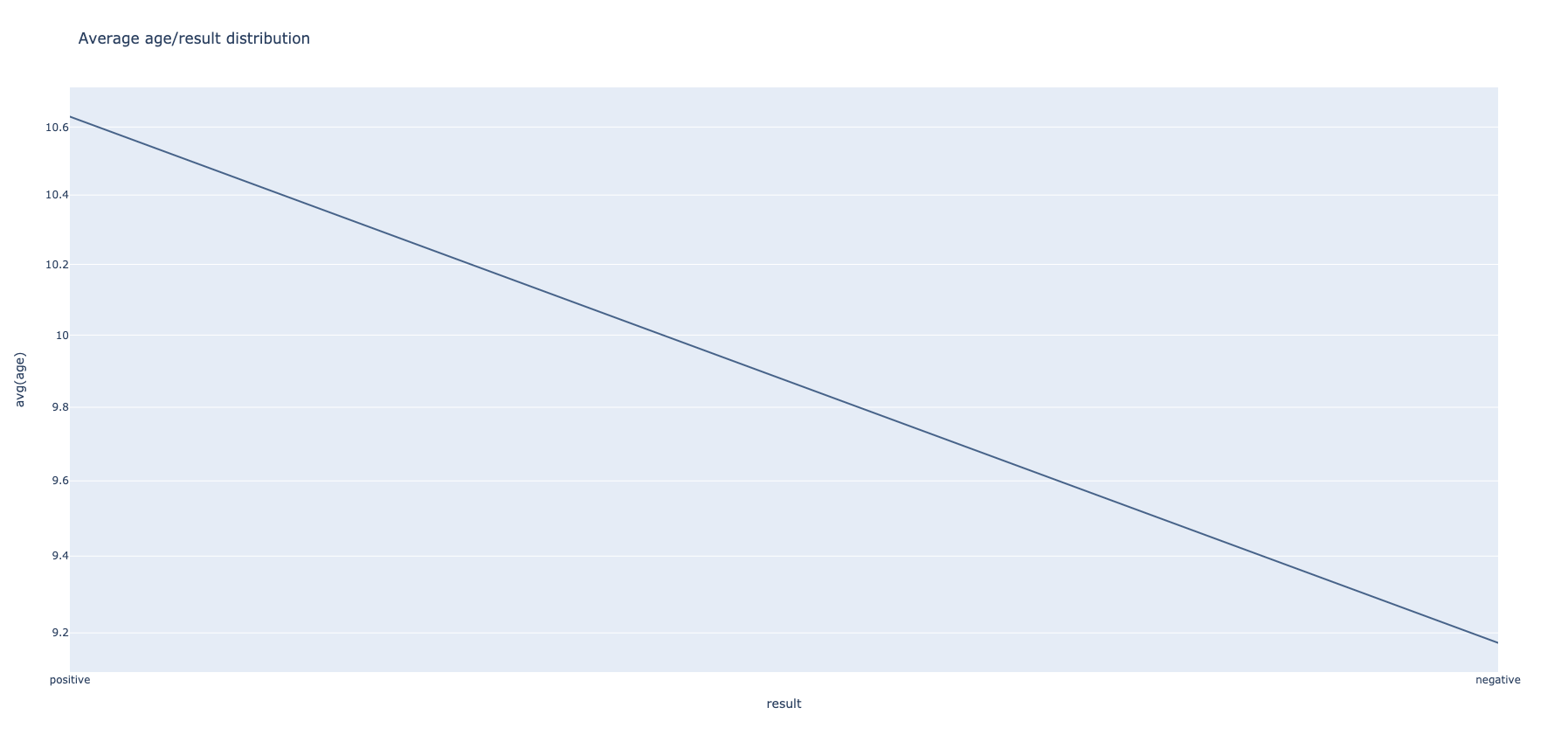
...Prva transformacija predstavlja odnos srednje vrednosti starosti pacijenta i verovatnoće pozitivnog testa. Polazi se od grupacije po rezultatu testa a zatim se nalaze agregacije vrednosti uzrasta.
def transform_aggregate(dataframe, sql_context):
df_age_select = dataframe.select(func.col("SARS-Cov-2 exam result").alias("result"), func.col('Patient age quantile').alias('age'))
df_age_select.write.mode('overwrite').option("header", "true").save("temporary.parquet",format="parquet")
df_sql = sql_context.sql("SELECT * FROM parquet.`./temporary.parquet`")
df_aggregate = df_sql.groupBy("result").agg(func.max("age"), func.avg("age"))
...Druga transformacija primenjuje udf funkcije za označavanje pozitvnih/negativnih pacijenata i formira dva podskupa koja sadrže kolone pozitivnosti na COVID i uzrasta.
def transform_age_relations(dataframe, sql_context):
udf_function_positive = func.udf(is_positive, StringType())
udf_function_negative = func.udf(is_negative, StringType())
df_age = dataframe.select(func.col("SARS-Cov-2 exam result").alias("result"), func.col('Patient age quantile').alias('age'))
df_age_positive = df_age.withColumn("positive", udf_function_positive("result"))
df_age_negative = df_age.withColumn("negative", udf_function_negative("result"))
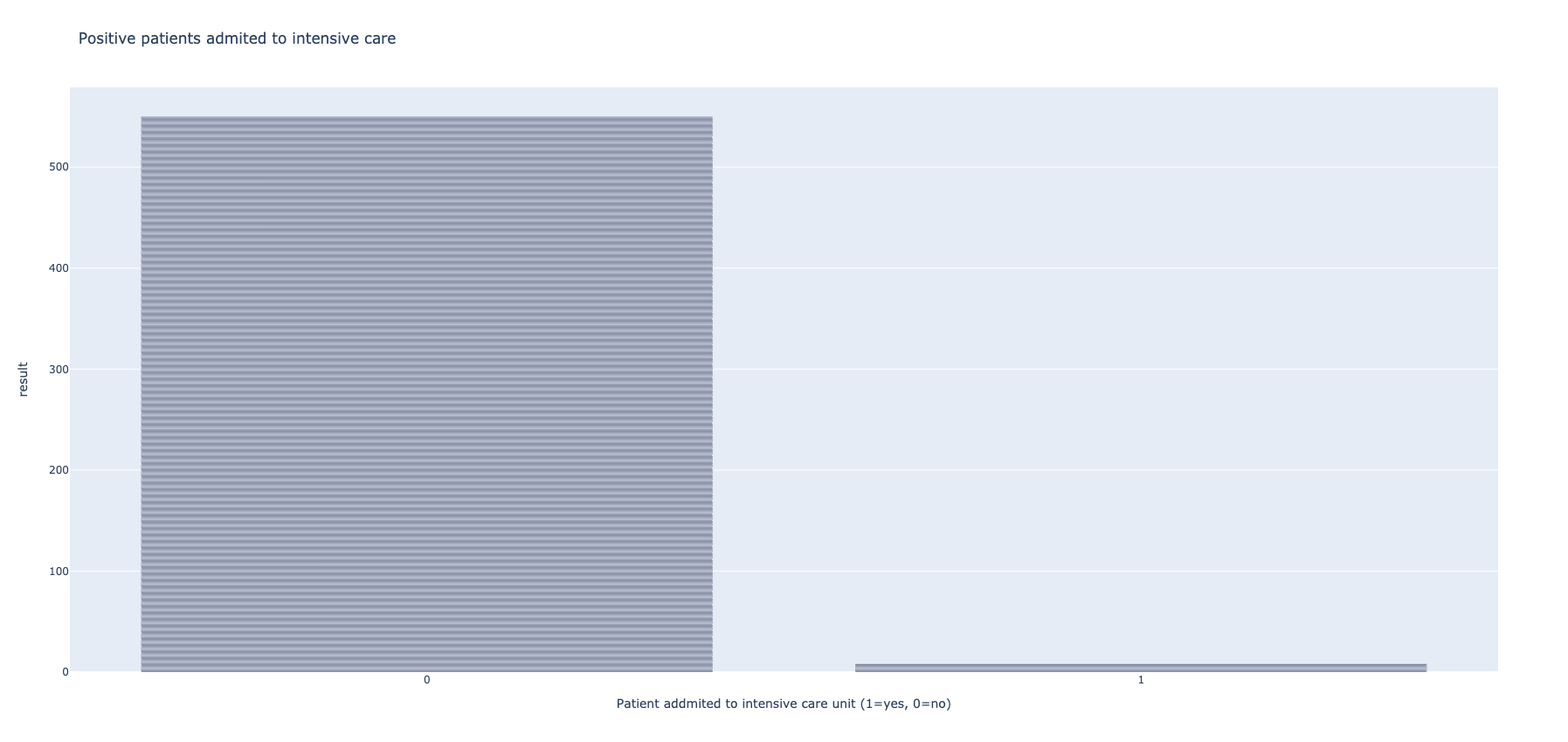
...Primenom slične tehnike kao u prošlom primeru, udf funkcijama se označavaju pacijenti koji su pozitivni, a zatim se vrši prikaz onih pacijenata koji su zbrinuti na intenzivnoj nezi.
def transform_care_relations(dataframe, sql_context):
udf_function_to_numeric = func.udf(negative_positive_to_numeric, IntegerType())
df_transformed_numeric = dataframe.withColumn("result", udf_function_to_numeric("SARS-Cov-2 exam result"))
df_transformed_positive = df_transformed_numeric.filter(df_transformed_numeric.result == 1)
df_transformed_positive_display = df_transformed_positive
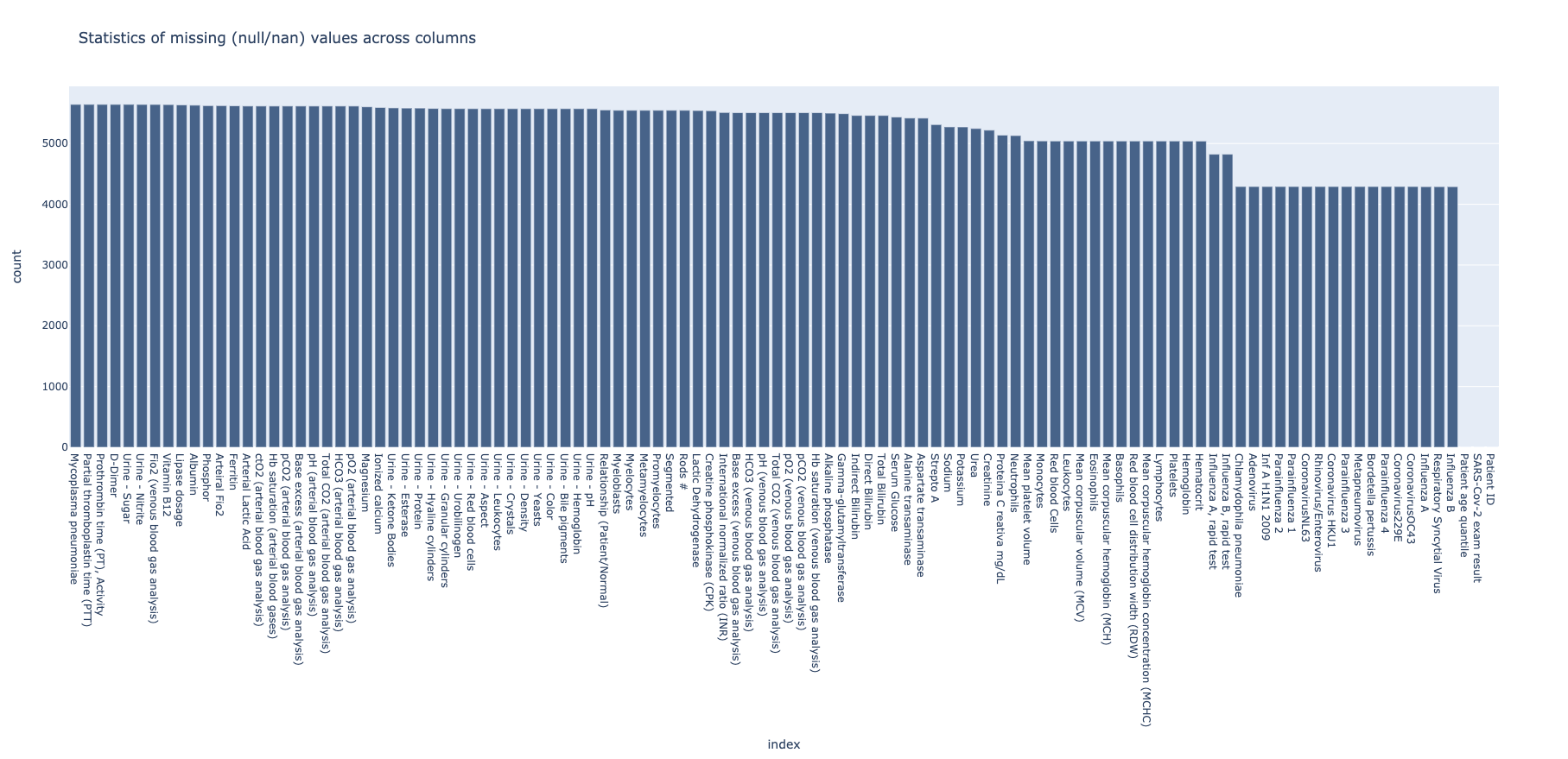
...Potrebno je pre svega razumeti koje stavke dataseta su nepotpune i eliminisati kolone koje ne bi doprinele treniranju efikasnih modela.
df_transformed_null = dataframe.select(
[func.count(func.when(func.isnan(c) | func.isnull(c), c)).alias(c) for (c, c_type) in
dataframe.dtypes])Iz priloženih rezultata može se videti da postoji nekoliko kolona koje su zanemarive.
df_transformed = df_transformed.drop("Mycoplasma pneumoniae", "Urine - Sugar",
"Prothrombin time (PT), Activity", "D-Dimer",
"Fio2 (venous blood gas analysis)", "Urine - Nitrite",
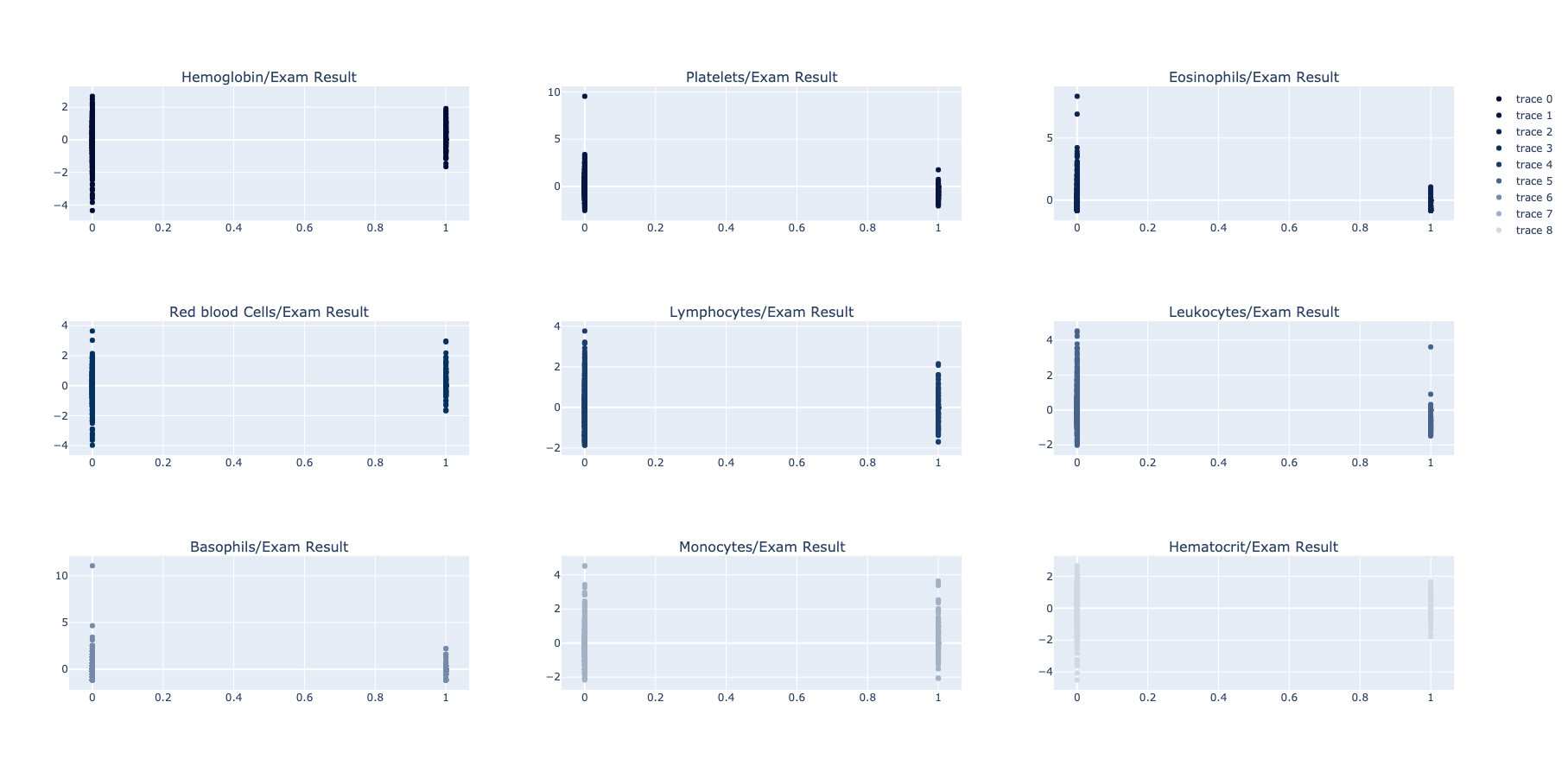
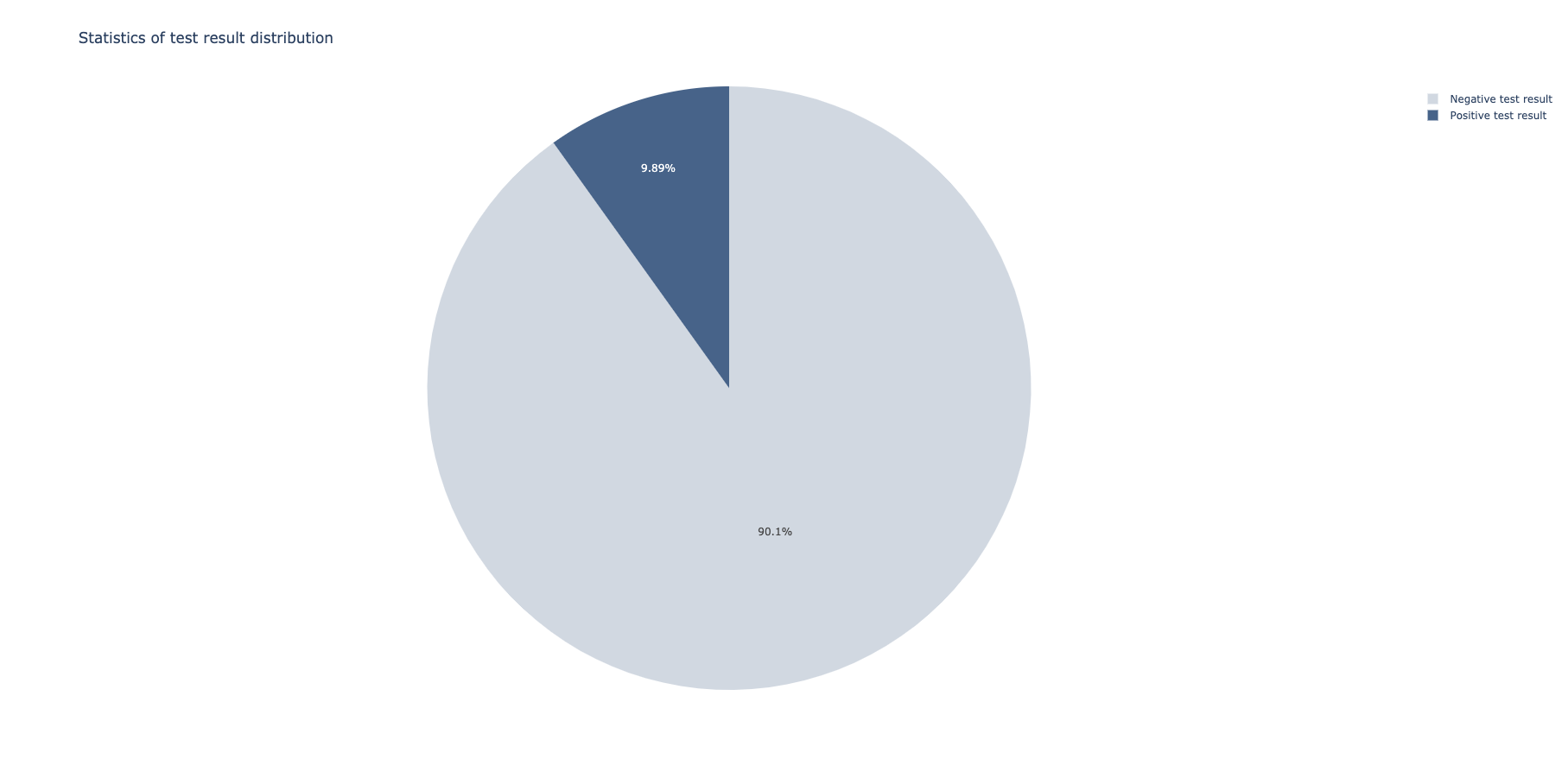
"Vitamin B12")Zatim, možemo prikazati odnose između svih preostalih parametara i pozitivnog/negativnog testa. Takođe, grupacijama možemo prikazati distribuciju rezultata pacijenata.
udf_function_result = func.udf(transform_result, StringType())
df_transformed = dataframe.withColumn("result", udf_function_result("SARS-Cov-2 exam result"))
df_transformed_collected = df_transformed.groupBy('result').count()Na kraju, izgrađuju se različiti modeli nad delom dataseta koji je predodređen za testiranje. Prilikom podele modela, izabran je odnos po kome se 80% ulaznih podataka koristi za treniranje. Biraju se kolone koje će se koristiti kao ulazne i jedna izlazna (u ovom slučaju rezultat testa).
# build the dataset to be used as a rf_model base
outcome_features = ["SARS-Cov-2 exam result"]
required_features = ['Hemoglobin', 'Hematocrit', 'Platelets', 'Eosinophils', 'Red blood Cells', 'Lymphocytes',
'Leukocytes', 'Basophils', 'Monocytes']
df_transformed_model = df_transformed.select(required_features + outcome_features)
assembler = VectorAssembler(inputCols=required_features, outputCol='features')
model_data = assembler.transform(df_transformed_model)
model_data.show()
# split the dataset into train/test subgroups
(training_data, test_data) = model_data.randomSplit([0.8, 0.2], seed=2020)
print("Training Dataset Count: " + str(training_data.count()))
print("Test Dataset Count: " + str(test_data.count()))
# Random Forest classifier
rf = RandomForestClassifier(labelCol='SARS-Cov-2 exam result', featuresCol='features', maxDepth=5)
rf_model = rf.fit(training_data)
rf_predictions = rf_model.transform(test_data)
# Decision Tree Classifier
dt = DecisionTreeClassifier(featuresCol='features', labelCol='SARS-Cov-2 exam result', maxDepth=3)
dt_model = dt.fit(training_data)
dt_predictions = dt_model.transform(test_data)
dt_predictions.select(outcome_features + required_features).show(10)
# Logistic Regression Model
lr = LogisticRegression(featuresCol='features', labelCol='SARS-Cov-2 exam result', maxIter=10)
lr_model = lr.fit(training_data)
lr_predictions = lr_model.transform(test_data)
# Gradient-boosted Tree classifier Model
gb = GBTClassifier(labelCol='SARS-Cov-2 exam result', featuresCol='features')
gb_model = gb.fit(training_data)
gb_predictions = gb_model.transform(test_data)Uspešnost svih modela evaluira se odvojeno, nad preostalim delom dataseta koji je označen za ovu svrhu.