尾崎遼 haruka.ozaki@riken.jp 理化学研究所 情報基盤センター バイオインフォマティクス研究開発ユニット 基礎科学特別研究員
統合データベース講習会:AJACS京都2 (2016/09/02) における講義「NGSデータから新たな知識を導出するための高次解析」の実習用資料です。
Google Chrome (>= 13) のインストールをお願いします
Jupyter Notebook がサポートするブラウザが必要となります。 ※Jupyter Notebookのインストールの必要はありません。
- Chrome >= 13
Safari >= 5Firefox >= 6
(手順)端末のブラウザがサポートしていないバージョンの場合は、以下URLにアクセスしダウンロード・インストールします。
- Chrome https://www.google.com/chrome/browser/desktop/index.html
Safari https://support.apple.com/downloads/safariFirefox https://www.mozilla.org/en-US/firefox/new/
当日までにこちらでアナウンスする予定です。
Chrome の方は "ADVANCED" をクリックし、"Proceed to (unsafe)" をクリックしてアクセスしてください
- 01 - 10 の方: https://52.196.207.243/hub/login
- 11 - 20 の方: https://52.198.132.139/hub/login
- 21 - 30 の方: https://52.196.153.89/hub/login
- 31 - 40 の方: https://52.198.94.14/hub/login
- 41 - 50 の方: https://52.198.127.36/hub/login
実行結果を記録しながら、プログラムを実行できるプログラミング・解析環境です。 ここでは、python、R、terminal (シェルスクリプト)が使える環境を準備してます。
Terminal を開いてください。
deepTools: http://deeptools.readthedocs.io/en/latest/
データのダウンロード
wget https://www.encodeproject.org/files/ENCFF002CRA/@@download/ENCFF002CRA.bed.gz
gunzip ENCFF002CRA.bed.gz
grep chr21 ENCFF002CRA.bed > ENCFF002CRA.chr21.bed
wget https://raw.githubusercontent.com/yuifu/AJACS_Kyoto_2/master/ENCFF831SAH.chr21.bam
wget https://raw.githubusercontent.com/yuifu/AJACS_Kyoto_2/master/ENCFF831SAH.chr21.bam.bai
wget https://raw.githubusercontent.com/yuifu/AJACS_Kyoto_2/master/ENCFF002CRG.chr21.bed
BAMのBigWigファイルへの変換
bamCoverage -b ENCFF831SAH.chr21.bam -o myfile.bw
matrix ファイル(deepTools独自のフォーマット)を作成
computeMatrix reference-point \
--referencePoint center \
-b 1000 -a 1000 \
-R ENCFF002CRG.chr21.bed \
-S myfile.bw \
--skipZeros \
-o matrix_center.gz \
--outFileSortedRegions regions00.bed
aggregation plot の作成
plotProfile -m matrix_center.gz \
-out ExampleProfile1.png \
--plotTitle "Test data profile"
R を開いてください。
Monocle: http://bioconductor.org/packages/release/bioc/html/monocle.html
###################################################
### code chunk number 1: monocle-vignette.Rnw:26-30
###################################################
library(Biobase)
library(knitr)
library(reshape2)
library(ggplot2)
###################################################
### code chunk number 2: init_monocle
###################################################
library(HSMMSingleCell)
library(monocle)
data(HSMM_expr_matrix)
data(HSMM_gene_annotation)
data(HSMM_sample_sheet)
###################################################
### code chunk number 4: build_cell_data_Set
###################################################
pd <- new("AnnotatedDataFrame", data = HSMM_sample_sheet)
fd <- new("AnnotatedDataFrame", data = HSMM_gene_annotation)
HSMM <- newCellDataSet(as.matrix(HSMM_expr_matrix), phenoData = pd, featureData = fd)
###################################################
### code chunk number 5: detect_genes
###################################################
HSMM <- detectGenes(HSMM, min_expr = 0.1)
print(head(fData(HSMM)))
expressed_genes <- row.names(subset(fData(HSMM), num_cells_expressed >= 50))
###################################################
### code chunk number 6: show_pData
###################################################
print(head(pData(HSMM)))
###################################################
### code chunk number 8: lognormal_plot
###################################################
# Log-transform each value in the expression matrix.
L <- log(exprs(HSMM[expressed_genes,]))
# Standardize each gene, so that they are all on the same scale,
# Then melt the data with plyr so we can plot it easily"
melted_dens_df <- melt(t(scale(t(L))))
# Plot the distribution of the standardized gene expression values.
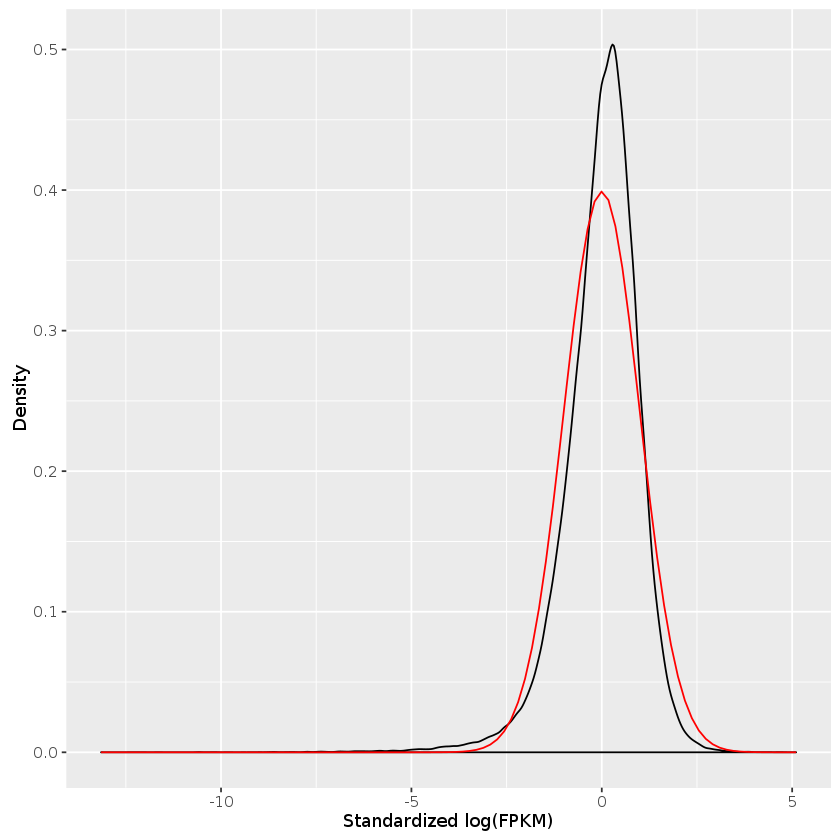
qplot(value, geom="density", data=melted_dens_df) + stat_function(fun = dnorm, size=0.5, color='red') +
xlab("Standardized log(FPKM)") +
ylab("Density")
###################################################
### code chunk number 9: select_genes
###################################################
marker_genes <- row.names(subset(fData(HSMM),
gene_short_name %in% c("MEF2C", "MEF2D", "MYF5", "ANPEP", "PDGFRA",
"MYOG", "TPM1", "TPM2", "MYH2", "MYH3", "NCAM1", "TNNT1", "TNNT2", "TNNC1",
"CDK1", "CDK2", "CCNB1", "CCNB2", "CCND1", "CCNA1", "ID1")))
###################################################
### code chunk number 10: basic_diff
###################################################
diff_test_res <- differentialGeneTest(HSMM[marker_genes,],
fullModelFormulaStr="expression~Media")
# Select genes that are significant at an FDR < 10%
sig_genes <- subset(diff_test_res, qval < 0.1)
# Attach the HUGO symbols and other featureData for these genes
sig_genes <- merge(fData(HSMM), sig_genes, by="row.names")
sig_genes[,c("gene_short_name", "pval", "qval")]
###################################################
### code chunk number 11: plot_myog_jitter
###################################################
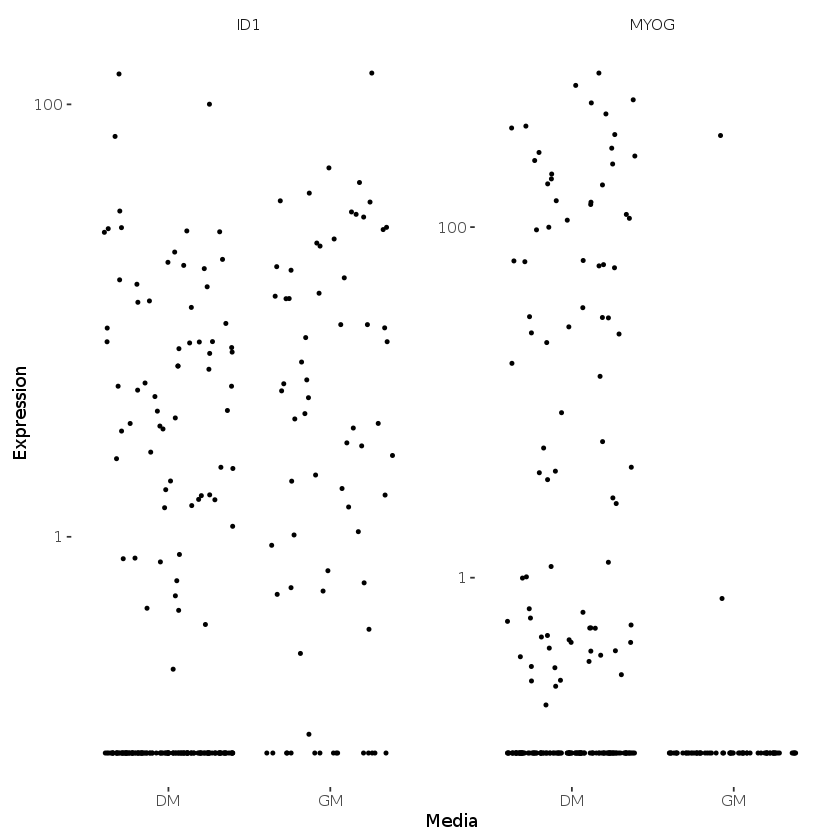
MYOG_ID1 <- HSMM[row.names(subset(fData(HSMM),
gene_short_name %in% c("MYOG", "ID1"))),]
plot_genes_jitter(MYOG_ID1, grouping="Media", ncol=2)
###################################################
### code chunk number 13: select_ordering_cells
###################################################
ordering_genes <- row.names (subset(diff_test_res, qval < 0.1))
#Only use genes are detectably expressed in a sufficient number of cells
ordering_genes <- intersect(ordering_genes, expressed_genes)
###################################################
### code chunk number 14: set_ordering_filter
###################################################
HSMM <- setOrderingFilter(HSMM, ordering_genes)
###################################################
### code chunk number 15: reduce_dimension
###################################################
HSMM <- reduceDimension(HSMM, use_irlba=FALSE)
###################################################
### code chunk number 16: order_cells
###################################################
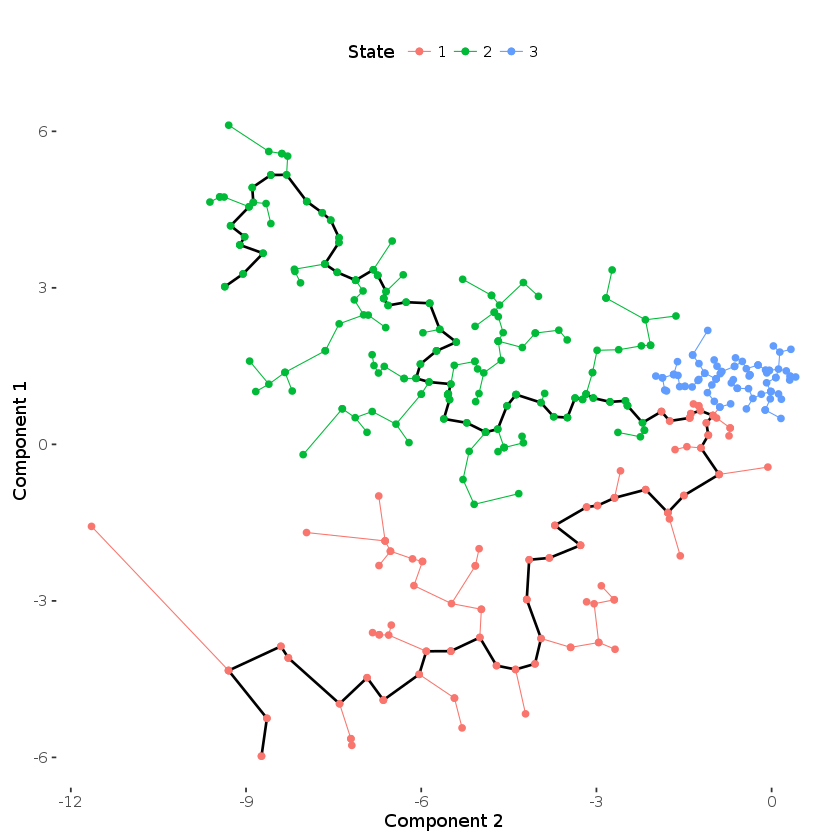
HSMM <- orderCells(HSMM, num_paths=2, reverse=TRUE)
###################################################
### code chunk number 17: plot_ordering_mst
###################################################
plot_spanning_tree(HSMM)
###################################################
### code chunk number 18: plot_markers
###################################################
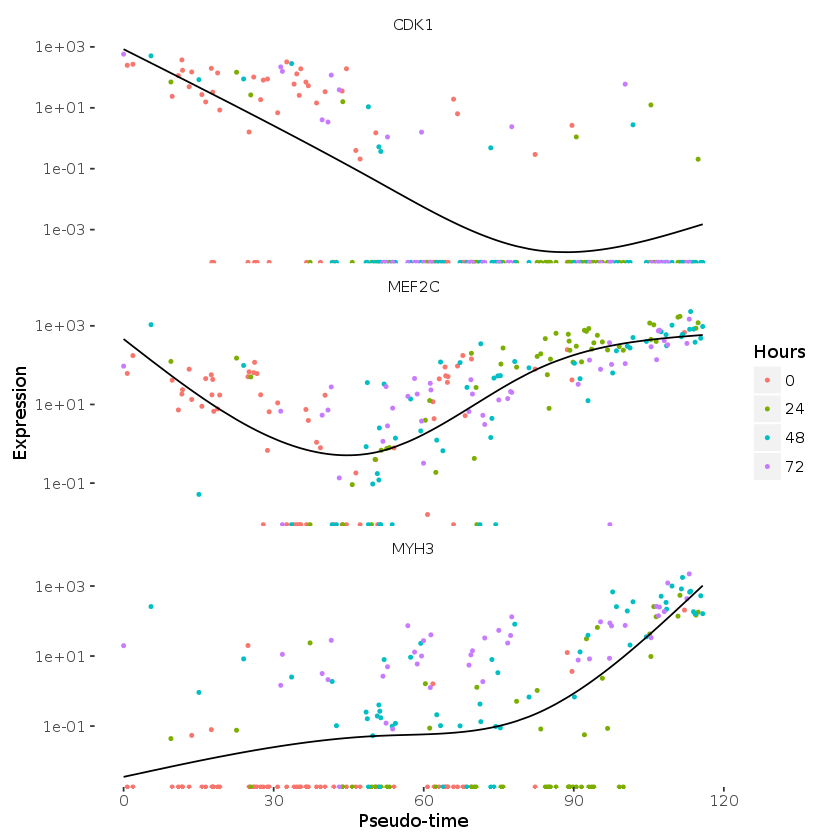
HSMM_filtered <- HSMM[expressed_genes, pData(HSMM)$State != 3]
my_genes <- row.names(subset(fData(HSMM_filtered),
gene_short_name %in% c("CDK1", "MEF2C", "MYH3")))
cds_subset <- HSMM_filtered[my_genes,]
plot_genes_in_pseudotime(cds_subset, color_by="Hours")
###################################################
### code chunk number 19: setup_test_genes
###################################################
to_be_tested <- row.names(subset(fData(HSMM),
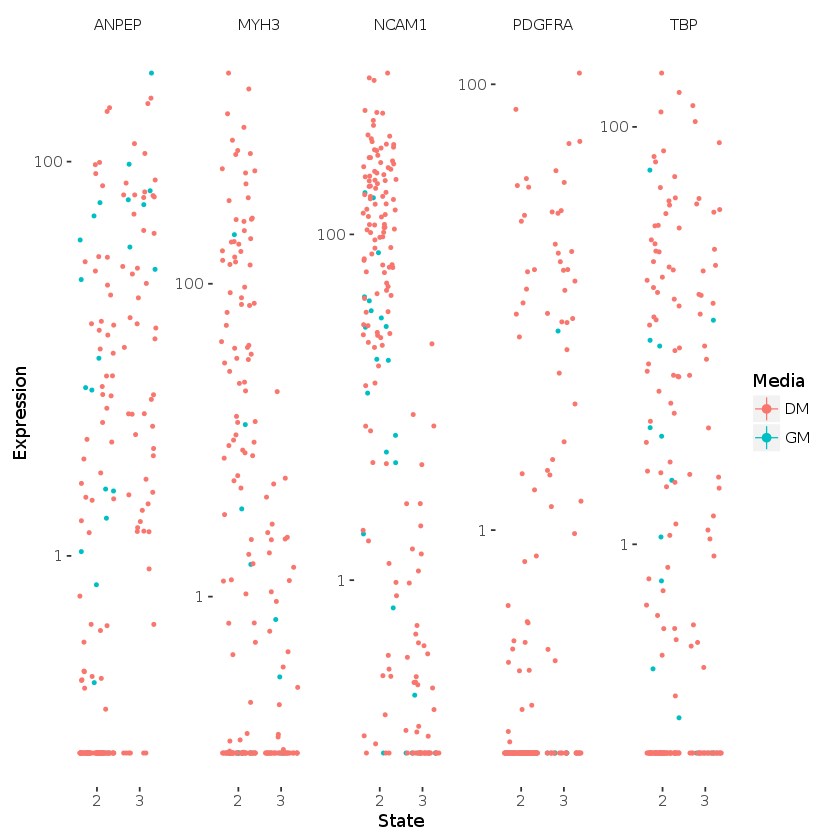
gene_short_name %in% c("TBP", "MYH3", "NCAM1", "PDGFRA", "ANPEP")))
cds_subset <- HSMM[to_be_tested, pData(HSMM)$State != 1]
###################################################
### code chunk number 20: all_in_one_test
###################################################
diff_test_res <- differentialGeneTest(cds_subset, fullModelFormulaStr="expression~State")
diff_test_res <- merge(fData(HSMM), diff_test_res, by="row.names")
diff_test_res[,c("gene_short_name", "pval", "qval")]
###################################################
### code chunk number 21: jitter_plot_diff_res
###################################################
plot_genes_jitter(cds_subset, color_by="Media", nrow=1, ncol=NULL, plot_trend=TRUE)
###################################################
### code chunk number 23: setup_test_genes_pt
###################################################
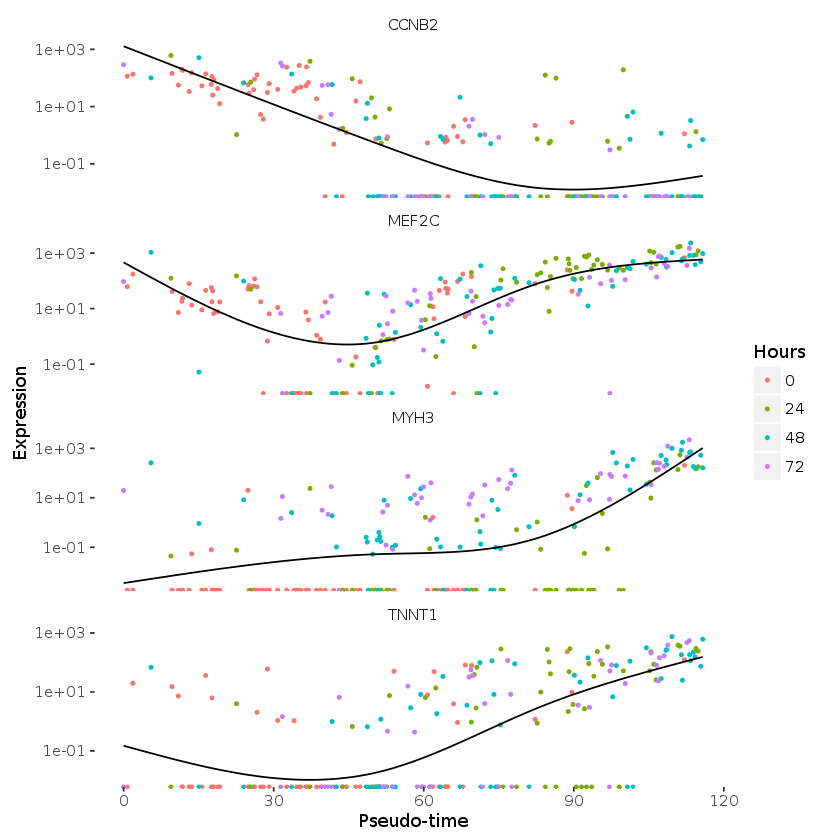
to_be_tested <- row.names(subset(fData(HSMM),
gene_short_name %in% c("MYH3", "MEF2C", "CCNB2", "TNNT1")))
cds_subset <- HSMM[to_be_tested, pData(HSMM)$State != 3]
###################################################
### code chunk number 24: piecewise_test_pt
###################################################
diff_test_res <- differentialGeneTest(cds_subset, fullModelFormulaStr="expression~sm.ns(Pseudotime)")
###################################################
### code chunk number 25: all_in_one_test_pt
###################################################
diff_test_res <- merge(fData(HSMM), diff_test_res, by="row.names")
diff_test_res[,c("gene_short_name", "pval", "qval")]
###################################################
### code chunk number 26: plot_diff_res_pt
###################################################
plot_genes_in_pseudotime(cds_subset, color_by="Hours")
###################################################
### code chunk number 27: plot_diff_res_multi
###################################################
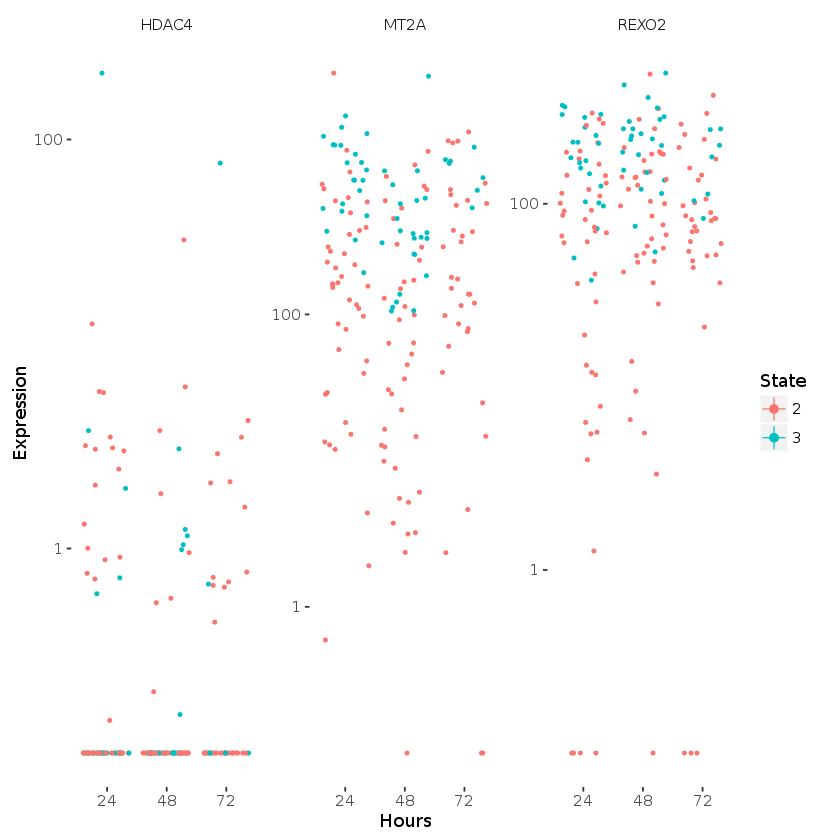
to_be_tested <- row.names(subset(fData(HSMM),
gene_short_name %in% c("MT2A", "REXO2", "HDAC4")))
cds_subset <- HSMM[to_be_tested, pData(HSMM)$Media == "DM" & pData(HSMM)$State != 1]
diff_test_res <- differentialGeneTest(cds_subset,
fullModelFormulaStr="expression~State * Hours",
reducedModelFormulaStr="expression~Hours")
diff_test_res <- merge(fData(cds_subset), diff_test_res, by="row.names")
diff_test_res[,c("gene_short_name", "pval", "qval")]
plot_genes_jitter(cds_subset, grouping="Hours", color_by="State", plot_trend=TRUE) + facet_wrap( ~ feature_label, scales="free_y")
###################################################
### code chunk number 28: fit_full_model_for_cluster
###################################################
sampled_gene_cds <- HSMM_filtered[sample(nrow(fData(HSMM_filtered)), 100),]
full_model_fits <- fitModel(sampled_gene_cds, modelFormulaStr="expression~sm.ns(Pseudotime, df=3)")
###################################################
### code chunk number 29: extract_response_curve
###################################################
expression_curve_matrix <- responseMatrix(full_model_fits)
dim(expression_curve_matrix)
###################################################
### code chunk number 30: cluster_and_plot
###################################################
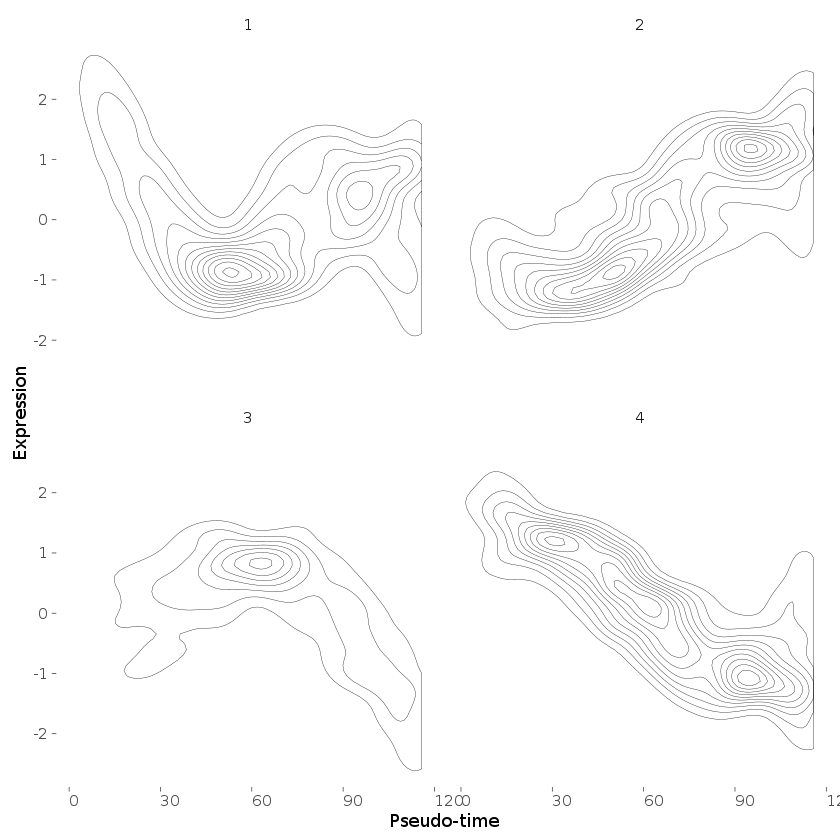
clusters <- clusterGenes(expression_curve_matrix, k=4)
plot_clusters(HSMM_filtered[ordering_genes,], clusters)
###################################################
### code chunk number 31: citation
###################################################
citation("monocle")
###################################################
### code chunk number 32: sessionInfo
###################################################
sessionInfo()