Na przestrzeni ostatnich lat zauważono stopniowy spadek rozmiaru śledzia oceanicznego wyławianego w Europie. Do analizy zebrano pomiary śledzi i warunków w jakich żyją z ostatnich 60 lat. Dane były pobierane z połowów komercyjnych jednostek. W ramach połowu jednej jednostki losowo wybierano od 50 do 100 sztuk trzyletnich śledzi.
Po przeanalizowaniu danych dochodzimy do wniosku, że wzrost natężenia połowów oraz wzrost temperatury wody mają wpływ na spadek rozmiaru śledzia oceanicznego wyławianego w Europie. Spadek rozmiaru nie jest jednak niepokojący, ponieważ jego wartość z końcowego okresu pomiarów jest zbliżona do wartości początkowych. Ponadto, wyniki obarczone są dość dużą niedokładnością z uwagi na szum w danych – podejrzanie podobne wartości atrybutów oraz brakujące wartości. Możliwe, że zbiór danych z mniejszą liczbą szumu pozwoliłby na dokładniejszą analizę i wykazanie mocniejszego związku między natężeniami połowów i temperaturą a rozmiarem śledzi.
library(dplyr)
library(ggplot2)
library(corrplot)
library(plotly)
library(caret)
library(tidyr)
set.seed(0)
download.file("http://www.cs.put.poznan.pl/alabijak/emd/projekt/sledzie.csv", "sledzie.csv")
df <- read.csv(
"sledzie.csv",
header=TRUE,
na.strings="?",
colClasses=c("xmonth"="factor")
) %>%
rename(time=X)
dim(df)
## [1] 52582 16
names(df)
## [1] "time" "length" "cfin1" "cfin2" "chel1" "chel2" "lcop1"
## [8] "lcop2" "fbar" "recr" "cumf" "totaln" "sst" "sal"
## [15] "xmonth" "nao"
Wszystkie atrybuty są numeryczne, poza atrybutem xmonth, który jest kategoryczny. Dane są ułożone chronologicznie i posortowane według atrybutu time. Jednak nie znamy odstępów między poszczególnymi połowami, atrybut time zachowuje jedynie porządek w zbiorze.
summary(df)
## time length cfin1 cfin2
## Min. : 0 Min. :19.0 Min. : 0.0000 Min. : 0.0000
## 1st Qu.:13145 1st Qu.:24.0 1st Qu.: 0.0000 1st Qu.: 0.2778
## Median :26290 Median :25.5 Median : 0.1111 Median : 0.7012
## Mean :26290 Mean :25.3 Mean : 0.4458 Mean : 2.0248
## 3rd Qu.:39436 3rd Qu.:26.5 3rd Qu.: 0.3333 3rd Qu.: 1.7936
## Max. :52581 Max. :32.5 Max. :37.6667 Max. :19.3958
## NA's :1581 NA's :1536
## chel1 chel2 lcop1 lcop2
## Min. : 0.000 Min. : 5.238 Min. : 0.3074 Min. : 7.849
## 1st Qu.: 2.469 1st Qu.:13.427 1st Qu.: 2.5479 1st Qu.:17.808
## Median : 5.750 Median :21.673 Median : 7.0000 Median :24.859
## Mean :10.006 Mean :21.221 Mean : 12.8108 Mean :28.419
## 3rd Qu.:11.500 3rd Qu.:27.193 3rd Qu.: 21.2315 3rd Qu.:37.232
## Max. :75.000 Max. :57.706 Max. :115.5833 Max. :68.736
## NA's :1555 NA's :1556 NA's :1653 NA's :1591
## fbar recr cumf totaln
## Min. :0.0680 Min. : 140515 Min. :0.06833 Min. : 144137
## 1st Qu.:0.2270 1st Qu.: 360061 1st Qu.:0.14809 1st Qu.: 306068
## Median :0.3320 Median : 421391 Median :0.23191 Median : 539558
## Mean :0.3304 Mean : 520366 Mean :0.22981 Mean : 514973
## 3rd Qu.:0.4560 3rd Qu.: 724151 3rd Qu.:0.29803 3rd Qu.: 730351
## Max. :0.8490 Max. :1565890 Max. :0.39801 Max. :1015595
##
## sst sal xmonth nao
## Min. :12.77 Min. :35.40 8 : 9920 Min. :-4.89000
## 1st Qu.:13.60 1st Qu.:35.51 10 : 7972 1st Qu.:-1.89000
## Median :13.86 Median :35.51 7 : 6922 Median : 0.20000
## Mean :13.87 Mean :35.51 9 : 5714 Mean :-0.09236
## 3rd Qu.:14.16 3rd Qu.:35.52 6 : 4218 3rd Qu.: 1.63000
## Max. :14.73 Max. :35.61 5 : 3736 Max. : 5.08000
## NA's :1584 (Other):14100
Sąsiednie wiersze mają często jednakowe atrybuty. Ma to uzasadnienie w tym, że było wiele pomiarów z jednego połowu, a poszczególne ryby różnią się długością.
knitr::kable(head(df, 10), caption = "Table with kable")
| time | length | cfin1 | cfin2 | chel1 | chel2 | lcop1 | lcop2 | fbar | recr | cumf | totaln | sst | sal | xmonth | nao |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 23.0 | 0.02778 | 0.27785 | 2.46875 | NA | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 1 | 22.5 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 2 | 25.0 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 3 | 25.5 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 4 | 24.0 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 5 | 22.0 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | NA | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 6 | 24.0 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 7 | 23.5 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 8 | 22.5 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
| 9 | 22.5 | 0.02778 | 0.27785 | 2.46875 | 21.43548 | 2.54787 | 26.35881 | 0.356 | 482831 | 0.3059879 | 267380.8 | 14.30693 | 35.51234 | 7 | 2.8 |
Do dalszej analizy odfiltrowujemy wiersze z niepełnymi danymi. To około 20% danych. Pozostałe 42,000 powinny wystarczyć do dalszej analizy. Dodatkowo przygotowujemy dane wyłącznie numeryczne do analizy korelacji atrybutów.
df_numeric <- subset(df, select=-c(time, xmonth))
df_numeric_without_na <- df_numeric[complete.cases(df_numeric),]
df_without_na <- df[complete.cases(df),]
dim(df_without_na)
## [1] 42488 16
Po spojrzeniu na dane oraz krótkim zastanowieniu doszliśmy do wniosku, że nie powinniśmy przewidywać długości pojedynczego śledzia, a jego średnią długość. Bez grupowania danych te same zestawy atrybutów zwracają różne długości śledzia, co jest zjawiskiem nieporządanym w algorytmach uczenia maszynowego. Z uwagi na brak atrybutu opisującego dokładny czas, musimy pogrupować przykłady na podstawie wartości pozostałych atrybutów.
Najpierw spróbowaliśmy pogrupować przykłady z użyciem metody group_by. Ku naszemu zdziwieniu okazało się, że przykłady znacznie oddalone od siebie w zbiorze danych mają dokładnie te same wartości atrybutów. Według nas jest to zbyt mało prawdopodobne i oznacza występowanie błędów w zbiorze danych.
df_grouped_globally <- df_without_na %>%
group_by(cfin1, cfin2, chel1, chel2, cumf, lcop1, lcop2, fbar, recr, totaln, sst, sal, nao) %>%
summarise(
avg_length = mean(length),
min_time = min(time),
max_time = max(time)
)
dim(df_grouped_globally)
## [1] 106 16
df_grouped_globally <- df_grouped_globally %>% ungroup() %>% select(min_time, max_time, avg_length) %>% arrange(min_time)
head(df_grouped_globally)
## # A tibble: 6 x 3
## min_time max_time avg_length
## <int> <int> <dbl>
## 1 1 9143 23.0
## 2 221 52580 24.1
## 3 290 40730 25.8
## 4 781 38018 26.2
## 5 864 52579 25.5
## 6 975 4366 25.3
df_without_na %>% filter(time == 1)
## time length cfin1 cfin2 chel1 chel2 lcop1 lcop2 fbar
## 1 1 22.5 0.02778 0.27785 2.46875 21.43548 2.54787 26.35881 0.356
## recr cumf totaln sst sal xmonth nao
## 1 482831 0.3059879 267380.8 14.30693 35.51234 7 2.8
df_without_na %>% filter(time == 9143)
## time length cfin1 cfin2 chel1 chel2 lcop1 lcop2 fbar
## 1 9143 21 0.02778 0.27785 2.46875 21.43548 2.54787 26.35881 0.356
## recr cumf totaln sst sal xmonth nao
## 1 482831 0.3059879 267380.8 14.30693 35.51234 4 2.8
Drugim podejściem grupowania było złączenie przykładów, które mają te same wartości atrybutów oraz występują obok siebie. Takie grupowanie wydaje się poprawne. Długości śledzi w grupach zostały uśrednione. Po takim grupowaniu zostało nam 2558 przykładów.
df_grouped <- df_numeric_without_na[FALSE,]
last_group_lengths <- vector()
last_row <- NULL
regression_col_names <- c("cfin1", "cfin2", "chel1", "chel2", "cumf", "lcop1", "lcop2", "fbar", "recr", "totaln", "sst", "sal", "nao")
for (row_i in 1:nrow(df_without_na)) {
row <- df_numeric_without_na[row_i,]
if (is.null(last_row) || all(last_row[regression_col_names] == row[regression_col_names])) {
} else {
last_row$length <- mean(last_group_lengths)
df_grouped[nrow(df_grouped) + 1,] <- last_row
last_group_lengths <- vector()
}
last_group_lengths <- c(last_group_lengths, row$length)
last_row <- row
}
if (length(last_group_lengths) > 0) {
last_row$length <- mean(last_group_lengths)
df_grouped[nrow(df_grouped) + 1,] <- last_row
}
dim(df_numeric_without_na)
## [1] 42488 14
dim(df_grouped)
## [1] 2558 14
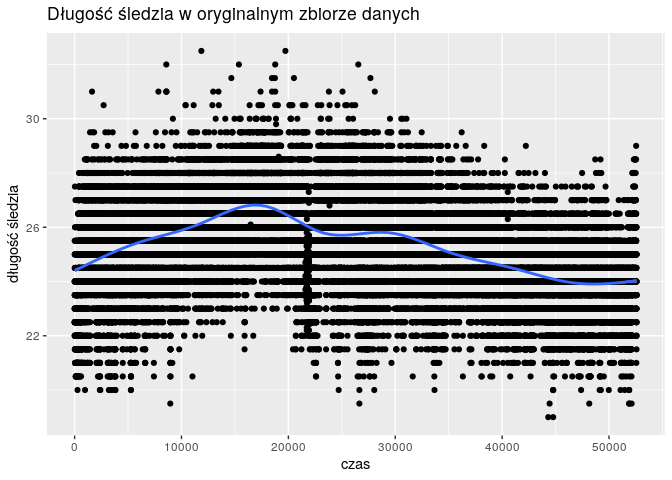
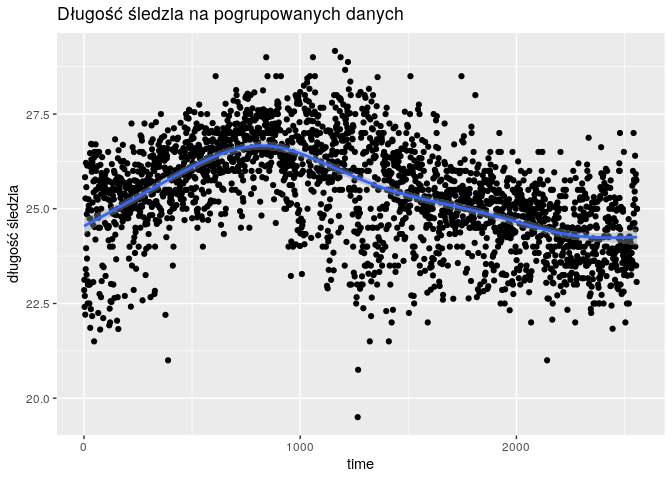
Grupowanie danych sprawiło, że wykres zmienności długości śledzi wygląda “lepiej”. Punkty pomiarowe są bliżej linii trendu i nie występuje już tak silna dyskretyzacja wartości długości.
qplot(
1:nrow(df),
df$length
) +
geom_point(alpha = 0.02) +
geom_smooth() +
xlab("czas") +
ylab("długość śledzia") +
ggtitle("Długość śledzia w oryginalnym zbiorze danych")
qplot(
1:nrow(df_grouped),
df_grouped$length
) +
geom_point(alpha = 0.2) +
geom_smooth() +
xlab("time") +
ylab("długość śledzia") +
ggtitle("Długość śledzia na pogrupowanych danych")
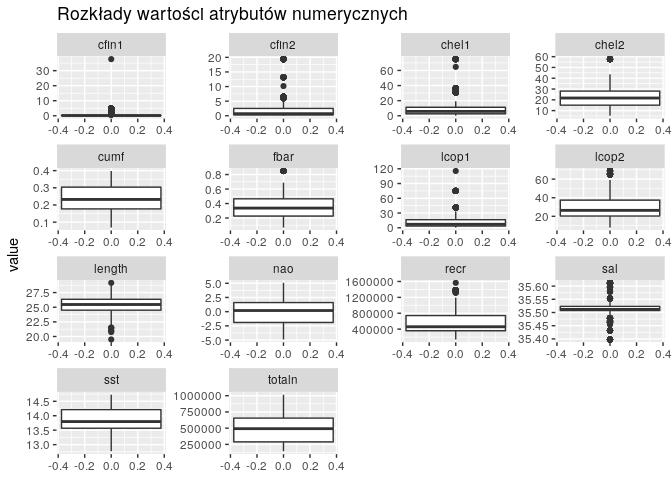
Następnym etapem analizy jest przyjrzenie się rozkładom wartości poszczególnych atrybutów. Dokonaliśmy tego z użyciem wykresów pudełkowych.
df_grouped %>%
gather() %>%
ggplot(aes(y=value)) +
facet_wrap(~ key, scales="free") +
geom_boxplot() +
labs(title = "Rozkłady wartości atrybutów numerycznych")
Na niektórych wykresach występuje stosunkowo sporo wartości odstających. Widać również spore różnice w wielkościach atrybutów, co sugeruje nam potrzebę przeskalowania ich w późniejszym etapie zadania, podczas tworzenia modelu regresji.
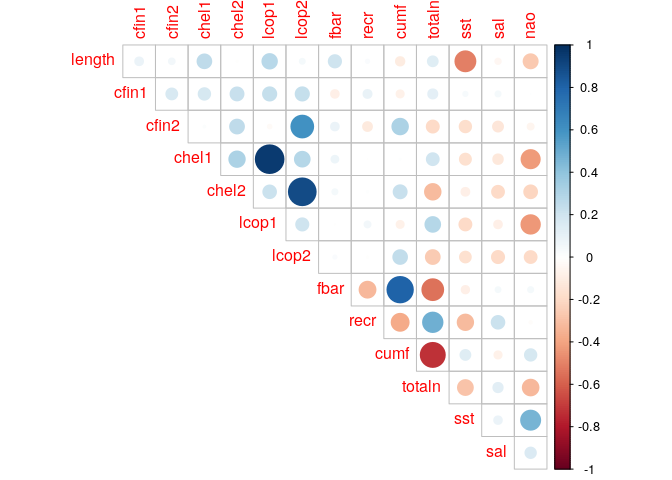
Wykresy korelacji atrybutów pokazuje kilka silnych zależności. Warto wyróżnić tutaj pary: chel1-lcop1, chel2-lcop2 oraz cumf-totaln. Pierwsze dwie pary to różne gatunki planktonu, występowanie jednego gatunku potencjalnie wpływa na rzadsze występowanie drugiego (korelacja bliska -1). Z kolei para cumf-totaln to natężenie połowów i łączna liczba złowionych ryb, ich pozytywna korelacja nie powinna zaskakiwać.
corrplot(
cor(df_grouped),
method="circle",
type="upper",
diag=FALSE
)
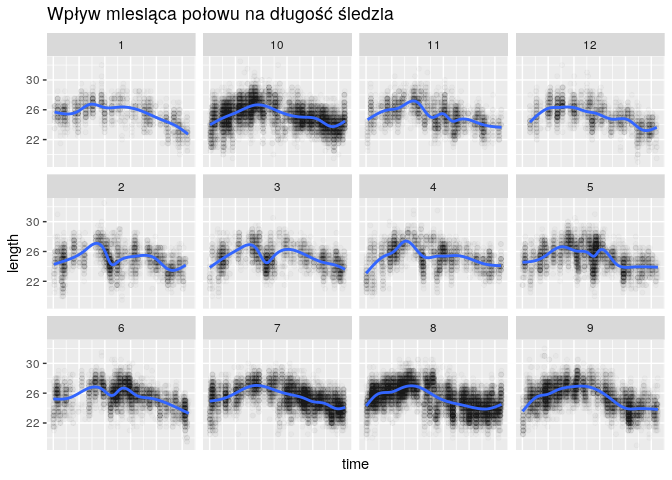
Postanowiliśmy również sprawdzić czy miesiąc znacząco wpływa na długość śledzi. Okazało się, że miesiąc nie ma zbyt dużego wpływu i można go pominąć w dalszej analizie. Dodatkowo, z uwagi na to, że miesiąc nie jest wartością liczbową, sprawiałby on problemy w niektórych modelach regresji.
ggplot(
df,
aes(x=time, y=length)
) +
facet_wrap(~xmonth) +
geom_point(alpha = 0.02) +
geom_smooth() +
labs(title = "Wpływ miesiąca połowu na długość śledzia") +
theme(
axis.text.x = element_blank(),
axis.ticks.x = element_blank()
)
Przed trenowanie modelu należy podzielić zbiór danych na zbiór treningowy i uczący. W naszym przypadku podzieliliśmy je w stosunku 1 do 3.
inTraining <- createDataPartition(
y = df_grouped$length,
p = .75,
list = FALSE
)
train_set <- df_grouped[inTraining,]
test_set <- df_grouped[-inTraining,]
dim(train_set)
## [1] 1920 14
dim(test_set)
## [1] 638 14
Do znalezienia regresora długości śledzi posługujemy się algorytmem regresji liniowej. Podczas uczenia używamy techniki walidacji krzyżowej z podziałem zbioru treningowego na 10 części.
fitControl <- trainControl(
method = "repeatedcv",
number = 10,
repeats = 10
)
model_cv <- train(
length ~ .,
data = as.matrix(train_set),
method = "lm",
na.action = na.omit,
trControl = fitControl,
preProcess = c("scale", "center")
)
model_cv
## Linear Regression
##
## 1920 samples
## 13 predictor
##
## Pre-processing: scaled (13), centered (13)
## Resampling: Cross-Validated (10 fold, repeated 10 times)
## Summary of sample sizes: 1728, 1728, 1729, 1727, 1729, 1727, ...
## Resampling results:
##
## RMSE Rsquared MAE
## 1.027292 0.4350031 0.7892772
##
## Tuning parameter 'intercept' was held constant at a value of TRUE
Za baseline uznaliśmy regresor, który zawsze zwraca średnią długość śledzia policzoną na wszystkich obserwacjach ze zbioru uczącego. Nauczony regresor jest znacząco lepszy niż baseline, co widać po wartościach RMSE. Niestesty nie udało nam się zrozumieć dlaczego Rsquared ma wartość NA w przypadku baseline’u.
test_pred <- predict(model_cv, test_set)
dim(test_set)
## [1] 638 14
postResample(pred = test_pred, obs = test_set$length)
## RMSE Rsquared MAE
## 0.9877959 0.4271115 0.7684870
postResample(pred = mean(train_set$length), obs = test_set$length)
## RMSE Rsquared MAE
## 1.302938 NA 1.040531
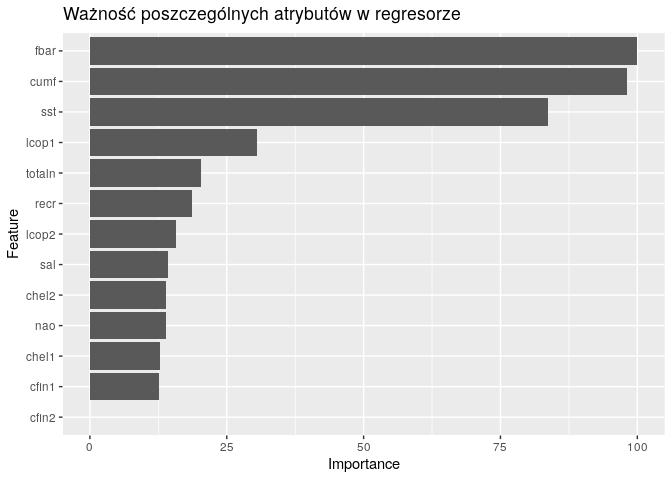
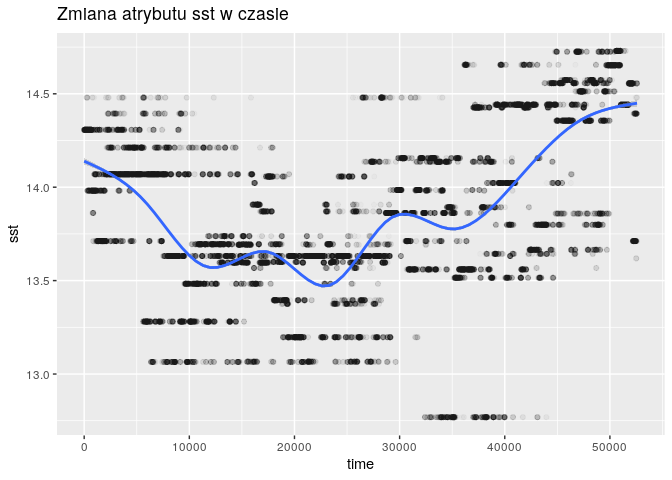
Spojrzenie na ważność cech w regresorze może pomóc nam w zrozumieniu występowania zjawiska stopniowego spadku rozmiaru śledzia. Dwa najbardziej znaczące atrybuty to natężenia połowów w regionie, wyrażone w ułamku pozostawionego narybku. Trzecim znaczącym atrybutem jest sst, czyli temperatura przy powierzchni wody.
ggplot(varImp(model_cv)) +
ggtitle("Ważność poszczególnych atrybutów w regresorze")
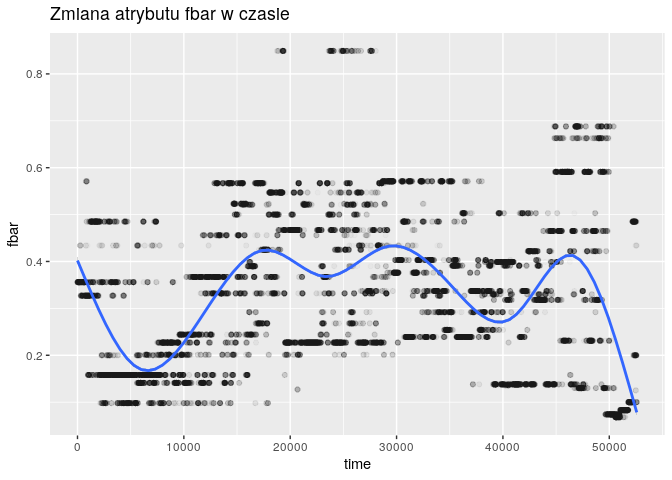
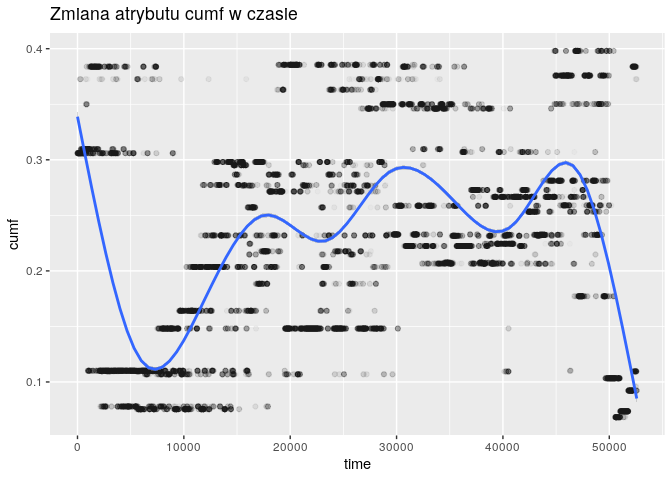
for (col in c("fbar", "cumf", "sst")) {
plt <- ggplot(
df,
aes_string(x="time", y=col)
) +
geom_point(alpha = 0.02) +
geom_smooth() +
ggtitle(paste("Zmiana atrybutu", col, "w czasie"))
print(plt)
}