打开 https://www.broadinstitute.org/medical-and-population-genetics/hapmap-3, 点击 How To Download This Release 下面的 A. SNP Genotype Data 段落的中间3个链接。 文件名字里面有 "b36",现在一般都用 b37(比如 UK Biobank),甚至有的用 b38, 所以下载后解压后需要将那个 .map 文件先用 liftOver 转化为 b37 格式,然后用 PLINK 生成 bed/bim/fam 文件。 这个基因数据可供 LDSC 和 GSMR 等软件使用。
在千人基因组官网 下载 Phase 3 对应的 VCF 链接, 有一个文件罗列了每一个样本的人群(pop)和人种 (super_pop),以及性别,可以用PLINK --keep 选取特定人种的样本。 下载下来的数据,有将近一个亿的SNP,每个染色体都是单独的文件。后续跑 GWAS 或提取 PRS 的时候,也是每条染色体的数据分开来跑。 PLINK的网站上也有“1000 genomes phase3” 数据。PLINK 不允许 SNP 名字有重复,可以用下面的命令来处理。
- awk '{if(array[$2]=="Y") {i++; $2=$2".DUP"i}; print $0; array[$2]="Y"}' chr1.bim.COPY > chr1.bim
从 ukbiobank 官网,点击 data showcase --> Essential information --> Accessing UK Biobank data,阅读 Data access guide 文件。
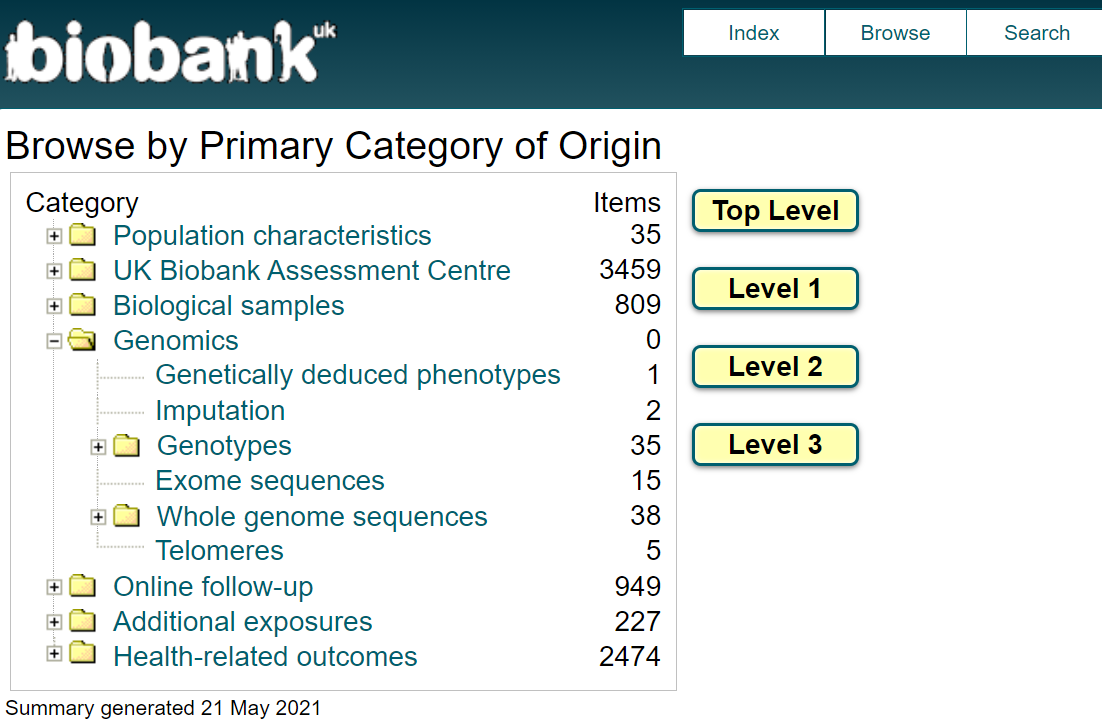
然后,可以通过 Search 或 Browse(截图如下)去熟悉 UKB 里面的数据结构。具体的数据,需要申请得到批准后,从最上面的 Researcher log in 登录后获取。
WINDOWS电脑建议安装系统自带的 Ubuntu Linux系统,然后用 cd /mnt/d/ (而不是 D:/)进入 D 盘。
-
先根据上述的Data access guide,执行 ukbunpack 解压表型数据的大文件。
-
写一个 VIP.fields.txt 文件,列出想提取的变量和对应的 data-field,比如
- 21022 age
-
然后手动或者用下面的命令,提取出该文件的第一列(需要提取的表型的ID),并确认没有重复的ID。
- awk '{print $1}' ukb.vip.fields > ukb.vip.fields.id
- sort ukb.vip.fields.id | uniq -d
-
用 ukbconv 提取上面的ID所对应的表型数据
- ukbconv ukb42156.enc_ukb r -iukb.vip.fields.id -ovip
-
用下面的R代码,通过上面生成的 vip.r 读入上面生成的 vip.tab 数据,并且给每个变量赋予上述 VIP.fields.txt给出的名字。需要注意 vip.r 第一行里面的路劲正确。
- source("D:/vip.r")
- pnames <- read.table("D:/ukb.vip.fields", header=F)
- pnames$V1 <- paste0("f.", pnames$V1, ".0.0")
- phe <- subset(bd, select=grep("f.eid|\.0\.0", names(bd)))
注:> 对于表型数据的提取,有一个 ukbtools R软件包。 但不是太好用,并且很慢。
ICD 这样的指标,包含了很多不同时间的时间点,量很大,建议分开来处理。
ukbconv ukb42156.enc_ukb r -s42170 -oicd sed -i 's/"//g icd.tab
将 icd.tab 文件整合为两列,便于读入R。
cat icd.tab | sed -e 's/\tNA//g' -e 's/\t/,/2g' |
awk '{ if(NR==1) print "IID icd"; else if (NF==1) print $1 " NA"; else print $0"," }' > icd.2cols
提取需要研究的表型数据和相关的covariates,比如 age, sex, PCs。 一般来说,quantitative的表型数据要 adjust for covariates 和转化成正态分布,这个可以在R里面用下面的命令来实现。
trait_res = residuals(lm(trait ~ age+sex+PC1+PC2, na.action=na.exclude) trait_inv = qnorm((rank(trait_res,na.last="keep")-0.5) / length(na.omit(trait_res)))
对于疾病的binary 表型,只需要把需要 adjust 的covarites 和表型数据放在同一个表型数据文件里面, 然后在 GWAS里面的plink命令指明哪个是表型,哪些是 covariates。
UKB的基因数据很大,所有申请者都能得到一样的数据(样本的ID不一样),一般下载到服务器上去储存和使用。
目前GWAS 由专人负责运行,一般来说就是通过下面这样的PLINK命令来跑
for chr in {1..22}; do
- plink2 --memory 12000 --threads 16 --pfile chr$chr --extract ukb.chr$chr.good.snps --pheno cvd.EUR.pheno --no-psam-pheno --pheno-name XXX --1 --glm cols=+ax,+a1freq,+a1freqcc,+a1count,+a1countcc,+beta,+orbeta,+nobs hide-covar no-x-sex --covar pheno/ukb.cov --covar-name age,sex,PC1-PC10 --out chr$chr
done
上述命令顺利跑完后,确认生成的文件没有问题后,可以把所有的染色体的数据串到一起,形成一个单一的 XXX.gwas.gz 文件。鉴于2千多万个SNP,文件太大,我们一般只保留:P<0.01的SNP 以及那些在Hapmap3 里面的SNP。最终合并成的 XXX.gwas.gz 文件用 TAB 分割,CHR:POS 排好序,要不然 LocusZoom 那样的软件不能处理。也可以用 tabix -f -S 1 -s 1 -b 2 -e 2 XXX.gwas.gz 对数据进行索引,便于 LocalZoom 那样的软件去处理。
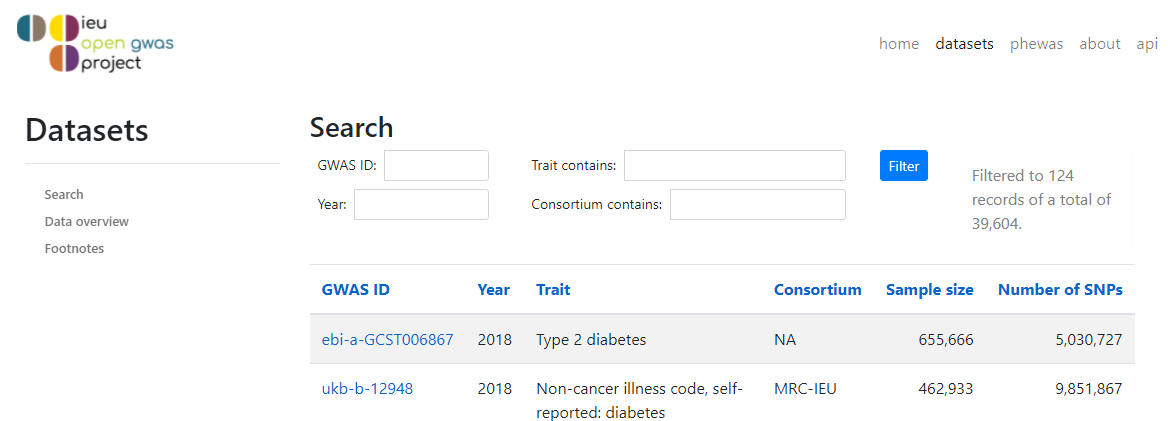
最经典的,起源于美国NIH 的 GWAS Catalog. 这个页面也罗列了一些大型GWAS数据联盟。
UKB GWAS 完整的分析结果,网上发布
- 美国哈佛大学:http://www.nealelab.is/uk-biobank
- 英国爱丁堡大学:geneatlas: http://geneatlas.roslin.ed.ac.uk
各大疾病联盟
- 哈佛大学的CVD knowlege portal: https://hugeamp.org/
- 南加州大学的神经影像基因组国际合作团队:http://enigma.ini.usc.edu/
如果不用上述的系统,也可以用 PLINK 人工操作。点击左边菜单中的 Report postprocess 中的 3个命令(--annotate, --clump, --gene-report)
trait=MI
gunzip -c $trait.gwas.gz | sed '1 s/ POS/ BP/' > $trait.gwas.txt # 以后就不需要 sed 这一步了
plink --annotate $trait.gwas.txt NA ranges=glist-hg19 --border 10 --pfilter 5e-8 --out $trait.top
# 由于千人基因组 (g1k) 的基因数据过大(将近1亿个SNP),一般讲每一个染色体的GWAS数据分开来 clump
# plink clump 的结果,不包括那些 --bfile 里面没有的SNP,所以得要把那些SNP再添加到 clump 的结果里。
# 已沟通,可惜 PLINK的作者不想让 PLINK 来直接处理这个问题,
for chr in {1..22}; do
plink1.9 --vcf g1k.chr$chr.vcf.gz --clump $trait.gwas.txt --clump-p1 5e-08 --clump-p2 5e-08 --clump-kb 1000 --clump-r2 0.2 --out $trait.chr$chr
awk '$1 !="" {print $3,$1, $4,$5}' $trait.chr$chr.clumped > $trait.chr$chr.top
done
# 通过LD的计算来找到GWAS数据里面的independent top hits,也有一些问题(比如g1k的LD不是金标准,r2也不是最合理的筛选办法),并且计算量很大。
# 如果不考虑 SNP之间的LD,只考虑距离,可以用下面这个简单的代码来寻找GWAS数据里面每1MB区间的top SNP。
# 假设GWAS的第1,2,3 列分别是 SNP, CHR, POS,最后一列是P。
zcat ABC.gwas.gz | awk 'NR==1 || $NF<5e-8 {b=sprintf("%.0f",$3/1e6); print $1,$2,$3,$NF,b}' | \
sort -k 2,2n -k 5,5n -k 4,4g | awk '{if (arr[$NF] !="Y") print $0; arr[$NF] ="Y"}'
# 要把上述得到的显著区域跟别人已经发表的 SNP进行比较,看是不是有重叠(1MB范围之内的重叠都算),可以用 bedtools 命令。
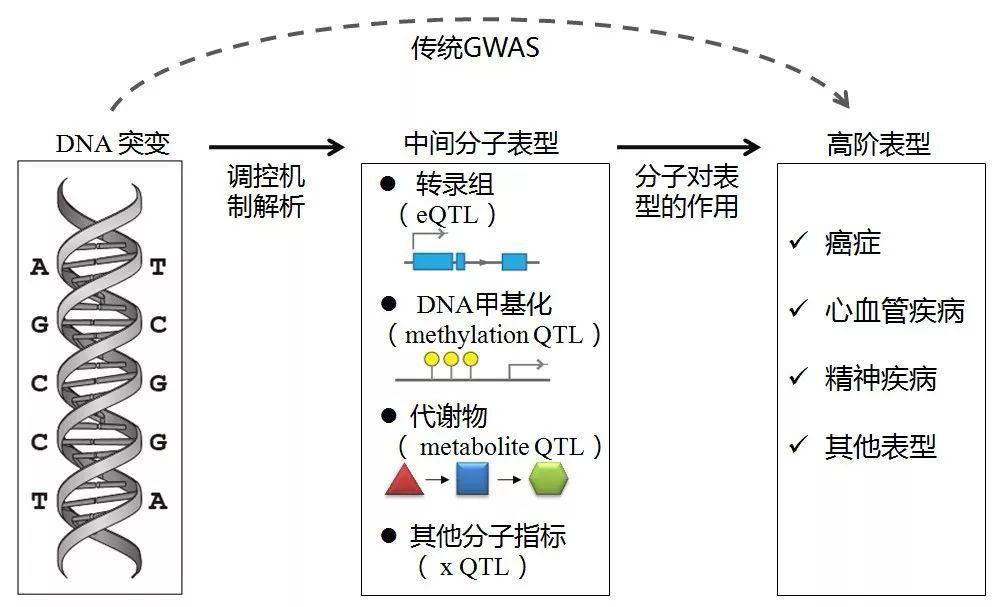
可阅读2020年的文章From GWAS to Function: Using Functional Genomics to Identify the Mechanisms Underlying Complex Diseases
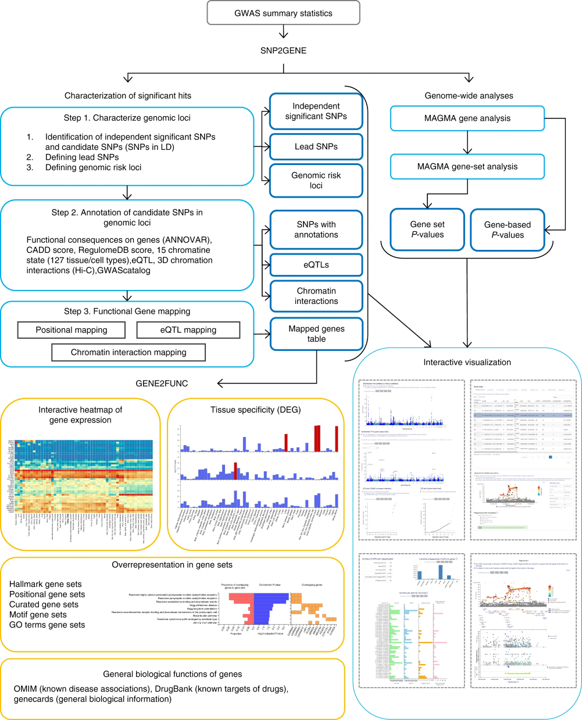
可先尝试傻瓜相机式的FUMA 网上解读系统
如果有简单的数据,别人文章里面已经报道了的 exposure 和 outcome 的 BETA 和 SE,最简单的是使用 MendelianRandomization R包。还有一个特别针对 UKB 处理海量数据的 TwoSampleMR R包。 MendelianRandomizaiton R包简单透明。只需要先把两个表型的summary数据运行一下 compare-B, 然后得到一个只有4列的小文件 (BETA1, SE1, BETA2, SE2)。TwoSampleMR 自然是不错,连数据都不需要了,只需要写一个 MRC-IEU的代码,就可以运行远程的数据。但是试想一下,哪天上不了那个网,或者对方将数据大量更新修改,我们的结果就再也重复不了了。建议两种方法都用,double check!
基因注释信息浏览器:
- dbSNP: https://www.ncbi.nlm.nih.gov/snp/
- UCSC genome browser: https://www.genome.ucsc.edu/
- 美国精准医学:https://databrowser.researchallofus.org/
- TopMed browser: https://bravo.sph.umich.edu/
- Gnomad browser: https://gnomad.broadinstitute.org/
- GlobalBiobankEngine:https://github.com/rivas-lab
GWAS 入门:
- 芬兰赫尔辛基大学 GWAS 课程:https://www.mv.helsinki.fi/home/mjxpirin/GWAS_course/
- Y 2020. NEJM. Genomewide Association Study of Severe Covid-19 with Respiratory Failure (https://www.nejm.org/doi/full/10.1056/NEJMoa2020283)
- Y 2021. Nature Reviews Methods Primers. Genome-wide association studies
多基因效应:Polygeny 以及 PRS
- Y 2019. Lancet Respiratory Medicine. Identification of risk loci and a polygenic risk score for lung cancer: a large-scale prospective cohort study in Chinese populations
- Y 2022. EHJ. A polygenic risk score improves risk stratification of coronary artery disease: a large-scale prospective Chinese cohort study
基因多效性:Pleiotropy (分为横向和纵向),其中纵向 Pleiotropy 是孟德尔随机化方法的基本要素。
- Y 2012. Lancet. Plasma HDL cholesterol and risk of myocardial infarction: a mendelian randomisation study
- Y 2017. Statistical methods to detect pleiotropy in human complex traits
- Y 2019. Meta-analysis and Mendelian randomization: A review
- Y 2019. Nature Genetics. A global overview of pleiotropy and genetic architecture in complex traits
- Y 2021. Genetic correlation and causal relationships between cardio-metabolic traits and lung function impairment
- Y 2022. Assessing the Causal Role of Sleep Traits on Glycated Hemoglobin: A Mendelian Randomization Study
- Y 2022. Genetically predicted sex hormone levels and health outcomes: phenome-wide Mendelian randomization investigation
R入门: