- Seleção de variáveis
- PCA (redução de dimensionalidade)
- Classificação com os modelos: Regressão Logistica, NB e GBM
- Realizar teste estatístico (Bootstrap)
- operações de resgate do trabalho escravo no Brasil de 2003 a 2018
- Inicializa pacotes e bibliotecas
- Carrega dados
- Padroniza variáveis independentes
- Seleção de variáveis por regularização - LASSO (L1)
- Principal Component Analysis PCA
- Modelagem com classificadores: Regressão Logística, Gaussian Naive Bayes (NB) e Gradient Boosting Machine (GBM)
- Teste estatístico com Bootstrapping
- Considerações finais
- Funções utilizadas
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
from sklearn import datasets
from sklearn.decomposition import PCA
from sklearn.linear_model import SGDClassifier
from sklearn.pipeline import Pipeline
from sklearn.model_selection import GridSearchCV
from multiprocessing import Pool, Process, Queue, Pipe, Lock, Value, Array, Manager, TimeoutError
import os
import pandas as pd
import numpy as np
import time
import seaborn as sns
from matplotlib import pyplot
from sklearn.preprocessing import MinMaxScaler
from sklearn.linear_model import LassoCV
from sklearn.naive_bayes import GaussianNB
from sklearn.svm import SVR
from sklearn.ensemble import GradientBoostingClassifier
from sklearn.neural_network import MLPClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from imblearn.metrics import geometric_mean_score
from sklearn.model_selection import train_test_split
from sklearn import model_selection
from sklearn import metrics
from sklearn.metrics import confusion_matrix,f1_score, precision_score, auc,roc_auc_score,roc_curve, precision_recall_curve, recall_score,accuracy_score
import matplotlib.pyplot as plt
from sklearn.feature_selection import SelectFromModel
from sklearn.model_selection import train_test_split
from sklearn.model_selection import GridSearchCV
from imblearn.over_sampling import RandomOverSampler
import matplotlib.pyplot as plt
import warnings
warnings.simplefilter(action='ignore')
from sklearn.exceptions import DataConversionWarning
warnings.filterwarnings(action='ignore', category=DataConversionWarning)
df = pd.read_csv('../data/merge.csv',low_memory=False)
# Retira variáveis não necessárias
df=df.drop(['trabalhadores_resgatados','municipio_key','nm_uf','id'], axis=1)
X = df.iloc[:,5:237]
y = df.escravo
XY = df.iloc[:,5:238]
scaler = MinMaxScaler()
X[X.columns] = scaler.fit_transform(X[X.columns])
#XY[X.columns] = scaler.fit_transform(XY[X.columns])XY.head(2).dataframe tbody tr th {
vertical-align: top;
}
.dataframe thead th {
text-align: right;
}
| espvida | fectot | mort1 | mort5 | razdep | sobre40 | sobre60 | t_env | e_anosestudo | t_analf11a14 | ... | pia18m | pop | popt | i_escolaridade | i_freq_prop | idhm | idhm_e | idhm_l | idhm_r | escravo | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 70.75 | 2.24 | 23.8 | 25.49 | 47.37 | 94.50 | 83.18 | 5.84 | 8.67 | 1.22 | ... | 16431.0 | 24097 | 22429 | 0.368 | 0.629 | 0.641 | 0.526 | 0.763 | 0.657 | 0 |
| 1 | 73.36 | 1.91 | 19.2 | 20.53 | 46.68 | 96.12 | 87.69 | 4.36 | 9.18 | 1.55 | ... | 60213.0 | 89766 | 88730 | 0.514 | 0.648 | 0.702 | 0.600 | 0.806 | 0.716 | 1 |
2 rows × 233 columns
A ideia foi utilizar uma técnica para seleção de variáveis, resultando em um subconjunto de variáveis com maior grau de importância para o modelo.
Devido ao alto grau de dimensionalidade, com 232 variáveis + 1 alvo (escravo), esta etapa é primordial.
def lasso_(X,y):
num_features = len(X.columns)
clf = LogisticRegression(C=0.1,penalty = 'l1')
clf.fit(X,y)
zero_feat = []
nonzero_feat = []
abs_feat = []
for i in range(num_features):
coef = clf.coef_[0,i]
if coef == 0:
zero_feat.append(X.columns[i])
else:
nonzero_feat.append((coef, X.columns[i]))
print ('Variaveis com coeficiente zero: ', len(zero_feat))
print ('Variaveis selecionadas:')
print (sorted(nonzero_feat, reverse = True))
# Chamada na função lasso_() para realizar a Seleção de variáveis.
lasso_(X,y)Variaveis com coeficiente zero: 216
Variaveis selecionadas:
[(1.530182571058944, 'prentrab'), (0.6342283001411675, 'parede'), (0.5877743502165127, 't_atraso_1_basico'), (0.40413755821255565, 't_agua'), (0.3876778951603006, 'pren10ricos'), (0.3572131187201117, 't_super25m'), (0.27981627466657805, 'trabsc'), (-0.14766392349401283, 't_freq4a5'), (-0.367663908053415, 'pren40'), (-0.4712133234766228, 'ren2'), (-0.5661567245409527, 'rind'), (-0.9408619886536362, 't_flpre'), (-1.0173559356708657, 'pren20'), (-1.1936811143772472, 'p_transf'), (-1.4450673364882742, 'ren1'), (-3.8072514873057925, 't_env')]
selecionados =['prentrab', 'parede', 't_atraso_1_basico', 't_agua', 'pren10ricos', 't_super25m',
'trabsc', 't_freq4a5', 'ren2', 'pren40','rind', 'pren20','t_flpre','p_transf', 'ren1','t_env']# Variável a ser prevista
atrib_prev = ['escravo']
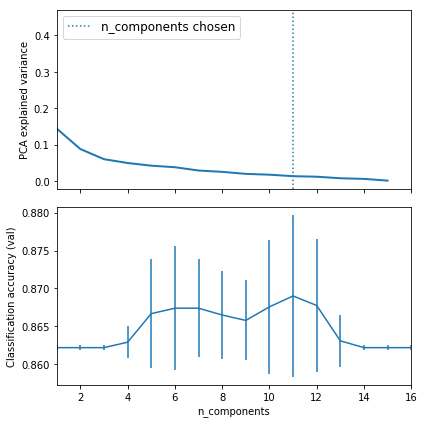
X_s=X[selecionados]Com o subgrupo de variáveis, a ideia foi explorar a linearidade entre variáveis, bem como verificar a melhor quantidade de componente principais para o subgrupo selecionado. Para isso foi utilizado uma pipeline de busca por melhores parametros, onde então é realizado combinações.
Os resultados indicam que o melhor número de componentes são: 11 (para o sub grupo de dados utilizados)
# Pipeline para busca de parâmetros no PCA
logistic = SGDClassifier(loss='log', penalty='l2', early_stopping=True,
max_iter=10000, tol=1e-5, random_state=0)
pca = PCA()
pipe = Pipeline(steps=[('pca', pca), ('logistic', logistic)])
# Configuracao do grid
param_grid = {
'pca__n_components': [1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16],
'logistic__alpha': np.logspace(-4, 4, 5),
}
search = GridSearchCV(pipe, param_grid, iid=False, cv=5)
search.fit(X_s, y)
print("Best parameter (CV score=%0.3f):" % search.best_score_)
print(search.best_params_)
# Plot the PCA spectrum
pca.fit(X_s)
fig, (ax0, ax1) = plt.subplots(nrows=2, sharex=True, figsize=(6, 6))
ax0.plot(pca.explained_variance_ratio_, linewidth=2)
ax0.set_ylabel('PCA explained variance')
ax0.axvline(search.best_estimator_.named_steps['pca'].n_components,
linestyle=':', label='n_components chosen')
ax0.legend(prop=dict(size=12))
# For each number of components, find the best classifier results
results = pd.DataFrame(search.cv_results_)
components_col = 'param_pca__n_components'
best_clfs = results.groupby(components_col).apply(
lambda g: g.nlargest(1, 'mean_test_score'))
best_clfs.plot(x=components_col, y='mean_test_score', yerr='std_test_score',
legend=False, ax=ax1)
ax1.set_ylabel('Classification accuracy (val)')
ax1.set_xlabel('n_components')
plt.tight_layout()
plt.show()Best parameter (CV score=0.869):
{'logistic__alpha': 0.0001, 'pca__n_components': 11}
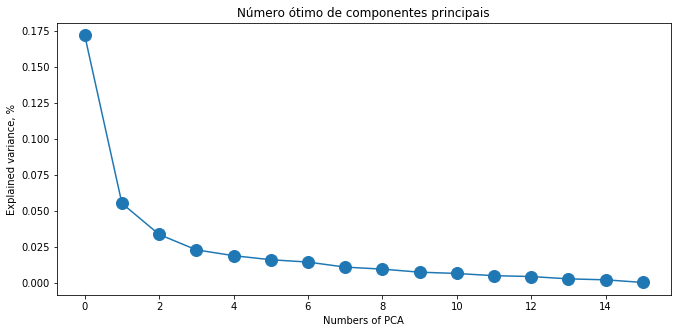
plt.figure(figsize=(11, 5))
plt.plot(pca.explained_variance_, 'o-', markersize=12)
plt.xlabel('Numbers of PCA')
plt.ylabel('Explained variance, %')
plt.title("Número ótimo de componentes principais")
plt.show() pca = PCA(n_components=11)
principalComponents = pca.fit_transform(X_digits)
principalDf = pd.DataFrame(data = principalComponents
, columns = ['pc1', 'pc2','pc3', 'pc4','pc5', 'pc6','pc7','pc8', 'pc9','pc10', 'pc11',])É possível pegar uma amostra dos valores dos componentes:
finalDf = pd.concat([principalDf, XY[['escravo']]], axis = 1)
finalDf.head().dataframe tbody tr th {
vertical-align: top;
}
.dataframe thead th {
text-align: right;
}
| pc1 | pc2 | pc3 | pc4 | pc5 | pc6 | pc7 | pc8 | pc9 | pc10 | pc11 | escravo | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.108799 | 0.632460 | -0.100407 | -0.059520 | -0.061458 | -0.158455 | 0.069243 | -0.048837 | -0.079518 | -0.110502 | -0.006094 | 0 |
| 1 | -0.283818 | 0.497275 | -0.166798 | 0.115194 | -0.019565 | 0.069817 | 0.078578 | -0.040387 | -0.072134 | -0.021477 | -0.006238 | 1 |
| 2 | -0.062615 | 0.149691 | -0.042640 | 0.011175 | 0.005149 | -0.033607 | 0.000416 | -0.053863 | -0.162688 | -0.082522 | -0.058702 | 0 |
| 3 | -0.245588 | 0.370297 | -0.244629 | -0.042920 | 0.021341 | -0.058265 | 0.019964 | -0.001229 | -0.038496 | -0.091237 | -0.018047 | 0 |
| 4 | -0.146514 | 0.240342 | -0.008499 | 0.020280 | 0.081181 | -0.051858 | -0.100560 | -0.009490 | -0.080348 | -0.060474 | -0.035422 | 1 |
Para melhor visualização:
- 0 - indica ausência do trabalho escravo
- 1 - indica presença do trablaho escravo
target_names = [0,1]
pca = PCA(n_components=11)
X_r = pca.fit(X_s).transform(X_s)
# Porcentagem de variância explicada para cada componente
print('Razão de variância explicada: %s'
% str(pca.explained_variance_ratio_))
plt.figure()
colors = ['navy', 'turquoise']
lw = 2
for color, i, target_name in zip(colors, [0,1], target_names):
plt.scatter(X_r[y == i, 0], X_r[y == i, 1], color=color, alpha=.8, lw=lw,
label=target_name)
plt.legend(loc='best', shadow=False, scatterpoints=1)
plt.title('PCA do Trabalho Escravo')
plt.show()Razão de variância explicada: [0.44789656 0.14410424 0.08790111 0.05999895 0.04951029 0.04214407
0.03791371 0.02882399 0.02516627 0.01965494 0.01733414]
6. Modelagem com classificadores: Regressão Logística, Gaussian Naive Bayes (NB) e Gradient Boosting Machine (GBM)
- 70% treinamento
- 30% treinamento
# Separa os dados
split_test_size =0.3
datasets3 = []
X_imb3, Y_imb3 = split_s_colunas(df,selecionados,atrib_prev) #usado para gerar testes com base original
X_treino_imb3, X_teste_imb3, Y_treino_imb3, Y_teste_imb3 = split_data(X_imb3, Y_imb3,split_test_size)- A técnica utilizada para balancemento de dados foi RandomOverSampling (ROS) Foi feito um benchmarking comparando com outras técnicas, ROS obteve melhor desempenho nos testes.
# chama função balance_datasets (disponível ao final do documento)
datasets3 = balance_datasets(X_treino_imb3,Y_treino_imb3)Transforming RandomOverSampler
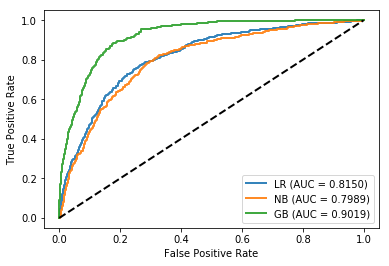
O algoritmo Gradient Boosting Machine, destacou-se com melhor desempenho nos treinamentos. Se comparado com Gaussian Naive Bayes e Regressão Logística.
# chama função analisa_modelo_train (disponível ao final do documento)
# a função passa um array de dados, e também os conjuntos de dados separados de variáveis independentes e variável dependente.
# Retorna: a avaliação para os modelos e respectiva curva roc, provida pelos valores de AUC.
analisa_modelo_train(datasets3, X_treino_imb3, Y_treino_imb3)Avaliação de treino
nome Sampling Type accuracy auc precision recall \
0 LR RandomOverSampler 0.748940 0.814979 0.747897 0.750880
1 NB RandomOverSampler 0.740402 0.798943 0.731932 0.758679
2 GB RandomOverSampler 0.832280 0.901938 0.809240 0.870795
mean NaN NaN 0.773874 0.838620 0.763023 0.793452
f1 g_mean
0 0.749267 0.748948
1 0.744972 0.740182
2 0.838894 0.831706
mean 0.777711 0.773612
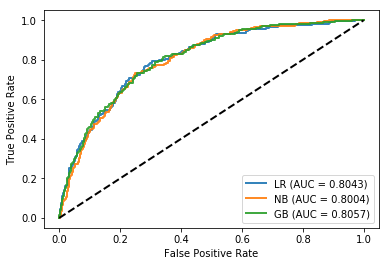
Os resultados indicam aproximidade entre os modelos, com uma pequena diferença para o modelo com uso de Gradient Boost. Também é possível observar que a curva ROC apresenta um pouco mais de Falsos positivos se comparado ao treinamento.
# Chamada da função que faz a modelagem em dados de teste
analisa_modelo_test(datasets3,X_teste_imb3,Y_teste_imb3)Avaliação de teste
nome Sampling Type accuracy auc precision recall \
0 LR RandomOverSampler 0.737126 0.804341 0.282486 0.721154
1 NB RandomOverSampler 0.732335 0.800359 0.281536 0.740385
2 GB RandomOverSampler 0.774251 0.805706 0.306636 0.644231
mean NaN NaN 0.747904 0.803469 0.290219 0.701923
f1 g_mean
0 0.405954 0.730219
1 0.407947 0.735773
2 0.415504 0.714642
mean 0.409802 0.726878
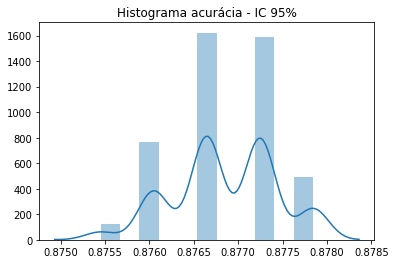
Com aplicação do teste estatístico com 150 interações é possível identificar que o modelo apresenta Robustez. Com bons resultados, o teste demonstra ótimo desempenho e dentro do intervalo de confiança de 95%.
# Bootstrapping
values = df.values
n_iter = 150
resultados = list()
start_time = time.time()
#tamanho a ser usado no treinamento
for i in range(n_iter):
# divisao de dados em treino e teste
X_treino_imb3, X_teste_imb3, Y_treino_imb3, Y_teste_imb3 = split_data(X_imb3, Y_imb3,split_test_size)
# fit model (o melhor modelo apresentado nos testes foi GB. Deste modo escolhido para fazer o teste de Robustez)
mod = GradientBoostingClassifier()
ml = mod.fit(X_treino_imb3, Y_treino_imb3)
model_pd = ml.predict(X_teste_imb3)
acc_=metrics.accuracy_score(Y_teste_imb3, model_pd)
resultados.append(acc_)
# Chama funcao com o Intervalo de confianca (IC)
intervalo(resultados)
print("\nO tempo de execução foi de %.2f minutos." % ((time.time() - start_time) / 60))Para o intervalo de confiança de 95.0%, os resultados estão entre 87.6% e 87.8%
O tempo de execução foi de 1.37 minutos.
# Imprimi histograma
fig=sns.distplot(resultados)
pyplot.title("Histograma acurácia - IC 95%")
pyplot.show(fig)Foi possível aplicar os conceitos aprendidos em sala de aula, principalmente referente a seleção de variáveis e testes com técnicas de robustez, as técnicas demonstram ser eficazes e de importante valor.
Para trabalhos futuros, pretendo utilizar um modelo multiclasse, logo implementarei uma abordagem com Naive Bayes para três categorias de classes.
# Função para o calculo do IC, usada no conjunto de treinamento
def intervalo(resultado):
alpha = 0.95
p = ((1.0-alpha)/2.0) * 100
lower = max(0.0, np.percentile(resultado, p))
p = (alpha+((1.0-alpha)/2.0)) * 100
upper = min(1.0, np.percentile(resultado, p))
print('Para o intervalo de confiança de %.1f%%, os resultados estão entre %.1f%% e %.1f%%' % (alpha*100, lower*100, upper*100))
# Pega o nome das colunas, para as variáveis selecionadas
def check_features(estimator,cols):
temp = pd.Series(estimator.support_,index = cols)
selected_features_rfe = temp[temp==True].index
print(selected_features_rfe)
# Ranking das n melhores variáveis
def best_worst (scores,x):
scores = sorted(scores, reverse = True)
print("As "+ str(x) +" melhores variaveis selecionadas :")
var=[]
for i in range(x):
print("%d° - " % i+1, scores[i][1])
var.append(scores[i][1])
print(var)
#numero de features
def n_features_score(X,y):
nof_list=np.arange(1,237)
high_score=0
#Variable to store the optimum features
nof=0
score_list =[]
for n in range(len(nof_list)):
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size = 0.3, random_state = 0)
model = LogisticRegression()
rfe = RFE(model,nof_list[n])
X_train_rfe = rfe.fit_transform(X_train,y_train)
X_test_rfe = rfe.transform(X_test)
model.fit(X_train_rfe,y_train)
score = model.score(X_test_rfe,y_test)
score_list.append(score)
if(score>high_score):
high_score = score
nof = nof_list[n]
print("Optimum number of features: %d" %nof)
print("Score with %d features: %f" % (nof, high_score))
def l2_regularization(X,y):
num_features = len(X.columns)
param_grid = {'logisticregression__C': [0.001, 0.01, 0.1, 1, 10, 100]}
pipe = make_pipeline(StandardScaler(), LogisticRegression(penalty = 'l2'))
grid = GridSearchCV(pipe, param_grid, cv = 10)
grid.fit(X, y)
print(grid.best_params_)
X_scaled = StandardScaler().fit_transform(X)
clf = LogisticRegression(penalty = 'l2', C = 1)
clf.fit(X_scaled,y)
abs_feat = []
for i in range(num_features):
coef = clf.coef_[0,i]
abs_feat.append((abs(coef), X.columns[i]))
print (sorted(abs_feat, reverse = True))
print_best_worst(abs_feat)
def split_data(X_split, y_split,split_test_size):
return train_test_split(X_split, y_split, test_size = split_test_size, random_state = 1)
def split_s_colunas(sample,atributos_selecionados,atrib_prev):
X = sample[atributos_selecionados].values
Y = sample[atrib_prev].values
return X,Y
def balance_datasets(X_treino_imb,Y_treino_imb):
datasets = []
datasets.append(transform(RandomOverSampler(random_state=10),X_treino_imb,Y_treino_imb))
return datasets
def transform(transformer,X,y):
print("Transforming {}".format(transformer.__class__.__name__))
X_resampled,y_resampled = transformer.fit_sample(X,y)
return transformer.__class__.__name__,pd.DataFrame(X_resampled), pd.DataFrame(y_resampled)
def inicializaModelos():
modelos = []
modelos.append(('LR', LogisticRegression()))#C=10
modelos.append(('NB', GaussianNB()))
modelos.append(('GB', GradientBoostingClassifier()))#n_estimators = 100
return modelos
def analisa_modelo_test(datasets,X_teste,Y_teste):
# Definindo os valores para o número de folds
num_folds = 10
seed = 59
# Preparando os modelo
modelos = inicializaModelos()
nomes = []
scores_test=[]
aucs=[]
for s_type,x_train,y_train in datasets:
for nome, modelo in modelos:
mod = modelo
modelo_fit = mod.fit(x_train, y_train)
modelo_pred = modelo_fit.predict(X_teste)
modelo_proba = modelo_fit.predict_proba(X_teste)
fpr, tpr, t = roc_curve(Y_teste, modelo_proba[:, 1])
roc_auc = auc(fpr, tpr)
g_mean=geometric_mean_score(Y_teste,modelo_pred)
acc_teste=metrics.accuracy_score(Y_teste, modelo_pred)
roc_teste=metrics.roc_auc_score(Y_teste, modelo_proba[:, 1])
precision=metrics.precision_score(Y_teste, modelo_pred)
recall=metrics.recall_score(Y_teste,modelo_pred)
f1=metrics.f1_score(Y_teste,modelo_pred)
# Armazena o modelo escolhido
if (s_type=='RandomOverSampler'):
if(nome=='GB'):
global melhor_modelo
melhor_modelo = modelo_fit
scores_test.append((nome,s_type,
acc_teste,
roc_auc,
precision,
recall,
f1,
g_mean
))
aucs.append(roc_auc)
plt.plot(fpr, tpr, lw=2, alpha=0.9, label='%s (AUC = %0.4f)' % (nome, roc_auc))
print("Avaliação de teste")
sampling_results_test = pd.DataFrame(scores_test,columns=['nome','Sampling Type','accuracy','auc','precision','recall','f1','g_mean'])
sampling_results_test.loc['mean'] = sampling_results_test.mean()
print(sampling_results_test)
print()
plt.plot([0,1],[0,1],linestyle = '--',lw = 2,color = 'black')
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.legend(loc="lower right")
plt.show()
scores_test=[]
def analisa_modelo_train(datasets,X_treino,Y_treino):
# Definindo os valores para o número de folds
num_folds = 10
seed = 30
# Preparando os modelo
modelos = inicializaModelos()
# Avaliando cada modelo
scores = []
aucs=[]
for s_type,x_train,y_train in datasets:
for nome, modelo in modelos:
kfold = model_selection.StratifiedKFold(n_splits = num_folds, random_state = seed, shuffle=True)
cv_results_ac = model_selection.cross_val_score(modelo, x_train, y_train, cv = kfold, scoring ='accuracy',n_jobs=-1)
cv_results_auc = model_selection.cross_val_score(modelo, x_train, y_train, cv = kfold, scoring ='roc_auc',n_jobs=-1)
cv_results_loss = model_selection.cross_val_score(modelo, x_train, y_train, cv = kfold, scoring ='neg_log_loss',n_jobs=-1)
cv_results_prec = model_selection.cross_val_score(modelo, x_train, y_train, cv = kfold, scoring = 'precision',n_jobs=-1)
cv_results_recall = model_selection.cross_val_score(modelo, x_train, y_train, cv = kfold, scoring ='recall',n_jobs=-1)
cv_results_f1 = model_selection.cross_val_score(modelo, x_train, y_train, cv = kfold, scoring = 'f1',n_jobs=-1)
cv_results_y_pred = model_selection.cross_val_predict(modelo,x_train,y_train,cv= kfold,n_jobs=-1)
g_mean_cv=geometric_mean_score(y_train,cv_results_y_pred)
#para plot
modelo_fit = modelo.fit(x_train, y_train)
modelo_pred = modelo_fit.predict(X_treino)
modelo_proba = modelo.predict_proba(X_treino)
fpr, tpr, t = roc_curve(Y_treino, modelo_proba[:, 1])
roc_auc = auc(fpr, tpr)
scores.append((nome,s_type,
cv_results_ac.mean(),
cv_results_auc.mean(),
cv_results_prec.mean(),
cv_results_recall.mean(),
cv_results_f1.mean(),
g_mean_cv
))
aucs.append(roc_auc)
plt.plot(fpr, tpr, lw=2, alpha=0.9, label='%s (AUC = %0.4f)' % (nome, cv_results_auc.mean()))
print("Avaliação de treino")
sampling_results = pd.DataFrame(scores,columns=['nome','Sampling Type','accuracy','auc','precision','recall','f1','g_mean'])
sampling_results.loc['mean'] = sampling_results.mean()
print(sampling_results)
plt.plot([0,1],[0,1],linestyle = '--',lw = 2,color = 'black')
plt.xlabel('False Positive Rate')
plt.ylabel('True Positive Rate')
plt.legend(loc="lower right")
plt.show()
scores=[]