[TOC]
整理了几种常见的将DNA表示为数学特征的方法,以及该方法的论文出处(在目录下paper文件夹里)。
在生物信息学中,k-mers是包含在生物序列中的长度为k的子串。主要用于计算基因组学和序列分析,其中k-mers由核苷酸(即A、T、G和C)组成,术语k-mer是指长度为{ k}的序列的所有子序列,因此序列AGAT将具有四个单体(a、G、a和T)、三个2-mer(AG、GA、AT)、两个3-mer(AGA和GAT)和一个4-mer(AGAT)。更一般地,长度为{ L}的序列将具有{ L-k+1}k-mers和{n ^{k}总的可能k-mers,其中{n}是可能的单体的数量(例如,在DNA的情况下为四个)。
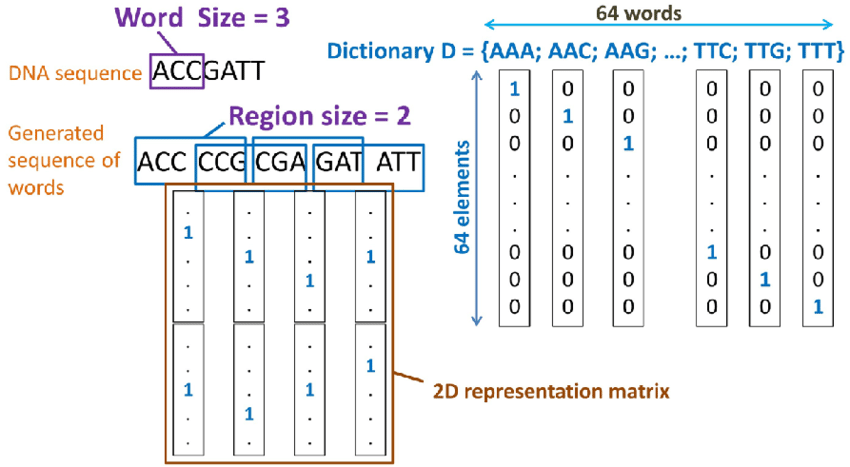
我们可以将DNA序列按k取k-mer,(k常取3,4,5,6,7),接着计算一条DNA中每一种k-mer对应的频率,这样就得到了一条序列的向量表示。
DNA2VEC/kmer_to_freqs/cal.py可以使用reshape的方法将二维k-mer频率特征转化为4维,接着送入卷积网络提取特征以及实现下游任务。
DNA2VEC/kmer_freqs+CNN/DeepTE/DeepTE.pyhot-hot编码,独热编码,又称一位有效编码,其方法是使用N位状态寄存器来对N个状态进行编码,每个状态都有它独立的寄存器位,并且在任意时候,其中只有一位有效。
例如:
自然状态码为:000,001,010,011,100,101
独热编码为:000001,000010,000100,001000,010000,100000
可以这样理解,对于每一个特征,如果它有m个可能值,那么经过独热编码后,就变成了m个二元特征。并且,这些特征互斥,每次只有一个激活。因此,数据会变成稀疏的。
这样做的好处主要有:
- 解决了分类器不好处理属性数据的问题
- 在一定程度上也起到了扩充特征的作用
下图为DNA序列的one-hot编码
该程序使用one-hot+LSTM网络的方法实现DNA序列的分类
DNA2VEC/one-hot+LSTM/DeepGenGrep.pyWord2vec是Word Embedding 的方法之一,是2013 年由谷歌的 Mikolov提出了一套新的词嵌入方法。在word embedding之前出现的文本表示方法有one-hot编码和整数编码,one-hot编码和整数编码的缺点均在于无法表达词语之间的相似性关系。如何解决这个问题呢?自动学习向量之间的相似性表示,用更低维度的向量来表示每一个单词。
将DNA序列数据集以k-mer法或整切法分割成一个word,接着使用word2vec训练词库,便得到了该数据集的词库。该词库可以在NLP网络中使用Eembedding方法嵌入,后序可使用常见NLP网络例如LSTM/TEXTCNN/Transformer等。
word2vec+CNN/word2vec.ipynb破译非编码DNA的语言是基因组研究的基本问题之一。由于存在多义性和远距离语义关系,基因调控码是高度复杂的,而以前的信息学方法往往无法捕捉到这一点,尤其是在数据稀缺的情况下。
DNABERT是一种预训练的双向编码表示法,以基于上游和下游核苷酸上下文捕获基因组DNA序列的全局和可转移理解。DNABERT与最广泛使用的全基因组调控元件预测程序进行了比较,并证明了其易用性、准确性和效率。
该方法证明了带有人类基因组的预先训练的DNABERT甚至可以很容易地应用于其他生物。预训练的DNABERT模型可以微调到许多其他序列分析任务。