GTEx,The Genotype-Tissue Expression (GTEx) project,首次被提出来是2013年,上百位科学家联名在Nature Genetics杂志发表的文章首次介绍了“基因型-组织表达工程”,并成立了“基因型-组织表达研究联盟”(Genotype-Tissue Expression Consortium,GTEx)以下简称“GTEx”)。
已经发表的相关文献见:https://gtexportal.org/home/publicationPage 除了上面在science和nature大量发paper外,还在一下nature子刊发了不少。
这些数据还有着非常大的挖掘价值,希望大家踊跃思考!
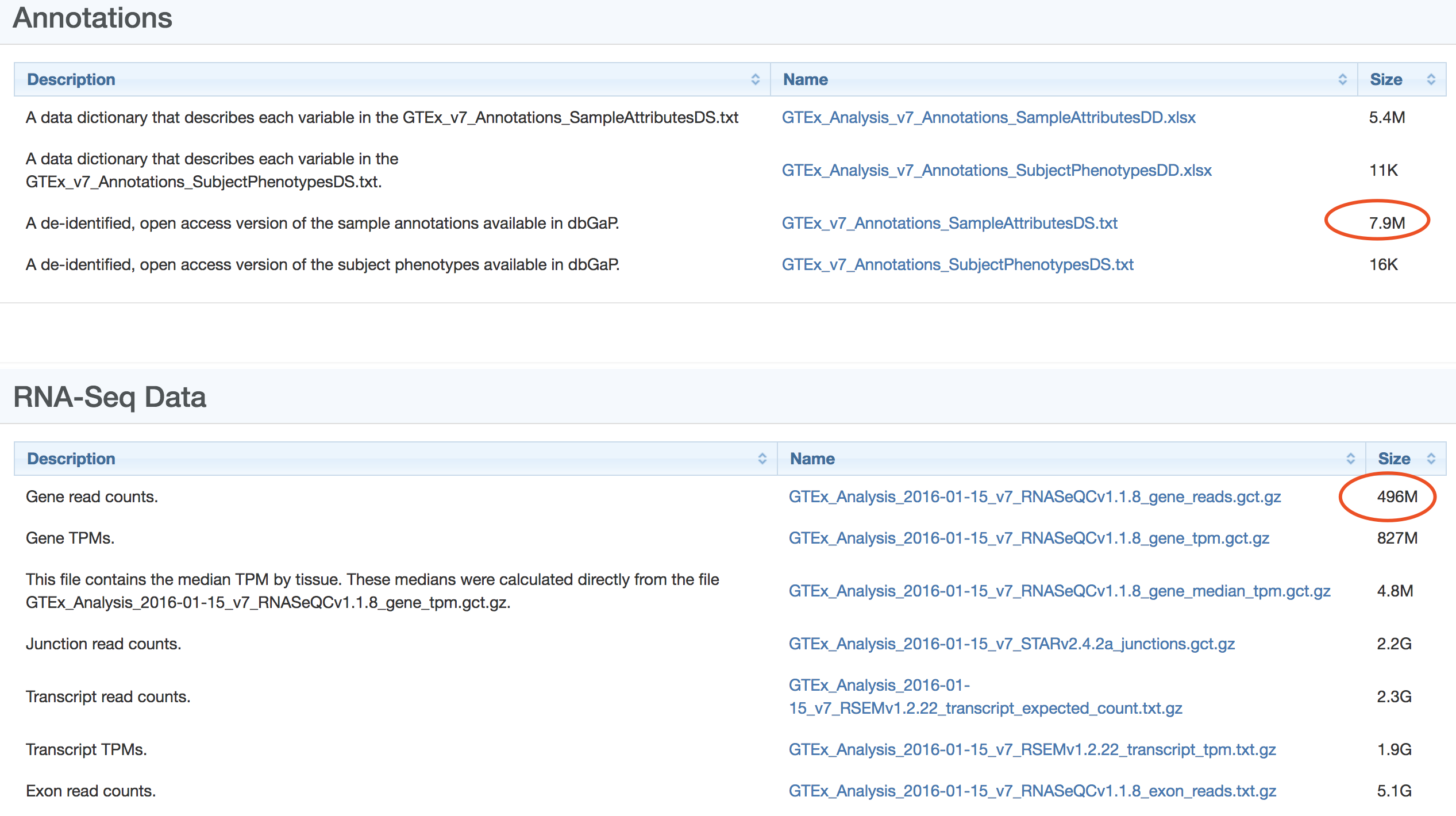
这个文件有点大,注意网速。
GTEx_Analysis_2016-01-15_v7_RNASeQCv1.1.8_gene_reads.gct.gz
如下所示:
代码是:
options(stringsAsFactors = F)
GTEx=read.table('~/Downloads/GTEx_Analysis_2016-01-15_v7_RNASeQCv1.1.8_gene_reads.gct.gz'
,header = T,sep = '\t',skip = 2)
GTEx[1:4,1:4]
h=head(GTEx)
save(h,file = 'GTEx_head.Rdata')
save(GTEx,file = 'GTEx_all.Rdata')在 GTEx_v7_Annotations_SampleAttributesDS.txt
load('~/Desktop/GTEx_all.Rdata')
a[1:4,1:4]
colnames(a)
# SMTS Tissue Type, area from which the tissue sample was taken.
# SMTSD Tissue Type, more specific detail of tissue type
b=read.table('GTEx_v7_Annotations_SampleAttributesDS.txt',
header = T,sep = '\t',quote = '')
table(b$SMTS)
breat_gtex=a[,gsub('[.]','-',colnames(a)) %in% b[b$SMTS=='Breast',1]]
rownames(breat_gtex)=a[,1]
dat=breat_gtex
ids=a[,1:2]
head(ids)
colnames(ids)=c('probe_id','symbol')
dat=dat[ids$probe_id,]
dat[1:4,1:4]
ids$median=apply(dat,1,median)
ids=ids[order(ids$symbol,ids$median,decreasing = T),]
ids=ids[!duplicated(ids$symbol),]
dat=dat[ids$probe_id,]
rownames(dat)=ids$symbol
dat[1:4,1:4]
breat_gtex=dat
save(breat_gtex,file = 'breat_gtex_counts.Rdata')理论上PAM50是针对乳腺癌患者的表达矩阵,这里仅仅是运行代码:
load(file = 'breat_gtex_counts.Rdata')
dat=log2(edgeR::cpm(breat_gtex)+1)
dat[1:4,1:4]
if(T){
ddata=t(dat)
ddata[1:4,1:4]
s=colnames(ddata);head(s)
library(org.Hs.eg.db)
s2g=toTable(org.Hs.egSYMBOL)
g=s2g[match(s,s2g$symbol),1];head(g)
# probe Gene.symbol Gene.ID
dannot=data.frame(probe=s,
"Gene.Symbol" =s,
"EntrezGene.ID"=g)
ddata=ddata[,!is.na(dannot$EntrezGene.ID)]
dannot=dannot[!is.na(dannot$EntrezGene.ID),]
head(dannot)
library(genefu)
# c("scmgene", "scmod1", "scmod2","pam50", "ssp2006", "ssp2003", "intClust", "AIMS","claudinLow")
s<-molecular.subtyping(sbt.model = "pam50",data=ddata,
annot=dannot,do.mapping=TRUE)
table(s$subtype)
tmp=as.data.frame(s$subtype)
subtypes=as.character(s$subtype)
}
library(genefu)
pam50genes=pam50$centroids.map[c(1,3)]
pam50genes[pam50genes$probe=='CDCA1',1]='NUF2'
pam50genes[pam50genes$probe=='KNTC2',1]='NDC80'
pam50genes[pam50genes$probe=='ORC6L',1]='ORC6'
x=dat
x=x[pam50genes$probe[pam50genes$probe %in% rownames(x)] ,]
tmp=data.frame(subtypes=subtypes)
rownames(tmp)=colnames(x)
library(pheatmap)
pheatmap(x,show_rownames = T,show_colnames = F,
annotation_col = tmp,
filename = 'ht_by_pam50_raw.png')
x=t(scale(t(x)))
x[x>1.6]=1.6
x[x< -1.6]= -1.6
pheatmap(x,show_rownames = T,show_colnames = F,
annotation_col = tmp,
filename = 'ht_by_pam50_scale.png')
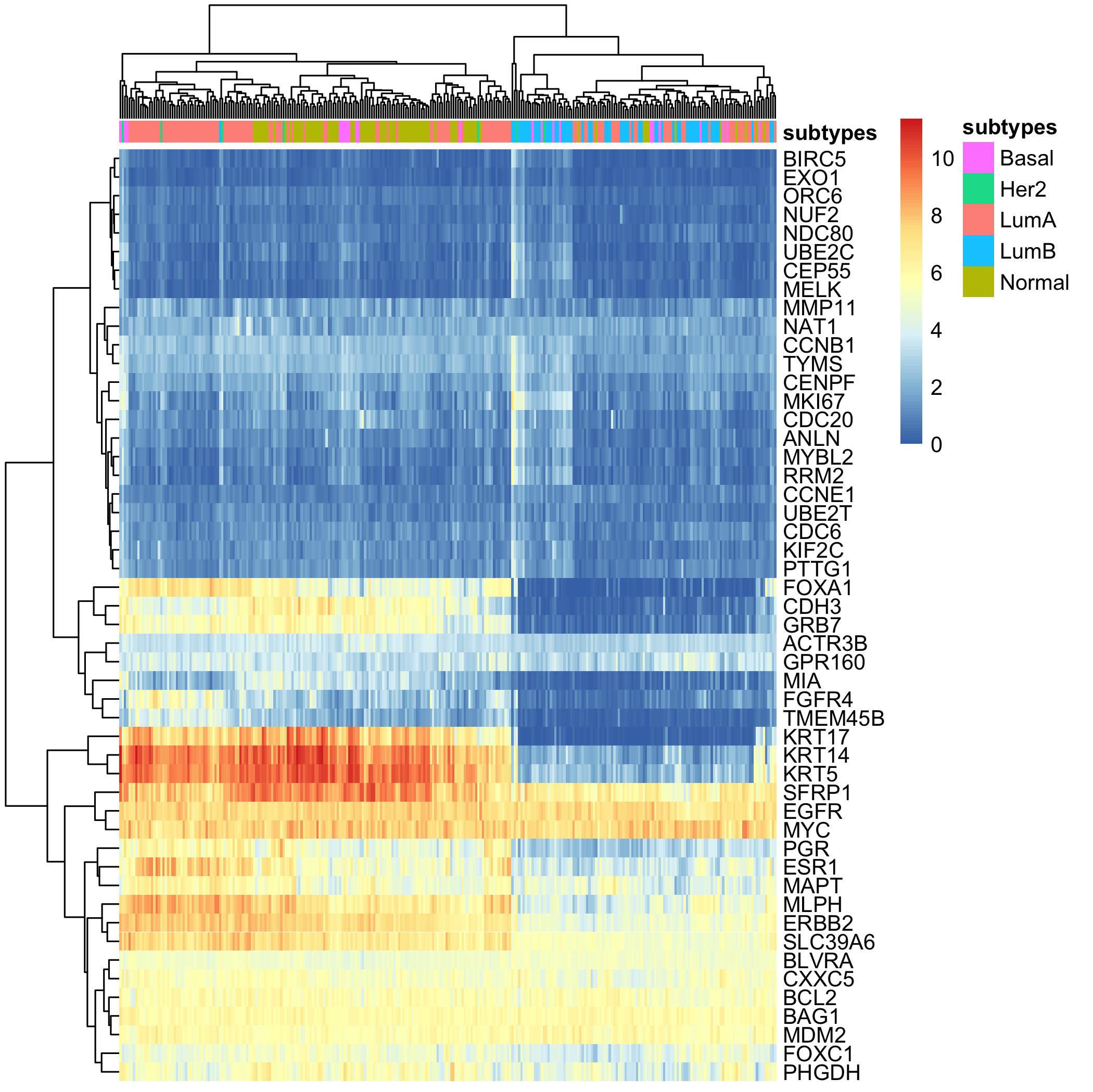
首先是原始表达量的热图:
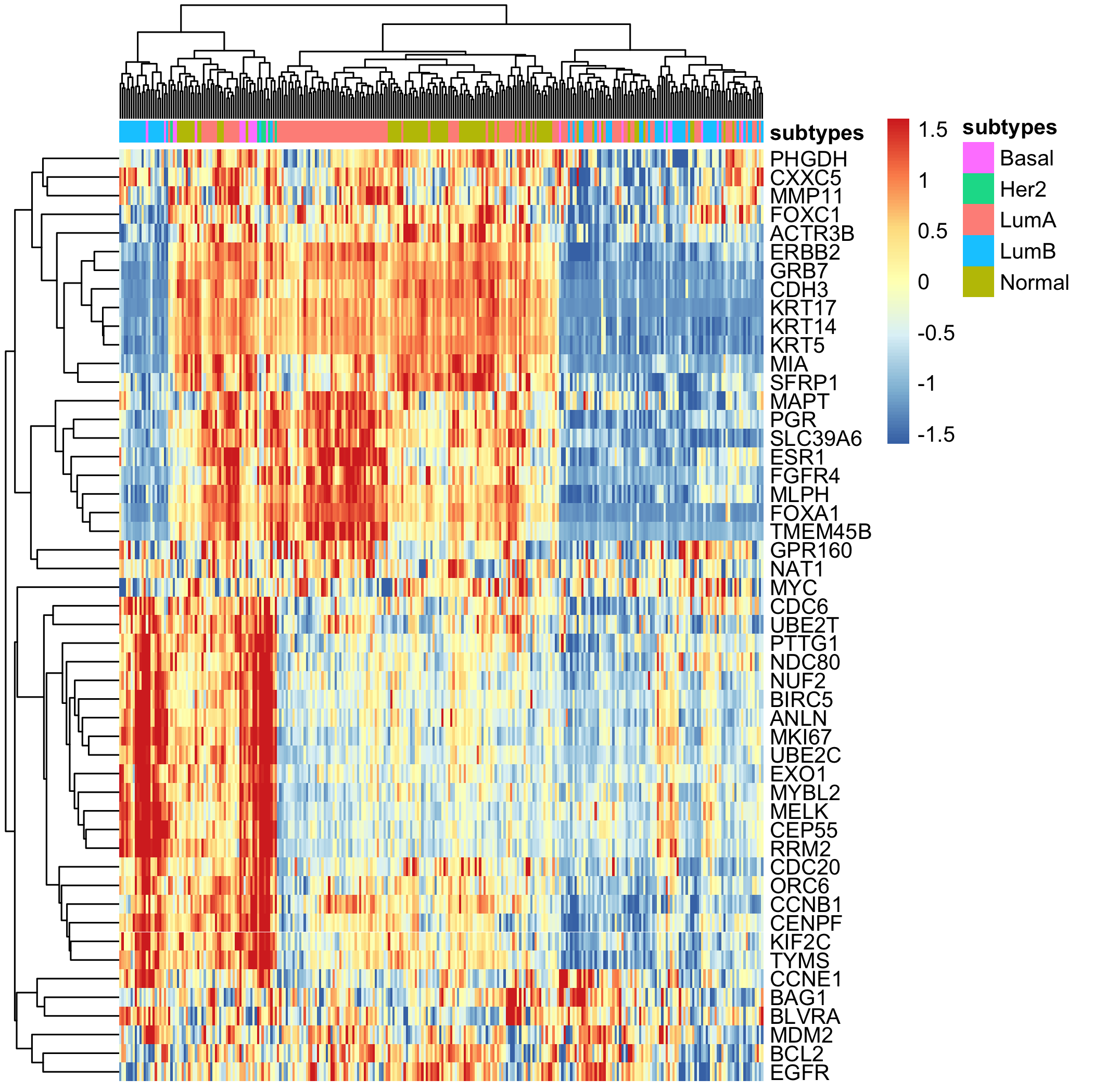
上面的图没办法得知感兴趣基因在不同样本的表达量高低,所以需要归一化:
相信经过这个简单的教程,你对GTEX数据库应该是有所了解了,而且也很容易把数据拿到,利用到自己的课题里面。
如果需要更深入的分析,欢迎发邮件咨询我哈。