El objetivo de la práctica es abordar un problema de data mining realista siguiendo la metodología y buenas prácticas explicadas durante las clases teóricas.
Dicho lo cual, en estas instrucciones no se especifican los pasos exactos que el alumno tiene que llevar a cabo para realizar esta tarea con éxito. Es parte del trabajo aplicar las técnicas de procesamiento/transformación de variables que mejor se acondicionen al problema, identificar los modelos adecuados que proporcionen buenas prestaciones así como las variables potencialmente más relevantes, y elegir la métrica adecuada para contrastar los distintos modelos.
Las posibilidades son amplias, así que es recomendable abordar una aproximación incremental, esto es, comenzar por soluciones sencillas para progresivamente aumentar la complejidad de las técnicas utilizadas.
Se trabajará con uno de los dataset que ofrece SAS Institute para las prácticas y exámenes de data mining para resolver un problema de modelización.
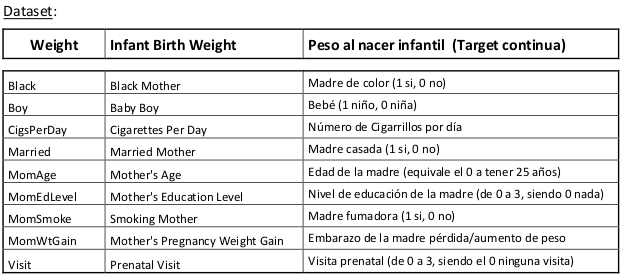
La descripción dada del dataset es la siguiente:
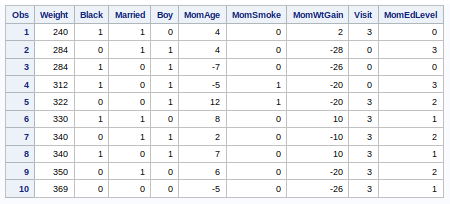
Para realizar el análisis exploratorio creamos una librería, cargamos el conjunto de datos y mostramos los primeros registros
*conexion de libreria;
libname lib_prac '/home/u38080140/ivanrubiomoreno/Practica';
* Cargamos el dataset ;
* Tenemos 50000 observations and 10 variables.
data bweight;
set lib_prac.bweight;
run;
proc print data= lib_data.babyWeigths (obs=10);
run;Al cargar el dataset vemos que tiene 50.000 filas y las 10 variables que se indicaban en la descripción del dataset.
Confirmamos que tenemos como variables categóricas categóricas las columnas Black, Married, Boy MomSmoke y MomEdLevel.
El campo MomAge tiene la edad de la madre, pero los valores no son la edad real. Los valores van a escala. 0 indica que tiene 25 años. Si tiene menos de 25 tiene valores negativos y si tiene mas de 25 años tiene valores positivos. Vamos a sumar 25 al campo MomAge para tener los valores reales de edad y así ser más legible a primera vista.
data bweight;
set bweight;
MomAge = MomAge + 25;
run;Hago un poco de limpieza eliminando las observaciones duplicadas:
proc sort nodupkey data= bweight;
by _all_;
run;Después de hacer limpieza nos quedamos con 48734 y 10 variables.
La variable objetivo de denomina weight. Vamos a analizar el contenido de esta variable mediante una tabla de frecuencias.
proc freq data=bweight;
tables weight;
run;
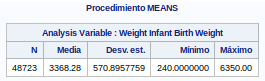
proc means data= bweight;
var weight ;
run;
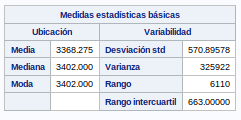
proc univariate data=bweight normal plot;
var weight;
qqplot Weight / NORMAL (MU=EST SIGMA=EST COLOR=RED L=1);
HISTOGRAM /NORMAL(COLOR=MAROON W=4) CFILL = BLUE CFRAME = LIGR;
INSET MEAN STD /CFILL=BLANK FORMAT=5.2;
run;
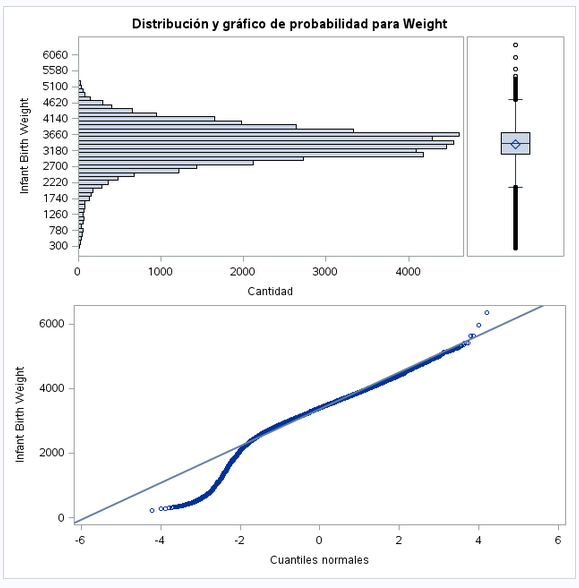
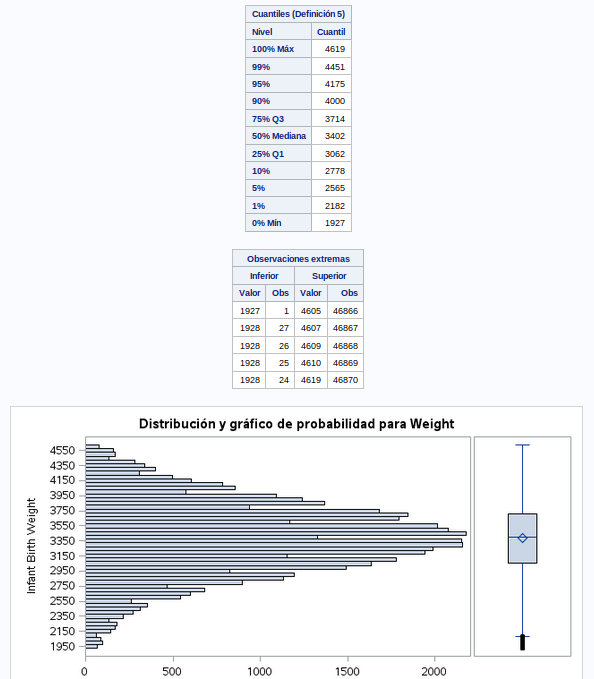
Por suerte vemos que todas las filas tienen valor en la variable objetivo.
Vemos que los datos están muy repartidos. Hay que analizar si tenemos outliers.
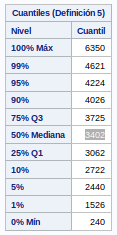
Se ve que tanto por arriba y por abajo hay algunos valores que se desvían mucho de la media, la mediana y la moda. Un 1% tienen valores por debajo de 1526 y otro 1% valores por encima de 4621. Decido eliminar ese 1% por encima y debajo ya que creo que es ruido que puede afectar negativamente a mi modelo.
data bweight;
set bweight;
if weight <= 1526 or weight >= 4621 then delete;
run;Después de eliminar estas observaciones nos quedamos con 47741 observaciones.
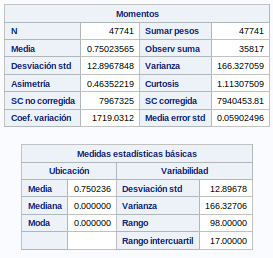
Vamos analizar la variables independientes. En primer lugar vamos a sacar una tabla con las medias de las variables numéricas:
proc means data=bweight;
var _numeric_;
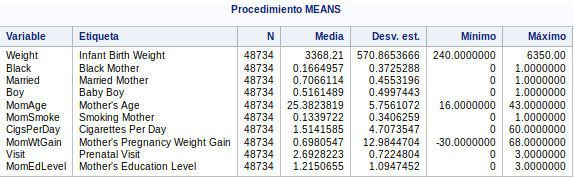
run;Vemos que ninguna variable tiene valores "missing". Todas tienen 48734 valores. Vemos que la desviación estándar no tiene valores muy altos (menos en la variableobjetivo).
Lo siguiente que hacemos es un TTest con la opción de gráficos activada:
ods graphics on;
proc ttest data=bweight;
var _numeric_;
run;
ods graphics off; Vamos a analizar la información que nos proporciona SAS:
- Como ya hemos visto antes, el peso sigue una distribución normal.
- La distribución estándar está muy alejada de la media.
- Hay outliers por arriba y por abajo.
- La mayoría de los datos siguen la recta Q-Q. Los valores más bajos son los que salen más desviados. Habría que averiguar si además de nacimientos también se han incluido abortos. En esos casos es posible que los fetos al no estar desarrollados arrojen estos datos que se desvían del resto.
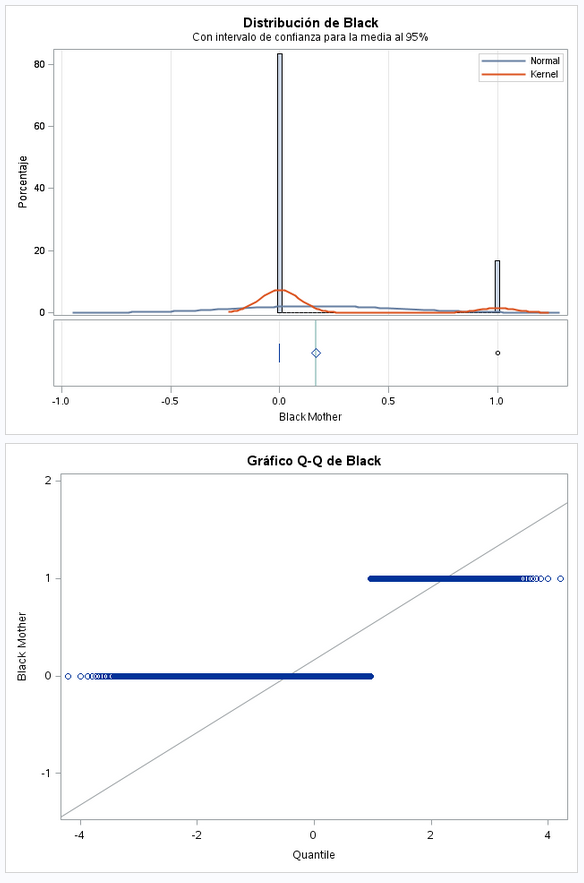
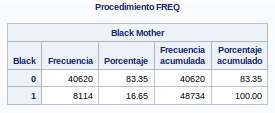
Este es un campo dicotómico. Vemos que hay muchos mas que no son de raza que negra que los que si.
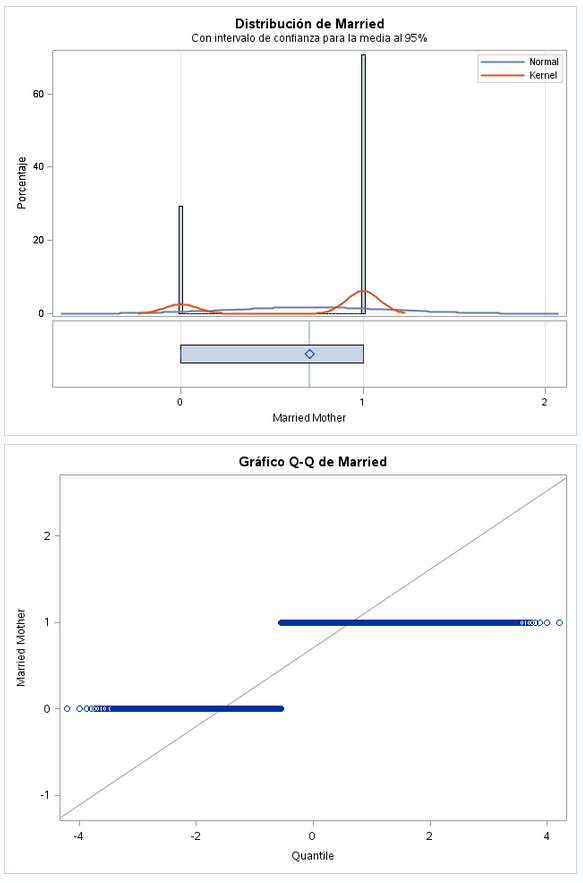
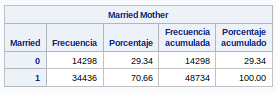
También es una variable dicotómica. En este caso vemos que hay más nacimientos de madres casadas que solteras.
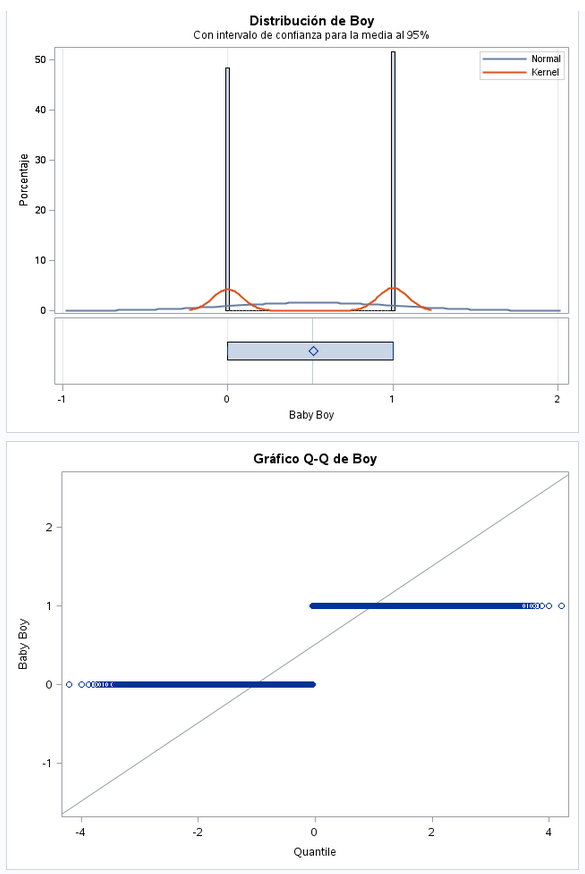
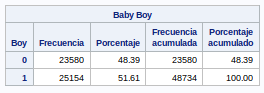
Variable dicotómica. Como se ve en la gráfica, en la media y la desviación estándar los valores está muy repartidos.
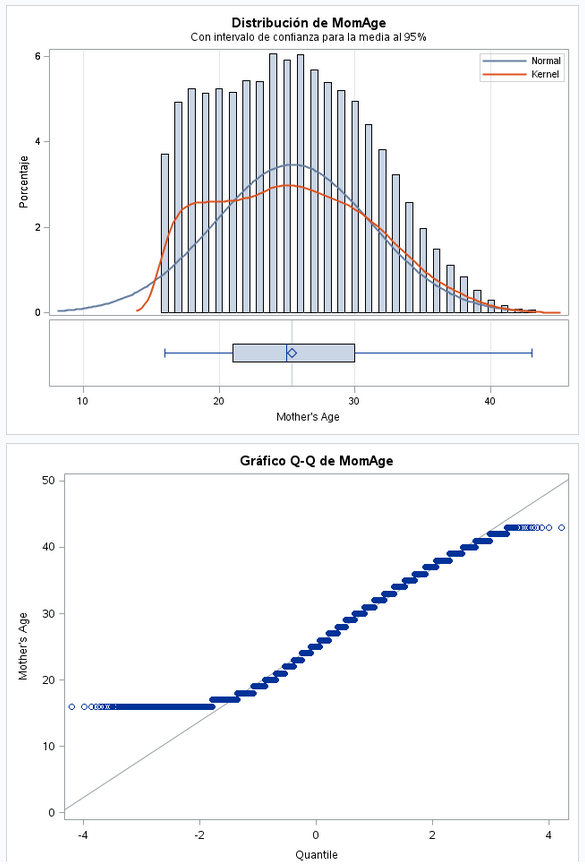
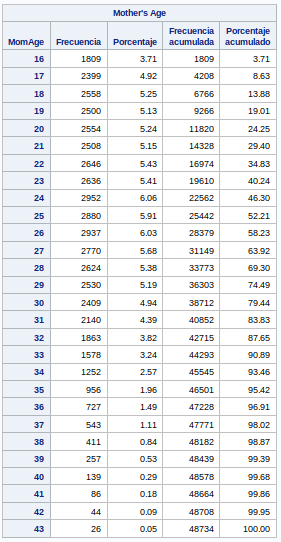
Los datos se desvían un poco de una distribución normal. El valor mínimo es 16 años y el máximo 43 años. Es lógico que no haya datos de edades muy bajos y muy altos.
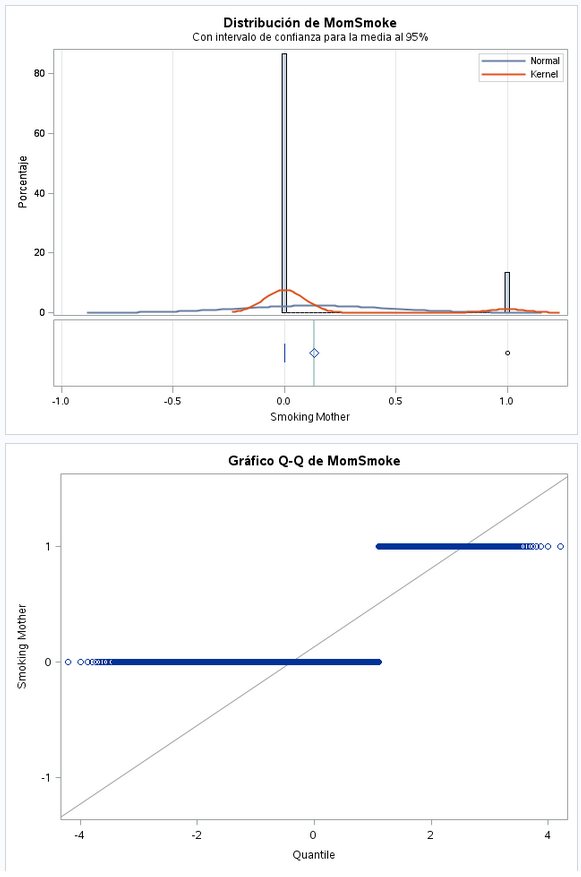
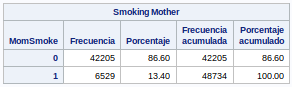
Variable dicotómica. Vemos que hay muchas menos madres fumadoras que no fumadoras.
Lógicamente, al haber muchas menos madres no fumadoras es normal que el valor que más se repita sea el 0.
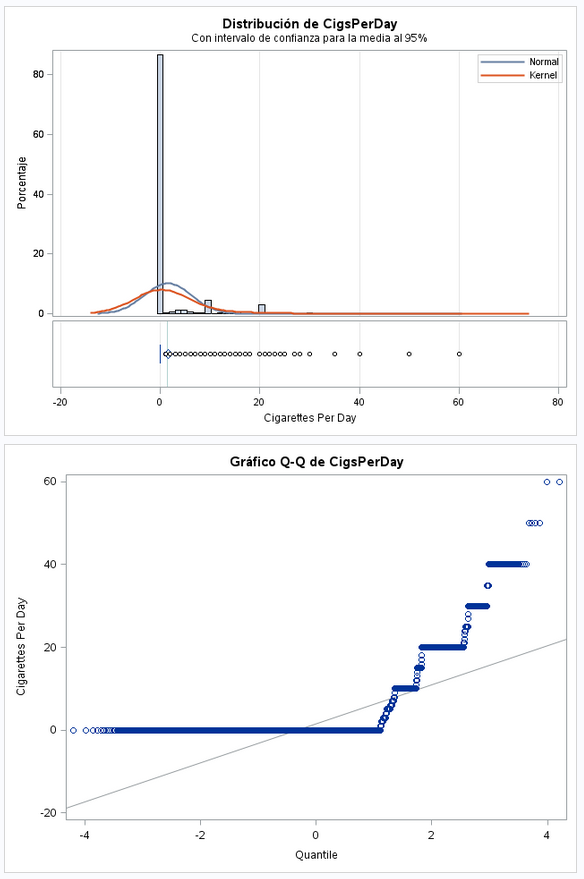
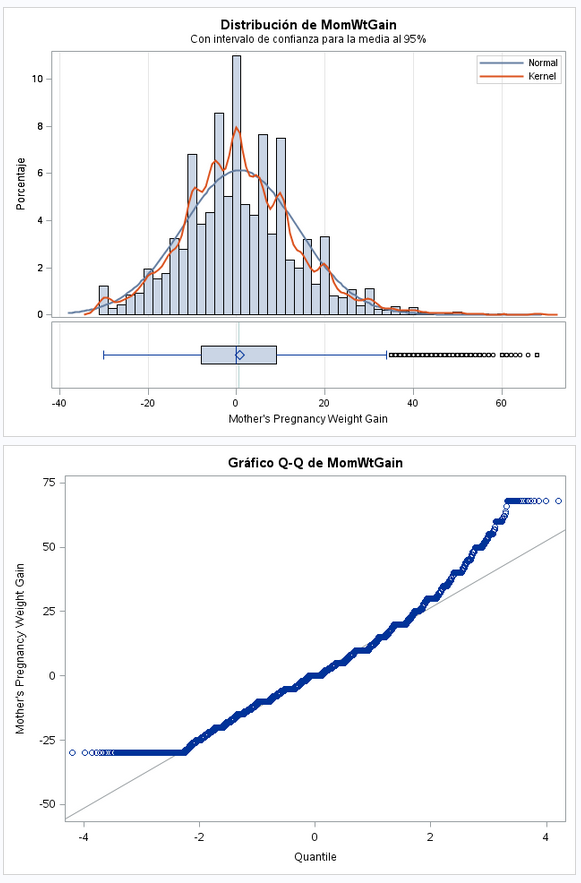
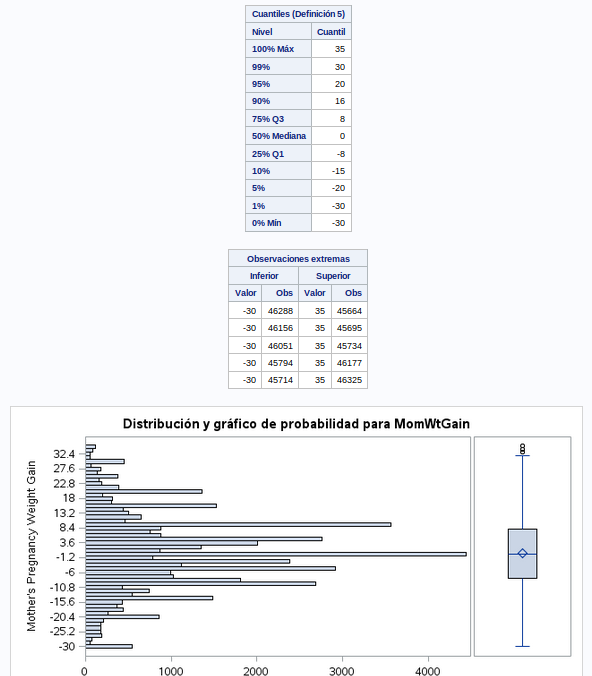
Este dato tiene una distribución muy extraña. Podría haber seguido una distribución normal, pero hace unos picos un poco raro. En el Q-Q podemos ver como se escalona. La desviación estándar es bastante superior a la media. Se pueden ver valores negativos muy grandes lo cual parece raro y en valores positivos vemos muchos ourliers.
Vamos ha hacer un estudio más detallado de esta variable.
proc univariate data=bweight normal plot;
var MomWtGain;
qqplot MomWtGain / NORMAL (MU=EST SIGMA=EST COLOR=RED L=1);
HISTOGRAM /NORMAL(COLOR=MAROON W=4) CFILL = BLUE CFRAME = LIGR;
INSET MEAN STD /CFILL=BLANK FORMAT=5.2;
run;Con los datos obtenidos decido eliminar del dataset el 1% de los pesos positivos para quitar los outliers que puedan meter ruido en los modelos, por lo que hay que eliminar las observaciones con valores mayores de 36.
data bweight;
set bweight;
if MomWtGain >= 36 then delete;
run;Después de esta limpieza nos quedamos con 47232 observaciones.
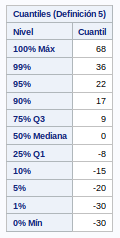
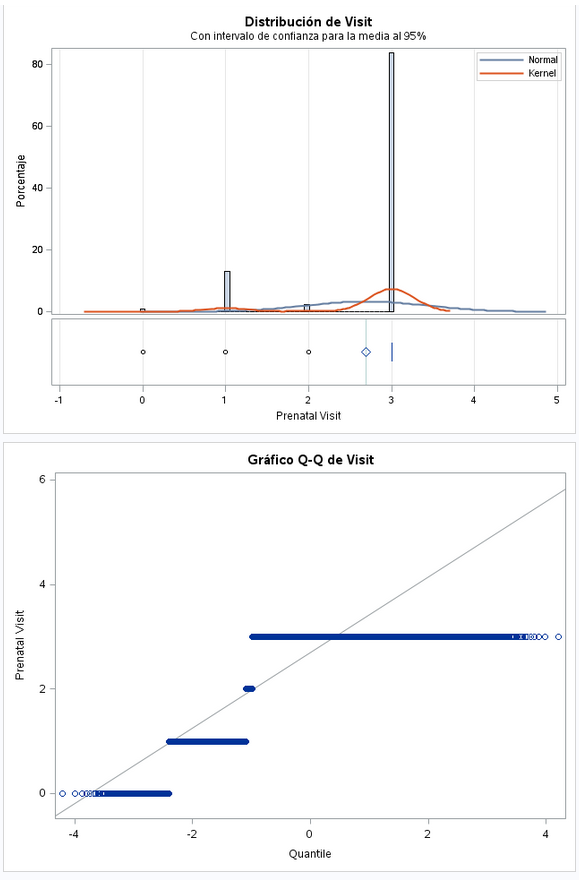
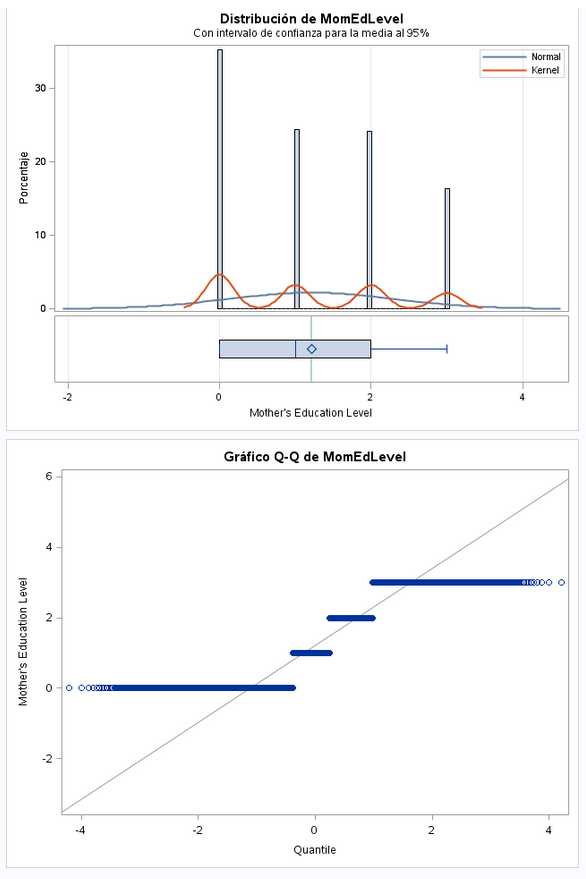
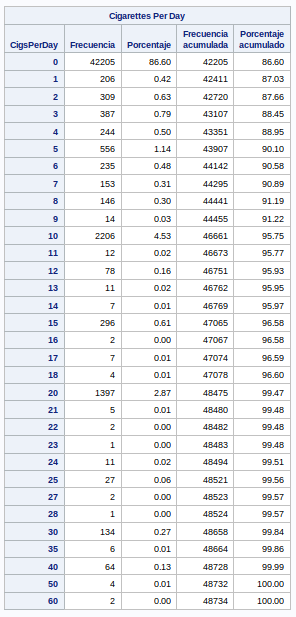
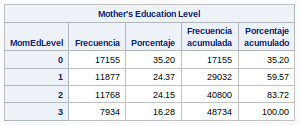
Este dato tiene tres posibles valores: Del 0 al 3. La distribución no es normal. Más adelante veremos valores concretos, pero el 3 es el valor que más se repite.
Los datos están bastante repartidos entre los cuatro posibles valores. La media y la desviación estándar tienen valores muy cercanos.
Voy obtener tablas de frecuencias de las variables independientes para comprobar si tienen valores todas las observaciones.
proc freq data=bweight;
tables black;
run;Vemos que no tenemos valores a missing, por lo que esta variable la dejamos como está.
proc freq data=bweight;
tables married;
run;Al igual que la variable anterior, en el caso de married no tenemos valores a missing, por lo que esta variable la dejamos como está.
proc freq data=bweight;
tables boy;
run;No tenemos valores a missing. La variable la dejamos como viene de origen.
proc freq data=bweight;
tables MomAge;
run;No tenemos valores a missing. La variable la dejamos como viene de origen.
proc freq data=bweight;
tables MomSmoke;
run;No tenemos valores a missing. La variable la dejamos como viene de origen.
proc freq data=bweight;
tables CigsPerDay;
run;No tenemos valores a missing. La variable la dejamos como viene de origen.
proc freq data=bweight;
tables MomWtGain;
run;En este caso no muestro la tabla de frecuencias, porque tiene muchos valores distintos y no me cabe. Aunque puedo decir que no tiene valores a missing, así que queda como está.
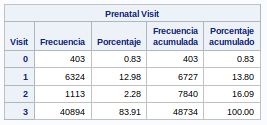
proc freq data=bweight;
tables Visit;
run;No tenemos valores a missing. La variable la dejamos como viene de origen.
proc freq data=bweight;
tables MomEdLevel;
run;No tenemos valores a missing. La variable la dejamos como viene de origen.
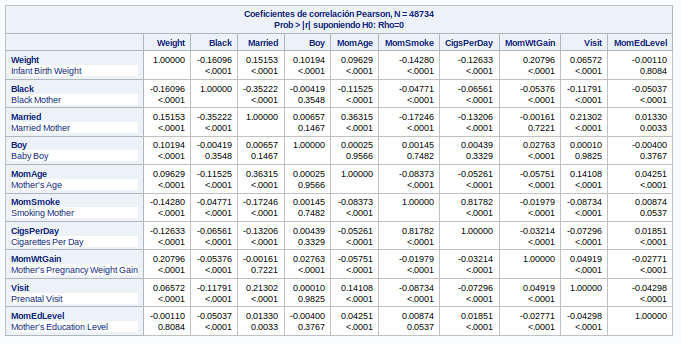
Vamos a sacar la tabla con los coeficientes de correlación para ver si hay variables que estén muy correladas entre si:
proc corr data=bweight;
var _numeric_;
run;En la tabla de correlaciones no se observan valores por encima de 90. El valor más alto es de 0.82 para las variables MomSmoke y CigsPerDay. Claramente estas variables está relacionadas por lo que voy a eliminar la columna CigsPerDay ya es que menos informativa.
Al eliminar la variable CigsPerDay del conjunto de datos, al eliminar duplicados seguimos manteniendo las 48734 observaciones.
Vamos a usar GMLSelect para buscar las mejores variables independientes para un modelo predictivo. Para ello creo una macro que ejecute varios modelos con distintos porcentajes, semillas y métodos de selección. Los resultados se guardarán en un fichero para su posterior estudio.
%let lib1 = '/home/u38080140/ivanrubiomoreno/output/glm_bweight.txt';
%macro glm_select (t_input, vardepen, varcategoricas, varindepen, interacciones, frac_ini, frac_fin, semi_ini, semi_fin, seleccion, selecc_name);
%do frac = &frac_ini. %to &frac_fin.;
data;
fra=&frac/10;
call symput('porcen',left(fra));
run;
%do semilla = &semi_ini. %to &semi_fin.;
ods graphics on;
ods output SelectionSummary=modelos;
ods output SelectedEffects=efectos;
ods output Glmselect.SelectedModel.FitStatistics=ajuste;
proc glmselect data=&t_input. plots=all seed=&semilla;
partition fraction(validate=&porcen);
class &varcategoricas.;
model &vardepen. = &varindepen. &interacciones.
/ selection=&seleccion details=all stats=all; run;
ods graphics off;
ods html close;
data union&semilla.;
i=12;
set efectos;
set ajuste point=i;
run; *observación 12 ASEval;
data _null_;
semilla=&semilla;
selecc_name=&selecc_name;
file &lib1 mod;
set union&semilla.;
put effects @201 nvalue1 @215 semilla @225 selecc_name;
run;
proc sql; drop table union&semilla.; quit;
%end;
%end;
/*proc sql; drop t able modelos,efectos,ajuste,union; quit;*/
%mend;Creo otra macro que invoca a la anterior con los métodos de selección stepwise_validate, stepwise_cv, forward y backward.
%macro glm_select_selections (t_input, vardepen, varcategoricas, varindepen, interacciones, frac_ini, frac_fin, semi_ini, semi_fin);
/* borramos el contenido del fichero */
data _null_;
file &lib1 OLD;
run;
%glm_select(&t_input.,
&vardepen., &varcategoricas., &varindepen., &interacciones.,
&frac_ini., &frac_fin., &semi_ini., &semi_fin.,
stepwise(select=aic choose=validate), 'stepwise_validate');
%glm_select(&t_input.,
&vardepen., &varcategoricas., &varindepen., &interacciones.,
&frac_ini., &frac_fin., &semi_ini., &semi_fin.,
stepwise(select=aic choose=cv), 'stepwise_cv');
%glm_select(&t_input.,
&vardepen., &varcategoricas., &varindepen., &interacciones.,
&frac_ini., &frac_fin., &semi_ini., &semi_fin.,
forward, 'forward');
%glm_select(&t_input,
&vardepen, &varcategoricas, &varindepen, &interacciones,
&frac_ini, &frac_fin, &semi_ini, &semi_fin,
backward, 'backward');
%mend;Ahora toca hacer pruebas.
Como vaiables categóricas indico: Black Married Boy Visit MomEdLevel MomSmoke Como vaiables independientes indico: MomAge MomWtGain Black Married Boy Visit MomEdLevel MomSmoke Como variables iteraciones indico: MomAgeBlack MomAgeMarried MomAgeBoy MomAgeMomSmoke MomAgeMomWtGain MomAgeVisit MomAge*MomEdLevel
%glm_select_selections(bweight, weight,
Black Married Boy Visit MomEdLevel MomSmoke,
MomAge MomSmoke MomWtGain Black Married Boy Visit MomEdLevel,
MomAge*Black MomAge*Married MomAge*Boy MomAge*MomSmoke MomAge*MomWtGain MomAge*Visit MomAge*MomEdLevel ,
3, 5, 12345, 12349);Obtenemos valores de ASE muy altos. Entre 208000 y 215000.
Lo siguiente que pruebo es no poner iteraciones para ver que variables independientes se repiten más en los modelos y crear combinaciones de estas variables para hacer iteraciones
%glm_select_selections(bweight, weight,
Black Married Boy Visit MomEdLevel MomSmoke,
MomAge MomWtGain Black Married Boy Visit MomEdLevel,
MomSmoke ,
3, 5, 12345, 12346);Las variables MomAge MomWtGain Black Married Boy MomSmoke siempre están presentes en todos los modelos. A veces entran Visit o MomEdLevel o ambas. Así que pruebo como iteraciones las combinaciones de las variables MomAge MomWtGain Black Married Boy Visit MomEdLevel MomSmoke
%glm_select_selections(bweight, weight,
Black Married Boy Visit MomEdLevel MomSmoke,
MomAge MomWtGain Black Married Boy Visit MomEdLevel MomSmoke,
MomAge*MomWtGain MomAge*Black MomAge*Married MomAge*Boy MomAge*Visit MomAge*MomEdLevel MomAge*MomSmoke
MomWtGain*Black MomWtGain*Married MomWtGain*Boy MomWtGain*Visit MomWtGain*MomEdLevel MomWtGain*MomSmoke
Black*Married Black*Boy Black*Visit Black*MomEdLevel Black*MomSmoke
Married*Boy Married*Visit Married*MomEdLevel Married*MomSmoke
Boy*Visit Boy*MomEdLevel Boy*MomSmoke
MomSmoke*MomEdLevel MomSmoke*Visit,
3, 5, 12345, 12349);Seguimos con valores de ASE por encima de 200.000. Entiendo que los modelos están bien y el problema está en que no he cocinado los datos bastante.
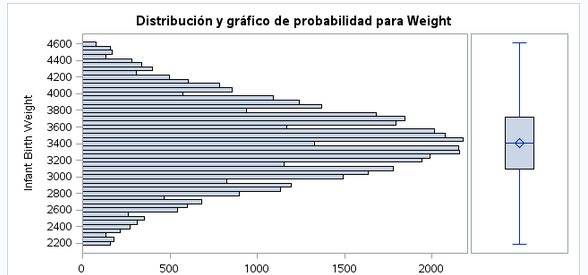
Volvemos a analizar la variable objetivo
proc univariate data=bweight normal plot;
var weight;
qqplot weight / NORMAL (MU=EST SIGMA=EST COLOR=RED L=1);
HISTOGRAM /NORMAL(COLOR=MAROON W=4) CFILL = BLUE CFRAME = LIGR;
INSET MEAN STD /CFILL=BLANK FORMAT=5.2;
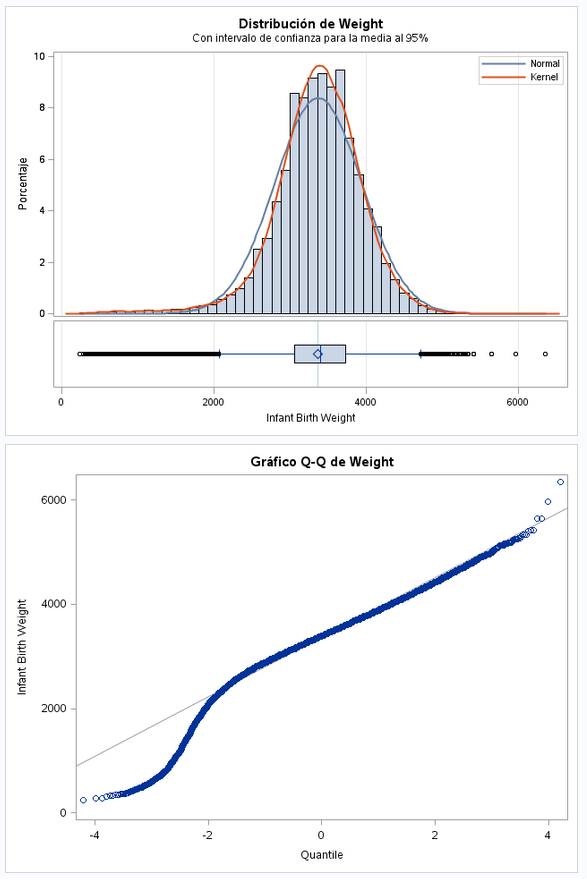
run;Vemos que aún habiendo eliminado varios pesos todavía tenemos outliers en los pesos mínimos. Voy a eliminar el 1% de los pesos inferiores, es decir, los pesos iguales o menores de 2182
data bweight;
set bweight;
if weight <= 2182 or weight >= 4621 then delete;
run;Parece que ya no tenemos outliers en la variable objetivo
Volvemos a estudiar la variable MomWtGain.
proc univariate data=bweight normal plot;
var MomWtGain;
qqplot MomWtGain / NORMAL (MU=EST SIGMA=EST COLOR=RED L=1);
HISTOGRAM /NORMAL(COLOR=MAROON W=4) CFILL = BLUE CFRAME = LIGR;
INSET MEAN STD /CFILL=BLANK FORMAT=5.2;
run;En lo pesos superiores tenemos todavía outliers. Voy a eliminar las observaciones con pesos superiores a 30:
data bweight;
set bweight;
if MomWtGain <= -30 or MomWtGain >= 30 then delete;
run;Tras hacer un poco más de limpieza, vuelvo a sacar unos modelos para ver como ha afectado estos cambios:
%glm_select_selections(bweight, weight,
Black Married Boy Visit MomEdLevel MomSmoke,
MomAge MomWtGain Black Married Boy Visit MomEdLevel MomSmoke,
MomAge*MomWtGain MomAge*Black MomAge*Married MomAge*Boy MomAge*Visit MomAge*MomEdLevel MomAge*MomSmoke
MomWtGain*Black MomWtGain*Married MomWtGain*Boy MomWtGain*Visit MomWtGain*MomEdLevel MomWtGain*MomSmoke
Black*Married Black*Boy Black*Visit Black*MomEdLevel Black*MomSmoke
Married*Boy Married*Visit Married*MomEdLevel Married*MomSmoke
Boy*Visit Boy*MomEdLevel Boy*MomSmoke
MomSmoke*MomEdLevel MomSmoke*Visit,
3, 5, 12345, 12346);Tras la nueva ejecución conseguimos valores de ASE ligeramente inferiores a 200.000.
Vamos a sacar modelos mas numerosos para luego obtener los modelos que más veces se has seleccionados como los mejores:
%glm_select_selections(bweight, weight,
Black Married Boy Visit MomEdLevel MomSmoke,
MomAge MomWtGain Black Married Boy Visit MomEdLevel MomSmoke,
MomAge*MomWtGain MomAge*Black MomAge*Married MomAge*Boy MomAge*Visit MomAge*MomEdLevel MomAge*MomSmoke
MomWtGain*Black MomWtGain*Married MomWtGain*Boy MomWtGain*Visit MomWtGain*MomEdLevel MomWtGain*MomSmoke
Black*Married Black*Boy Black*Visit Black*MomEdLevel Black*MomSmoke
Married*Boy Married*Visit Married*MomEdLevel Married*MomSmoke
Boy*Visit Boy*MomEdLevel Boy*MomSmoke
MomSmoke*MomEdLevel MomSmoke*Visit,
3, 5, 12345, 12400);En total son 660 modelos ejecutados. Ahora vamos a volcar los resultados del fichero en un dataset para ordenar y agrupar los modelos obtenidos y ver cuales han sido los modelos que mas se repiten.
data seleccion;
INFILE '/home/u38080140/ivanrubiomoreno/output/glm_bweight.txt';
length modelo $243;
input modelo $1-200 ase semilla tipo_seleccion $225-242;
run;
proc sort data=seleccion; by modelo;
proc freq data=seleccion; tables modelo / noprint out=frec_modelos; run;
proc sort data=frec_modelos out=modelos_ordenados; by descending count; run;Los modelos que más se repiten son:
- MomWtGain Black Married Boy MomAge*MomSmoke
- MomWtGain Black Married Boy MomEdLevel MomAge*MomSmoke
- Black Married Boy MomEdLevel MomAgeMomSmoke MomWtGainBoy
- MomWtGain Boy MomAgeBoy MomAgeMomSmoke BlackMomSmoke MarriedMomSmoke
Vamos a usar estos modelos en un GLM cada uno para interpretar los resultados que obtenemos:
ods graphics on;
proc glm data=bweight;
class Black Married Boy Visit MomEdLevel MomSmoke;
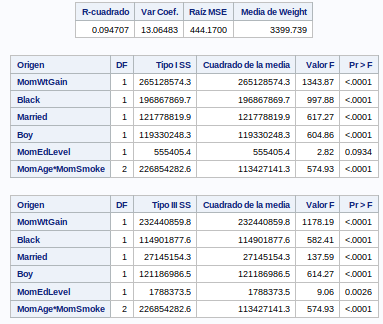
model weight = MomWtGain Black Married Boy MomAge*MomSmoke
/ solution e;
run;
ods graphics off;ods graphics on;
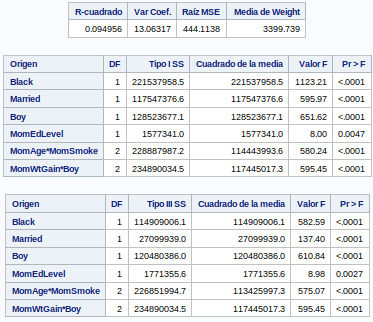
proc glm data=bweight;
class Black Married Boy Visit MomEdLevel MomSmoke;
model weight = MomWtGain Black Married Boy MomEdLevel MomAge*MomSmoke
/ solution e;
run;
ods graphics off;ods graphics on;
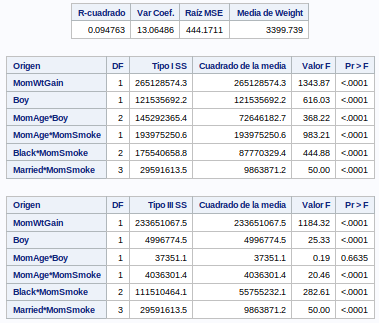
proc glm data=bweight;
class Black Married Boy Visit MomEdLevel MomSmoke;
model weight = Black Married Boy MomEdLevel MomAge*MomSmoke MomWtGain*Boy
/ solution e;
run;
ods graphics off;ods graphics on;
proc glm data=bweight;
class Black Married Boy Visit MomEdLevel MomSmoke;
model weight = MomWtGain Boy MomAge*Boy MomAge*MomSmoke Black*MomSmoke Married*MomSmoke
/ solution e;
run;
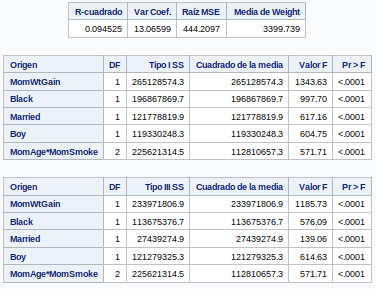
ods graphics off;Nos quedamos con el primer modelo que es el que menor R2 tiene: 0.094525.
Por lo tanto nuestro modelo champion es: MomWtGain Black Married Boy MomAge*MomSmoke
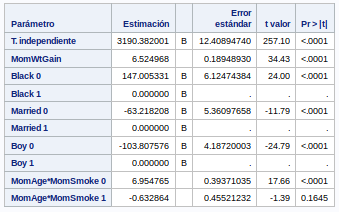
Esta es la tabla de estimaciones que obtenemos de nuestro modelo
Por lo tanto, según nuestro modelo, podemos estimar el valor de weigth con esta formula
weight = 3190.382001 + (MomWtGain * 6.524968) + (Black * 147.005331) + (Married * 63.218208) + (Boy * 103.807576) + (MomAge*MomSmoke0 * 6.954765) + (MomAge*MomSmoke1 * 0.632864 ) + Epsilon
Donde Epsilon = error aleatorio
Sinceramente no tengo ni idea de si esta formula es correcta. En las clases no hemos visto ningún caso parecido. En la consulta que he hecho no he entendido como abordar esta parte.
Vamos a usar dataminer para hacer una comparación de modelos entre un GML y una red neuronal. Para ello creamos una fuente de datos con el dataset de los datos cocinados.
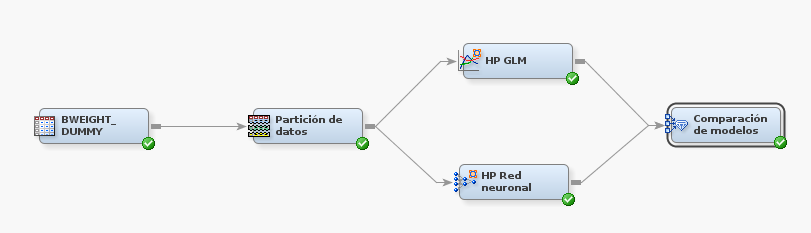
A continuación creamos un diagrama en el que insertamos la fuentes de datos.
Luego insertamos un nodo de partición de datos y cambiamos los conjuntos a:
- Entrenamiento: 70
- Validacion: 15
- Pruebas: 15
Luego incluimos un nodo HP GLM y otro nodo HP Red neuronal.
Por último incluimos un nodo de comparación de modelos.
El diagrama quedaría así:
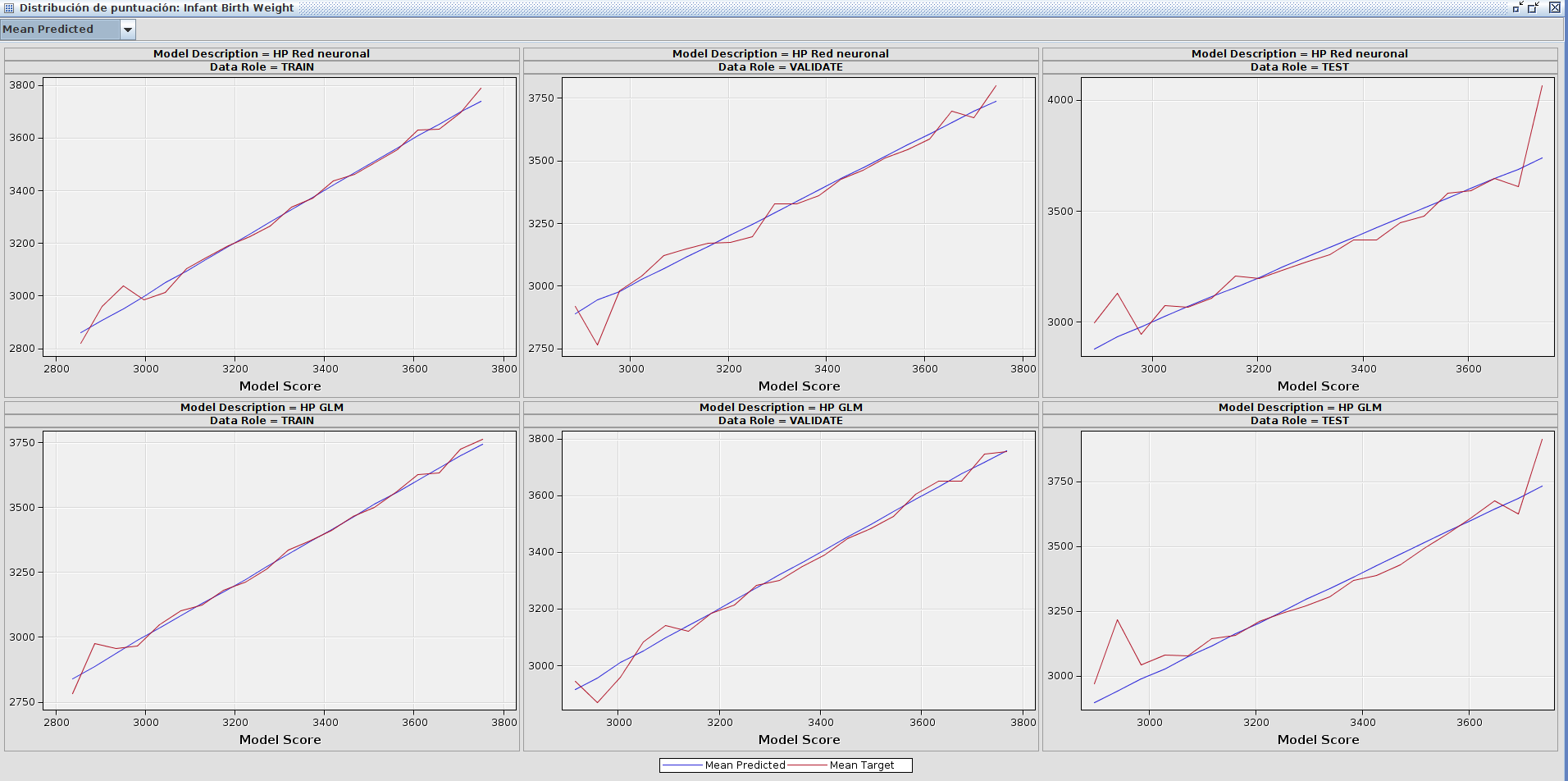
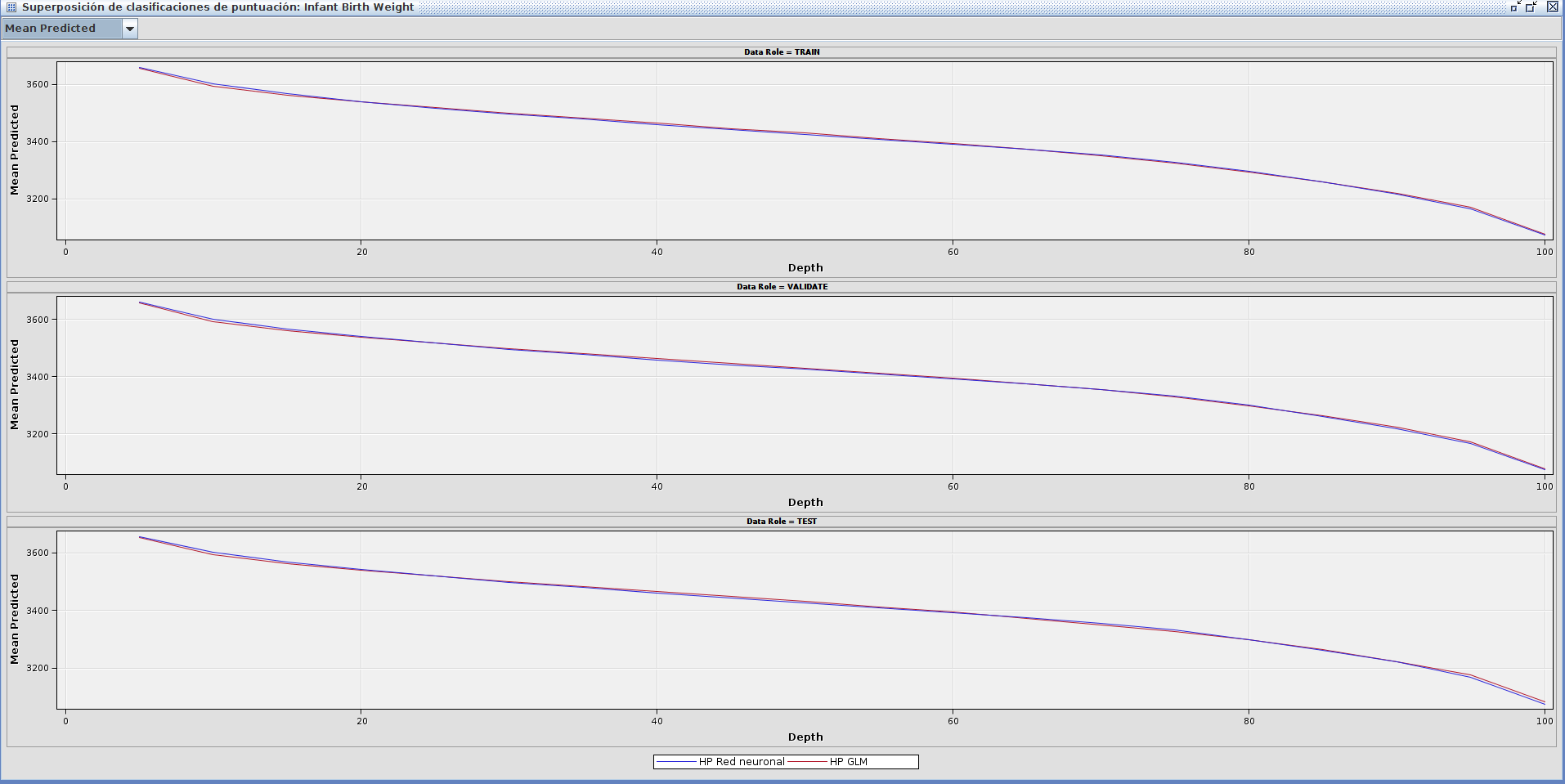
Tras ejecutar el diagrama obtenemos estos resultados:
Como podemos ver albos modelos tienen el mismo ASE de 6764. Sin embargo el la suma y la media de los errores cuadráticos son inferiores para la red neuronal. Por lo tanto podemos decir que el modelo de la red neuronal es algo más óptimo que modelo GLM que ha generado miner.
En las gráficas de puntuación podemos ver que ambos modelos son muy parecidos y describen lineas muy similares.
En las gráficas de clasificación pasa lo mismo. Ambos modelos describen unas líneas muy parejas.
En los datos de salida podemos ver de otra forma los datos de los dos modelos. al igual que en la tabla de ajuste y en las gráficas, vemos que ambos modelos tienen resultados muy similares, aunque en cifras absolutas podemos decir que el modelo de red neuronal da mejores resultados.