重构已经完成,代码更容易阅读,bug修复,增加新功能,但是目前没有教程,欢迎试用。https://github.com/beikwx/SailVina
Autodock Vina对接GUI界面
开发中……,已经完成:
- 准备受体
- 准备配置文件
- 准备配体
- 批量对接
- 生成配体-蛋白复合物
下一步计划:
- 提取分数
- 计算RMSD
方法一:下载main.zip到需要安装的目录,解压,直接运行内部的main.exe文件即可。
软件界面:
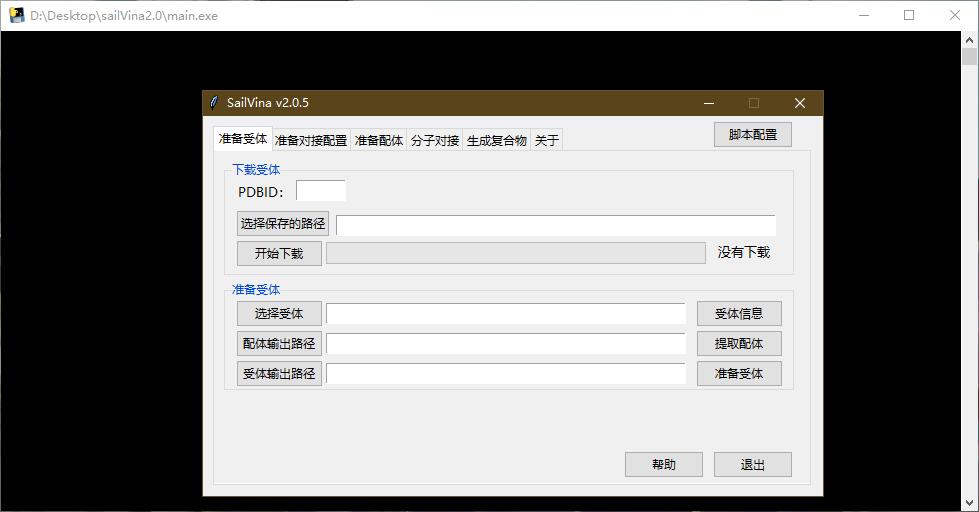 注意:本软件的解压路径不要包含空格和中文!
注意:本软件的解压路径不要包含空格和中文!
方法二:克隆本项目到本地,在SailVina中用python运行main.py
- 需要requests(下载pdb)、biopython(准备受体)模块,使用pip安装即可。
系统需求:
可以通过本教程测试软件是否配置正确,建议使用同样的文件夹。
新建本教程文件夹:
D:/test_sailvina/Ligands/mol
D:/test_sailvina/Ligands/pdbqt
D:/test_sailvina/Proteins/1IEP_imatinib
D:/test_sailvina/Docking_output
- 配置脚本
- 点击脚本配置,点击"选择adt的python路径",选择ADT中的python.exe。比如C:/DrugDesign/mgltools/python.exe。
- 点击"选择obabel.exe的路径",选择安装的obabel的obabel.exe。比如C:/OpenBabel-2.4.1/obabel.exe。点击确定。
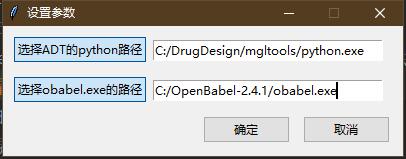 出现路径配置成功表示配置完成。
出现路径配置成功表示配置完成。
- 下载pdb
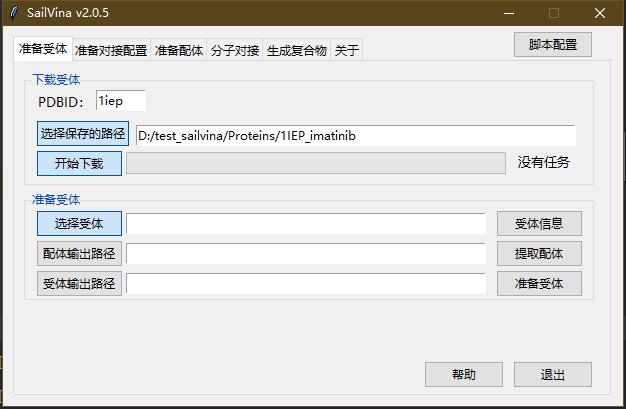
准备受体界面,下载受体。
- PDBID输入1iep,点击"选择保存的路径"选择D:/test_sailvina/Proteins/1IEP_imatinib,点击开始下载。
- 准备受体
准备受体界面,准备受体。
- 点击"选择受体",选择D:/test_sailvina/Proteins/1IEP_imatinib/1iep.pdb
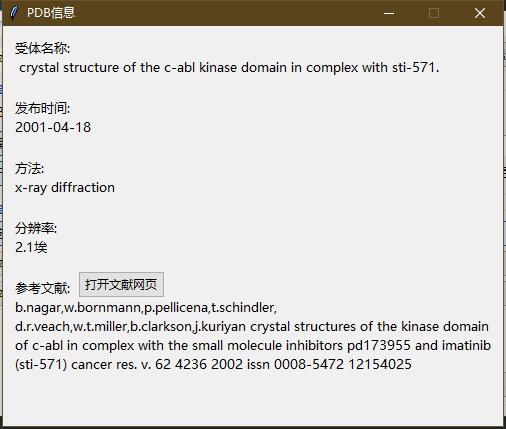
- 点击"受体信息"可以看到选择受体的基本信息,比如名称,结晶方法,打开文献的网页等
- 点击"配体输出路径",选择D:/test_sailvina/Ligands/pdbqt
- 点击"提取配体"。model选择0,下一步,chain选择A,下一步,选择STI点击提取配体。
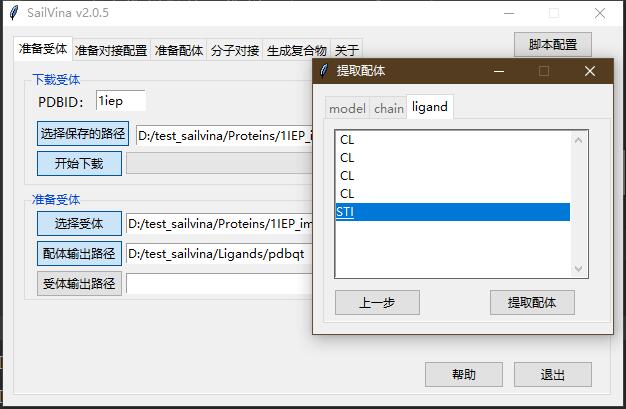 关闭"提取配体"窗口。
关闭"提取配体"窗口。
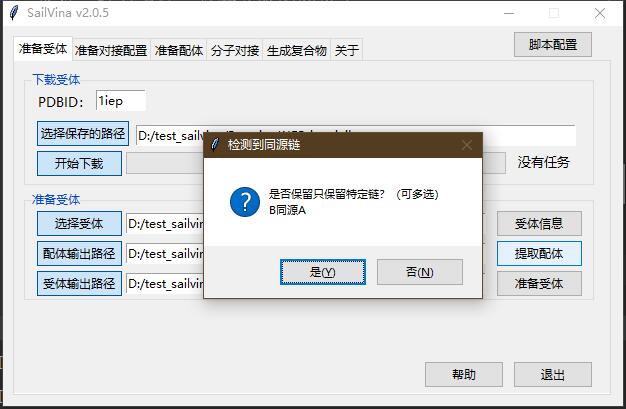
- 点击"受体输出路径",选择D:/test_sailvina/Proteins/1IEP_imatinib。点击准备受体。
- 会自动检测同源链,点击否不删除同源链(测试发现删除同源链会出现一些问题)
- 生成对接config文件
准备对接配置界面,工具
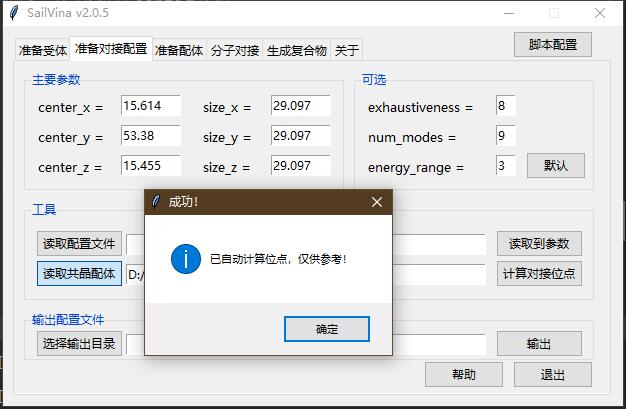
- 点击"读取共晶配体"。选择D:/test_sailvina/Ligands/pdbqt/A_STI.pdbqt
- 点击"计算对接位点",点击"确定",参数会变成如下(由于盒子总大小不能超过27000,所以如果过大需要根据实际需求修改盒子大小)
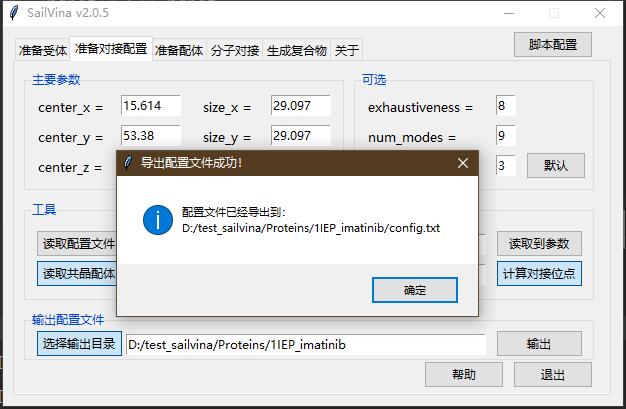
- 点击"选择输出目录",选择D:/test_sailvina/Proteins/1IEP_imatinib,点击"输出"
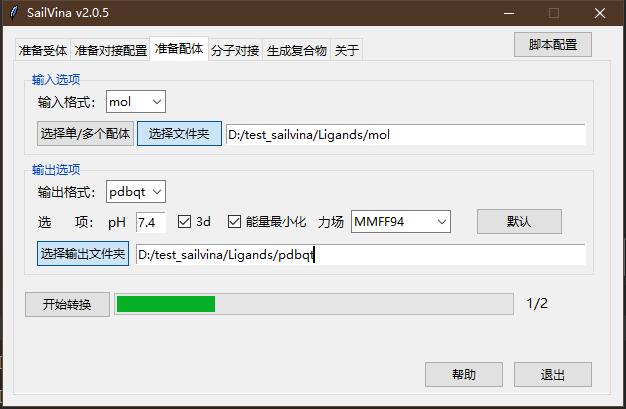
- 准备配体
使用化学作图软件(Chemdraw等)绘制出要对接的配体,保存成mol格式。(作为对接的验证,需要重新绘制原始共晶配体进行再对接re-docking,根据rmsd来判断对接的可靠程度。)本教程绘制2个,伊马替尼和尼洛替尼。保存到D:/test_sailvina/Ligands/mol文件夹中
准备配体界面中,输入选项。
- 输入格式选择mol
- 点击"选择文件夹",选择D:/test_sailvina/Ligands/mol
- 输出选项中,输出格式选择pdbqt,其余保持默认。
- 点击"选择输出文件夹", 选择D:/test_sailvina/Ligands/pdbqt。
- 点击"开始转换"
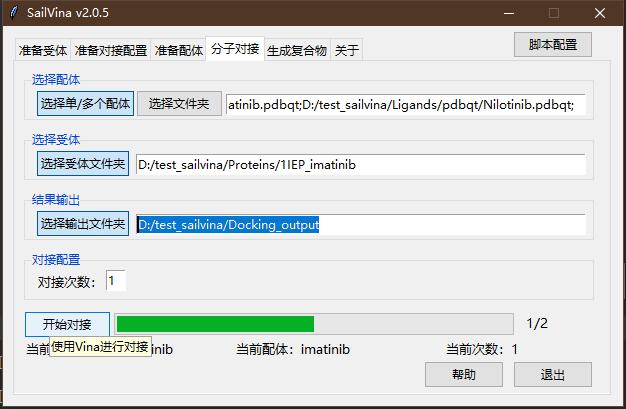
- 分子对接
分子对接界面中
- 选择配体中,点击"选择单/多个配体",选择imatinib.pdbqt和Nilotinib.pdbqt;
- 选择受体中,点击"选择受体文件夹",选择D:/test_sailvina/Proteins/1IEP_imatinib
- 结果输出中,点击"选择输出文件夹",选择D:/test_sailvina/Docking_output
- 点击“开始对接”即可进行对接。(由于Vina对接会消耗大量CPU资源,如果出现未响应为正常现象,请等待对接完成)
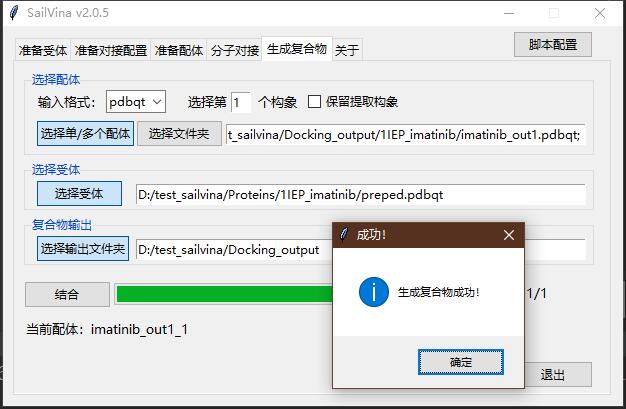
- 生成共晶配合物
生成复合物界面中
- 输入格式选择pdbqt,其余保持默认。
- 点击"选择单/多个配体",选择D:/test_sailvina/Docking_output/1IEP_imatinib/imatinib_out1.pdbqt
- 点击"选择受体", 选择D:/test_sailvina/Proteins/1IEP_imatinib/preped.pdbqt
- 点击"选择输出文件夹",选择D:/test_sailvina/Docking_output
- 点击"结合"即可生成配体受体复合物。
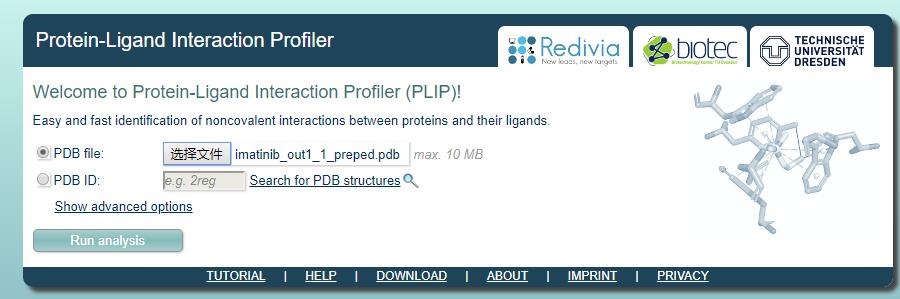
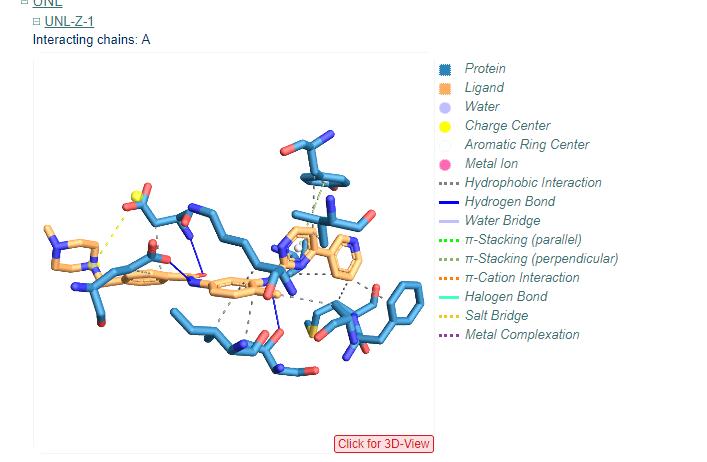
- 将该pdb上传到plip即可查看分子间作用力(注意这里没有水分子,vina对接也没有水分子)
 注意:本软件的解压路径不要包含空格和中文!
注意:本软件的解压路径不要包含空格和中文!










