打开 https://www.broadinstitute.org/medical-and-population-genetics/hapmap-3, 点击 How To Download This Release 下面的 A. SNP Genotype Data 段落的中间3个链接。 文件名字里面有 "b36",现在一般都用 b37(比如 UK Biobank),甚至有的用 b38, 所以下载后解压后需要将那个 .map 文件先用 liftOver 转化为 b37 格式,然后用 PLINK 生成 bed/bim/fam 文件。 这一步已经完成,生成的 PLINK 格式文件已经放到百度网盘,请大家下载。
这个基因数据将作为我们组进行 LDSC 和 GSMR 分析的标准文件。
打开 https://www.internationalgenome.org/data,在 Available data 下面,点击该页面 Phase 3 对应的 VCF 链接, 可以看到以 “ALL.” 开头的文件,可以一个一个直接点击链接下载。 也可以用下面的命令下载, 并且随之将下载的VCF文件转换为PLINK格式 除了下载上述页面上以 “ALL.” 开头的 VCF 文件,倒数第二个 integrated_call_samples_v3.20130502.ALL.panel 文件罗列了每一个样本的人群(pop)和人种 (super_pop),以及性别。 根据这个文件,可以提取特定人种的样本,比如:
- awk '$3=="EUR" {print $1,$1}' integrated_call_samples_v3.20130502.ALL.panel > g1k.EUR.keep
- awk '$3=="EAS" {print $1,$1}' integrated_call_samples_v3.20130502.ALL.panel > g1k.EAS.keep
然后可以用 PLINK --keep g1k.EUR.keep 生成某一个特定人种的基因数据。 当然,如果不想生成太大的基因数据,就只保留一个所有人的数据,后续的PLINK命令记得用 --keep g1k.EUR.keep 就行。 不论是有所有2504个人基因数据的PLINK文件,还是只有某一个人种的PLINK文件,每个染色体都是单独的文件。 后续跑 GWAS 或提取 PRS 的时候,也是每条染色体的数据分开来跑,这样就可以进行并行计算(parallel computing)。 一般不建议把所有的染色体的数据合并成一个完整的单一的基因数据,毕竟将近一个亿的SNP,文件太大了,很多软件根本运行不了。
其实,PLINK的网站上也有千人基因组的数据,点击左下方菜单“1000 genomes phase3” 链接,按照操作下载和处理。 不管是哪个方法得到的PLINK格式的数据,有的软件不允许 .bim 文件里面的 SNP 名字有重复,这个时候可以用下面的命令来处理
- cp chr1.bim chr1.bim.COPY
- awk '{if(array[$2]=="Y") {i++; $2=$2".DUP"i}; print $0; array[$2]="Y"}' chr1.bim.COPY > chr1.bim
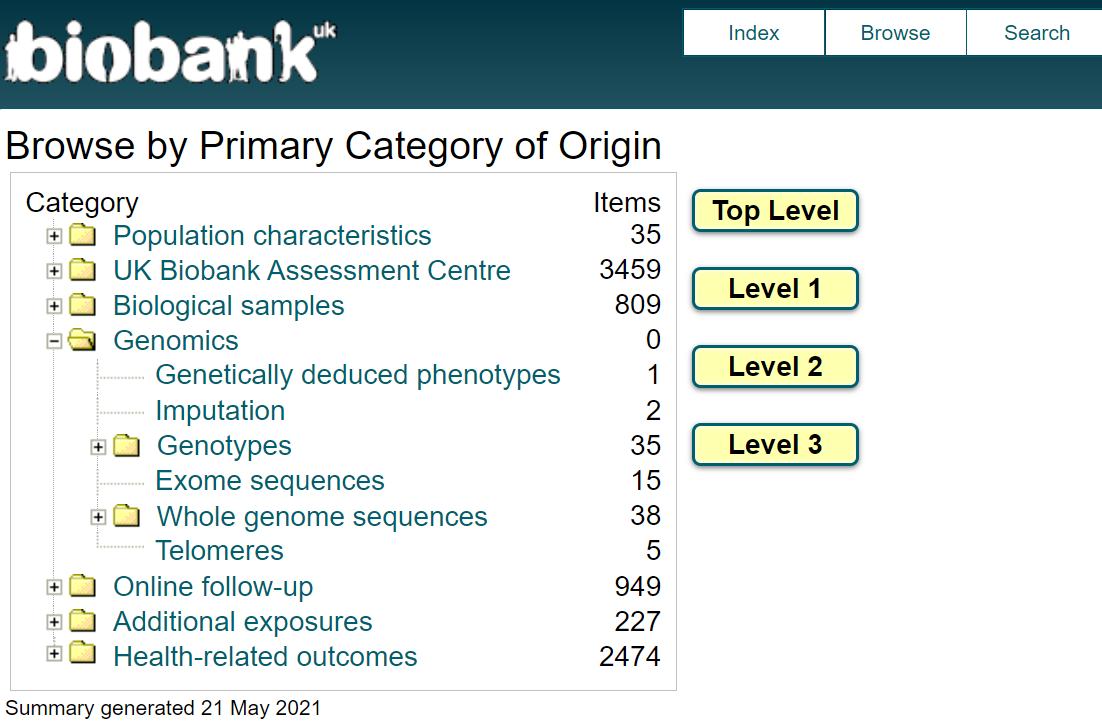
首先要明确,UKB的基因数据很大,所有申请者都能得到一样的数据(样本的ID不一样),一般下载到服务器上去储存和使用。 但是,对于基因型的 summary statistics,是可以人人免费下载的。 UKB里面的将近一亿个 SNP 的 rsID, CHR, POS, MAF等信息,可以点击上面这个页面上的 Imputation。 然后弹出来的页面上会有下面这句话, 点击链接下载就行了。
The information scores and minor allele frequency data for the imputed genotypes (computed with QCTOOL) can also be downloaded in Resource 1967。
对于表型数据的提取,有人做了一个 ukbtools R软件包 但我觉得不是太好用,并且很慢。可以参考这个,用两种不同的方法来提取数据,进行比较。
WINDOWS电脑建议安装系统自带的 Ubuntu Linux系统,然后用 cd /mnt/d/ (而不是 D:/)进入 D 盘。 打开ukbiobank.ac.uk, 点击 Data Showcase 菜单。然后点击第一个“Essential Information”,阅读 Access and using your data。 读完整个文档的话,你就什么都知道了。苹果电脑,参考 https://github.com/spiros/docker-ukbiobank-utils
-
先解压表型数据的大文件
- unbunpack ukb48807.enc e64?3f7811c2c8200ed4ecf?e9a4f32?0b5115aeb77e1cb1eba01f3?4e3463af
- 如果是下载基因数据,第一行写上66137,然后第二行写上上面的密码
-
写一个 VIP.fields.txt 文件,列出想提取的变量和对应的 data-field,比如
- 21022 age
-
然后用下面的命令,提取出该文件的第一列
- awk '{print $1}' ukb.vip.fields > ukb.vip.fields.id
-
确认没有重复的 data-field
- sort ukb.vip.fields.id | uniq -d
-
提前VIP 文件里面列出的变量
- ukbconv ukb42156.enc_ukb r -iukb.vip.fields.id -ovip
-
打开R ,用下面的几行代码,将上面生成的 vip.tab 数据读入,并且给每个变量赋予正确的名字。
- source("D:/vip.r")
- pnames <- read.table("D:/ukb.vip.fields", header=F)
- pnames$V1 <- paste0("f.", pnames$V1, ".0.0")
- phe <- subset(bd, select=grep("f.eid|\.0\.0", names(bd)))
-
上述Linux 系统生成的 vip.r文件,如果在Windows 系统里面运行R,需要将里面的 /mnt/d 改为 D:/。
ICD 这样的指标,包含了很多不同时间的时间点,量很大,建议分开来处理。
ukbconv ukb42156.enc_ukb r -s42170 -oicd sed -i 's/"//g icd.tab
将 icd.tab 文件整合为两列,便于读入R。
cat icd.tab | sed -e 's/\tNA//g' -e 's/\t/,/2g' |
awk '{ if(NR==1) print "IID icd"; else if (NF==1) print $1 " NA"; else print $0"," }' > icd.2cols
提取需要研究的表型数据和相关的covariates,比如 age, sex, PCs。 一般来说,quantitative的表型数据要 adjust for covariates 和转化成正态分布,这个可以在R里面用下面的命令来实现。
trait_res = residuals(lm(trait ~ age+sex+PC1+PC2, na.action=na.exclude) trait_inv = qnorm((rank(trait_res,na.last="keep")-0.5) / length(na.omit(trait_res)))
对于疾病的binary 表型,只需要把需要 adjust 的covarites 和表型数据放在同一个表型数据文件里面, 然后在 GWAS里面的命令指明哪个是表型,哪些是 covariates。
目前GWAS 由专人负责运行,一般来说就是通过下面这样的PLINK命令来跑
for chr in {1..22}; do
- plink2 --memory 12000 --threads 16 --pfile chr$chr --extract ukb.chr$chr.good.snps --pheno cvd.EUR.pheno --no-psam-pheno --pheno-name XXX --1 --glm cols=+ax,+a1freq,+a1freqcc,+a1count,+a1countcc,+beta,+orbeta,+nobs hide-covar no-x-sex --covar pheno/ukb.cov --covar-name age,sex,PC1-PC10 --out chr$chr
done
上述命令顺利跑完后,确认生成的文件没有问题后,可以把所有的染色体的数据串到一起,形成一个单一的 XXX.gwas.gz 文件。鉴于2千多万个SNP,文件太大,我们一般只保留:P<0.01的SNP 以及那些在Hapmap3 里面的SNP。最终合并成的 XXX.gwas.gz 文件用 TAB 分割,CHR:POS 排好序,要不然 LocusZoom 那样的软件不能处理。也可以用 tabix -f -S 1 -s 1 -b 2 -e 2 XXX.gwas.gz 对数据进行索引,便于 LocalZoom 那样的软件去处理。
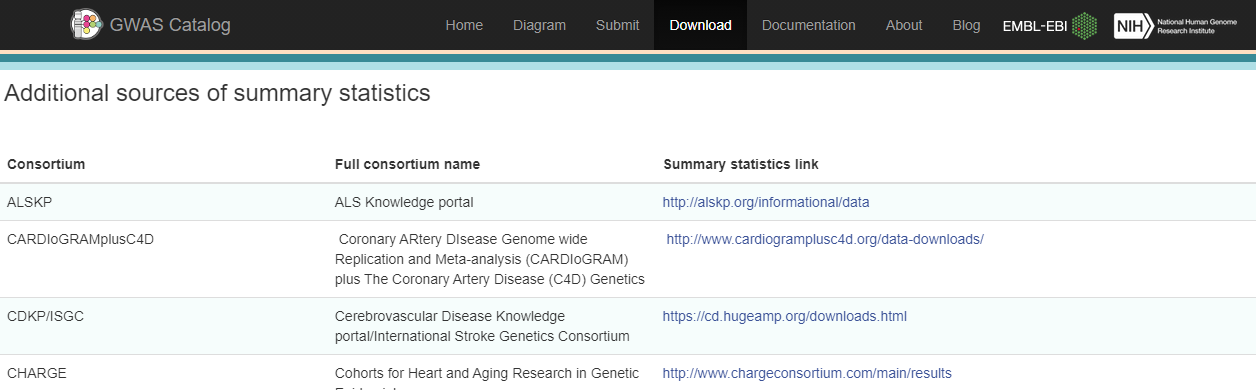
最经典的,起源于美国NIH 的 GWAS Catalog. 这个页面也罗列了一些大型GWAS数据联盟。
UKB GWAS 完整的分析结果,网上发布
- 美国哈佛大学:http://www.nealelab.is/uk-biobank
- 英国爱丁堡大学:geneatlas: http://geneatlas.roslin.ed.ac.uk
日本生物样本库的 GWAS:http://jenger.riken.jp/en/result
对于这上面的每一个表型,点击最后一列,查看曼哈顿图和QQ图,然后点击那个页面上的 Download summary statistics。否则,前面一页的 download 下来的数据没有 rsID
各大专项疾病领域的GWAS,比如:
- 哈佛大学的CVD knowlege portal: https://hugeamp.org/
- 南加州大学的神经影像基因组国际合作团队:http://enigma.ini.usc.edu/
如果不用上述的系统,可以使用 PLINK 人工操作。请点击左边菜单中的 Report postprocess 中的 3个命令(--annotate, --clump, --gene-report)
trait=MI
gunzip -c $trait.gwas.gz | sed '1 s/ POS/ BP/' > $trait.gwas.txt # 以后就不需要 sed 这一步了
plink --annotate $trait.gwas.txt NA ranges=glist-hg19 --border 10 --pfilter 5e-8 --out $trait.top
# 由于千人基因组 (g1k) 的基因数据过大(将近1亿个SNP),一般讲每一个染色体的GWAS数据分开来 clump
# plink clump 的结果,不包括那些 --bfile 里面没有的SNP,所以得要把那些SNP再添加到 clump 的结果里。
# 可惜 PLINK的作者不想让 PLINK 来直接处理这个问题,
for chr in {1..22}; do
plink1.9 --vcf g1k.chr$chr.vcf.gz --clump $trait.gwas.txt --clump-p1 5e-08 --clump-p2 5e-08 --clump-kb 1000 --clump-r2 0.2 --out $trait.chr$chr
awk '$1 !="" {print $3,$1, $4,$5}' $trait.chr$chr.clumped > $trait.chr$chr.top
done
# 通过LD的计算来找到GWAS数据里面的independent top hits,也有一些问题(比如g1k的LD不是金标准,r2也不是最合理的筛选办法),并且计算量很大。
# 如果不考虑 SNP之间的LD,只考虑距离,可以用下面这个简单的代码来寻找GWAS数据里面每1MB区间的top SNP。
# 假设GWAS的第1,2,3 列分别是 SNP, CHR, POS,最后一列是P。
zcat ABC.gwas.gz | awk 'NR==1 || $NF<5e-8 {b=sprintf("%.0f",$3/1e6); print $1,$2,$3,$NF,b}' | \
sort -k 2,2n -k 5,5n -k 4,4g | awk '{if (arr[$NF] !="Y") print $0; arr[$NF] ="Y"}'
# 要把上述得到的显著区域跟别人已经发表的 SNP进行比较,看是不是有重叠(1MB范围之内的重叠都算),可以用下面的 bedtools 命令。
# 该文件需要将 A 和 B 两个文件转换成 bed 格式,并把其中的一个文件的SNP的位置,左右各扩张1MB。如果起始位置出现负数,一定要将负数改为 0。
# 比如A文件中的某个 rsXYZ 位于1号染色体上的3000000 位置,那么bed 文件中就写: 1 2000000 2000000 rsXYZ
bedtools intersect -a A.bed -b B.bed -wo
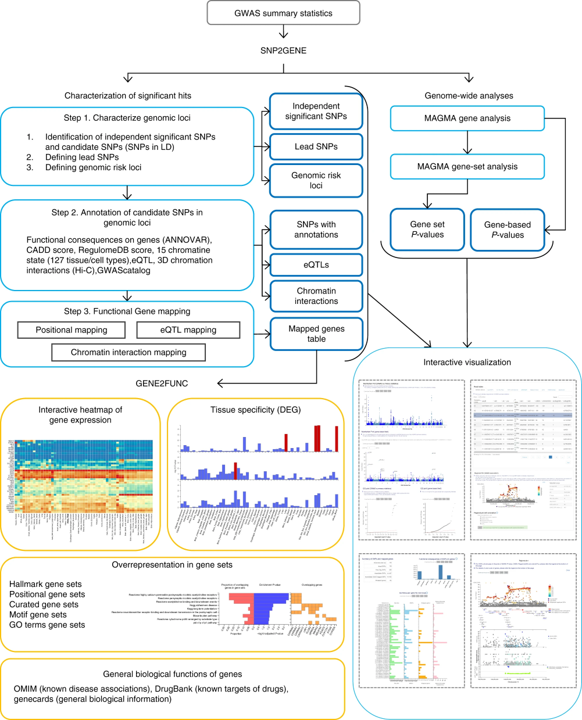
引用过千的FUMA 网上解读系统
本组开发的 PAGEANT 大力推荐!
- 如果有简单的数据,别人文章里面已经报道了的 exposure 和 outcome 的 BETA 和 SE,最简单的是使用 MendelianRandomization R包。还有一个特别针对 UKB 处理海量数据的 TwoSampleMR R包。
- 我喜欢 MendelianRandomizaiton 的简单透明。我们只需要先把两个数据运行一下 compare-B, 然后得到一个只有4列的小文件 (BETA1, SE1, BETA2, SE2),手机上都能看见文件的全部。然后两三行命令,就可以跑 MR并画图。TwoSampleMR 自然是不错,连数据都不需要了,只需要写一个 MRC-IEU的代码,远程的数据就用起来了。一个简单的 extract_instruments() 和 harmonise_data() 就把所有的QC 事情搞定了,连一个clump_data()这样的命令都不用写了。再也不用compare-B了,反正 harmonise_data()自会处理好各种问题。我们连 clump 的reference genome 都不知道在哪,就跑一个 clump_data() 或 extract_instruments(clump=T) 就可以了。
- 但是,除了MS office 和 GPS导航这样的软件,全世界都在用,都在测试,都在有大团队fix bug之外,别人写的大而全的东西,有大而全的问题。有些时候,我们并不知道那些命令真的包括了什么,可能会被错误的GPS导航带到沟里去,后者让我们完全不再记路了,指望GSP把我们直接导航到别人的客厅里。所以,就算我们用强大的 TwoSampleMR,得到什么有趣的分析结果的话,也需要用比较手工的方法再重复一下。
- 别人的数据,都不需要下载就能用,自然是好。但是试想一下,哪天上不了那个网,或者对方将数据大量更新修改,我们的结果就再也重复不了了。
基因注释信息浏览器:
dbSNP: https://www.ncbi.nlm.nih.gov/snp/
UCSC genome browser: https://www.genome.ucsc.edu/
美国精准医学:https://databrowser.researchallofus.org/ TopMed browser: https://bravo.sph.umich.edu/
Gnomad browser: https://gnomad.broadinstitute.org/
GlobalBiobankEngine 和 nanopore 数据分析等:https://github.com/rivas-lab
GWAS 入门介绍
2008. JAMA. How to interpret a genome-wide association study (pubmed.ncbi.nlm.nih.gov/18349094/)
2020. NEJM. Genomewide Association Study of Severe Covid-19 with Respiratory Failure (https://www.nejm.org/doi/full/10.1056/NEJMoa2020283)
2020. MolPsy. Genomic analysis of diet composition finds novel loci and associations with health and lifestyle (https://www.nature.com/articles/s41380-020-0697-5)
2021. Nature. Mapping the human genetic architecture of COVID-19
芬兰赫尔辛基大学 GWAS 课程:https://www.mv.helsinki.fi/home/mjxpirin/GWAS_course/
Mendelian Randomization 入门介绍
2012. Plasma HDL cholesterol and risk of myocardial infarction: a mendelian randomisation study (pubmed.ncbi.nlm.nih.gov/22607825/)
2017. Statistical methods to detect pleiotropy in human complex traits (pubmed.ncbi.nlm.nih.gov/29093210/)
2019. Meta-analysis and Mendelian randomization: A review (pubmed.ncbi.nlm.nih.gov/30861319/)
2022. Assessing the Causal Role of Sleep Traits on Glycated Hemoglobin: A Mendelian Randomization Study (pubmed.ncbi.nlm.nih.gov/35349659/)
2022. Genetically predicted sex hormone levels and health outcomes: phenome-wide Mendelian randomization investigation (pubmed.ncbi.nlm.nih.gov/35218343/)