数据来源于:http://www.cbioportal.org/data_sets.jsp
下载属于METABRIC的数据集:Breast Cancer (METABRIC, Nature 2012 & Nat Commun 2016)
也可以使用cbioportal的在线浏览器来了解基本项目信息:http://www.cbioportal.org/study?id=brca_metabric#summary
小文件如下:
─ [ 646] brca_metabric
│ ├── [ 410] LICENSE
│ ├── [ 136] case_lists
│ │ ├── [ 20K] cases_nat_comm_2016.txt
│ │ └── [ 16K] cases_nature_2012.txt
│ ├── [265K] data_clinical_patient.txt
│ ├── [102K] data_clinical_sample.txt
│ ├── [116K] data_gene_matrix.txt
│ ├── [1.2K] data_gene_panel.txt
│ ├── [3.8M] data_mutations_extended.txt
│ ├── [ 512] meta_CNA.txt
│ ├── [ 143] meta_clinical_patient.txt
│ ├── [ 141] meta_clinical_sample.txt
│ ├── [ 322] meta_expression.txt
│ ├── [ 146] meta_gene_matrix.txt
│ ├── [ 292] meta_mutations_extended.txt
│ ├── [ 380] meta_study.txt
│ ├── [ 55K] validation_error_file.txt
│ └── [ 42K] validation_report.html
里面值得注意的就是那个 3.8M的文件: data_mutations_extended.txt
还有一个 100M的 文件是 data_CNA.txt 和一个 500M的文件 data_expression.txt
主要是是删除本地文件后提交到GitHub上面。
rm tmp.pdf
git add -A
git commit -a -m 'rm tmp'
git push -u origin master
当然,每次修改自己本地文件,都需要同步更新到GitHub啦。
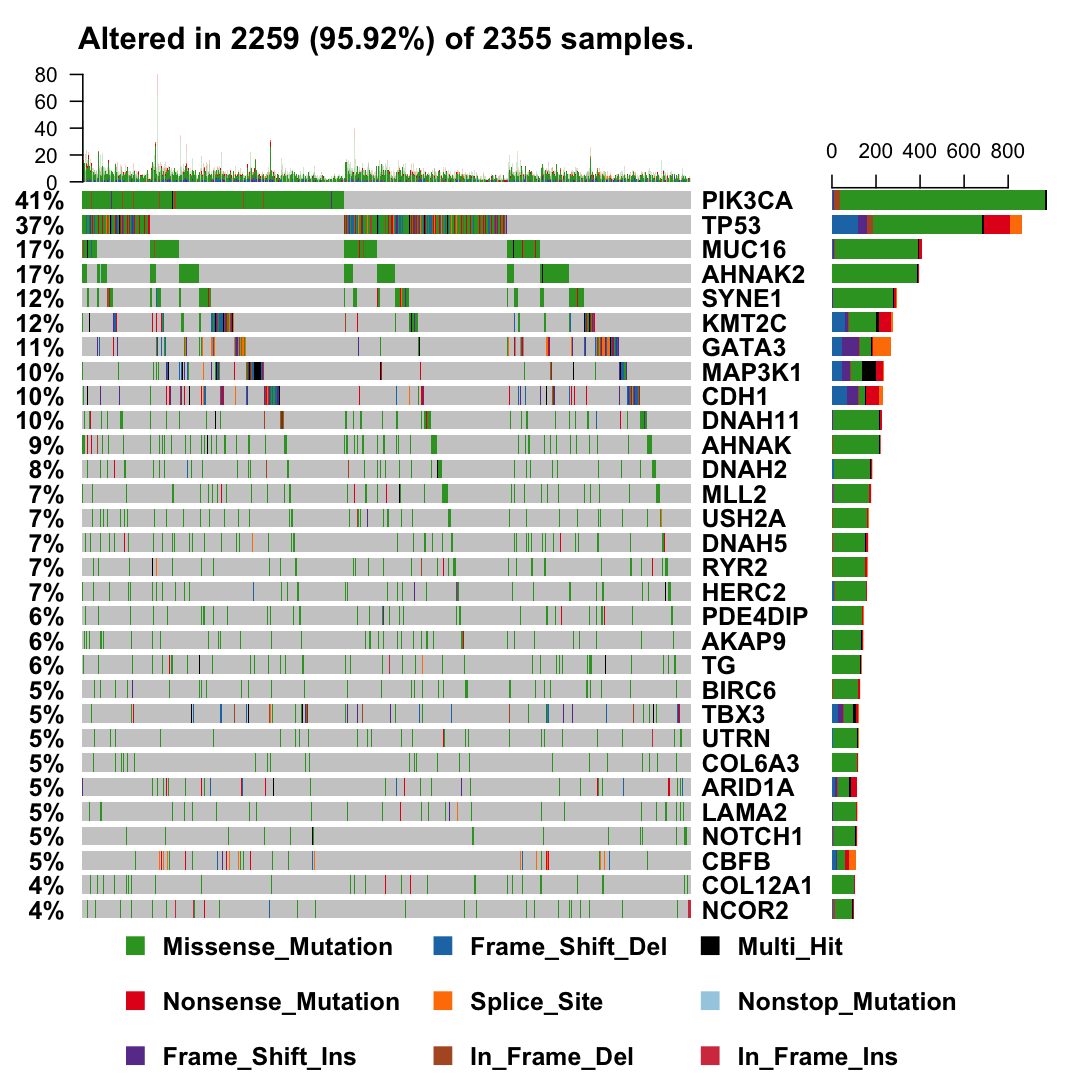
整理文章的突变信息,然后使用maftools即可出图,如下:
可以看到,突变频率比较高的基因是: PIK3CA,TP53,MUC16,AHNAK2,SYNE1,KMT2C,GATA3,MAP3K1,CDH1 , 接下来就可以对这些基因进行针对性分析。
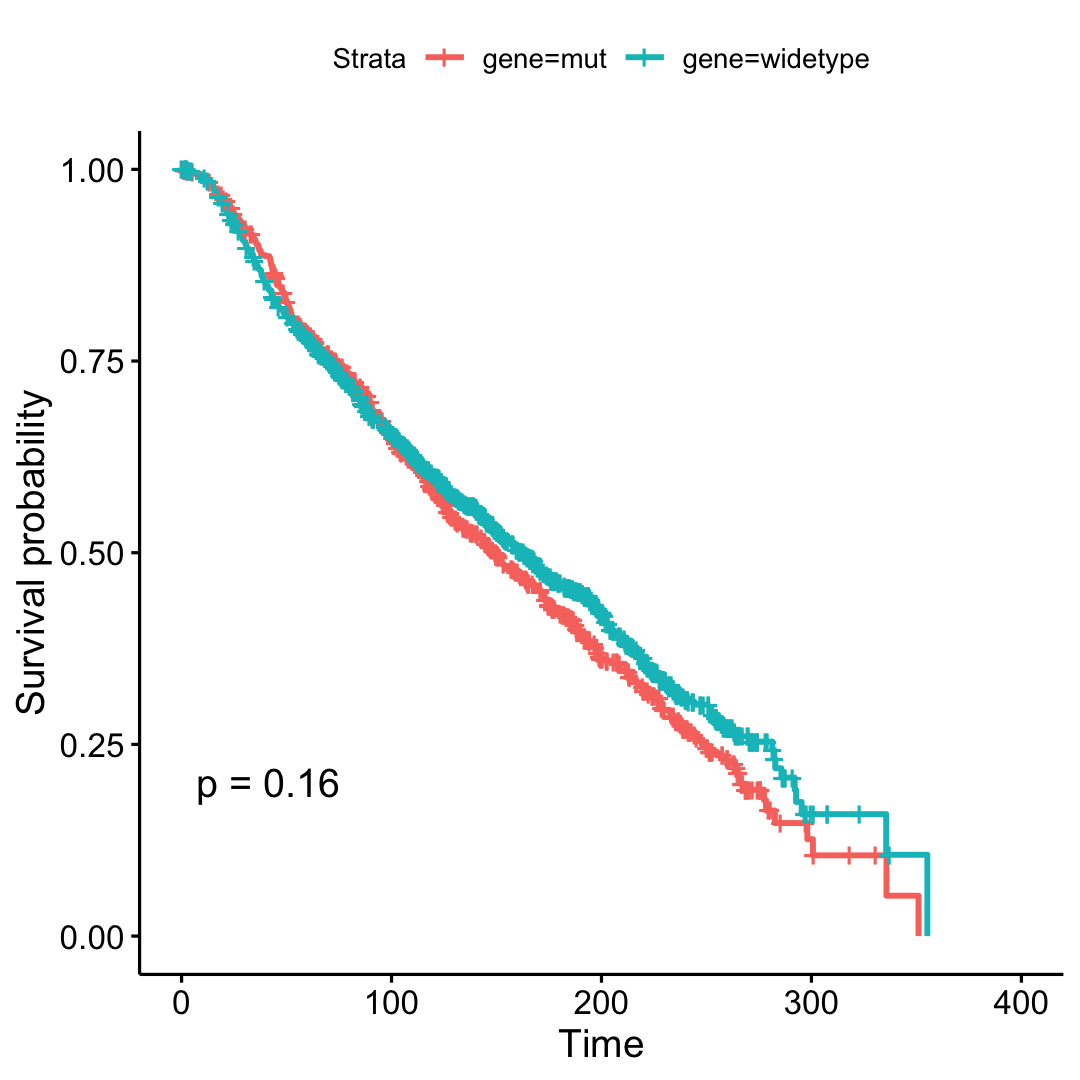
取突变频率比较高的基因做生存分析,发现PIK3CA的确是突变频率非常高,但是与生存无关,如下所示:
然后就查阅文献发现有人研究了这一点:
发表于 Breast Cancer Research 2017文章是:PIK3CA mutations are common in lobular carcinoma in situ, but are not a biomarker of progression
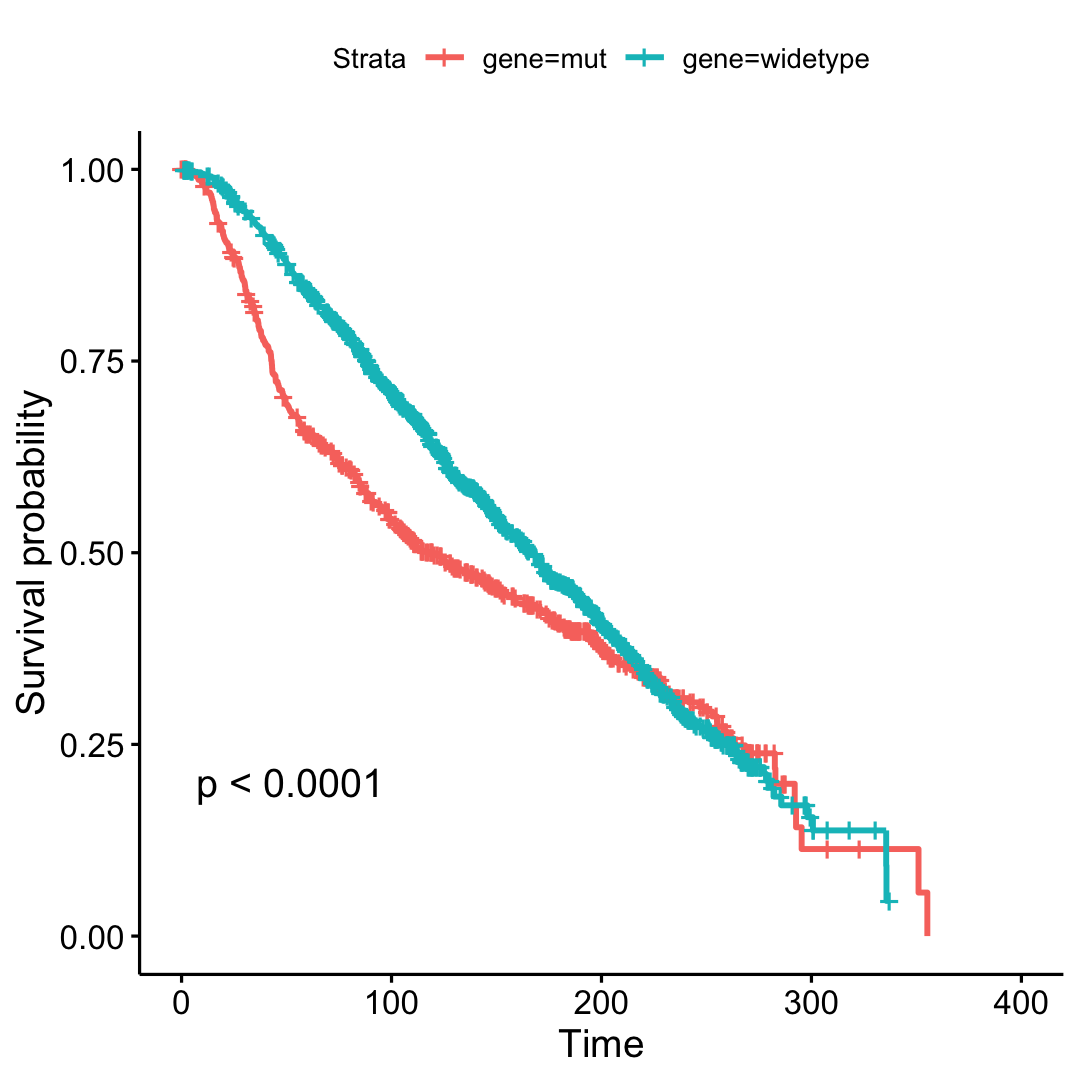
当然,就会有突变能显著影响生存的,比如:
实际上,我推测啊,这些突变影响生存,应该是要根据亚型区分后再重现技术,比如TNBC本身就比其它亚型生存差,而且TNBC里面的TP53突变比例非常高,就会两个因素混在一起,到底是TP53影响生存还是亚型影响着生存。
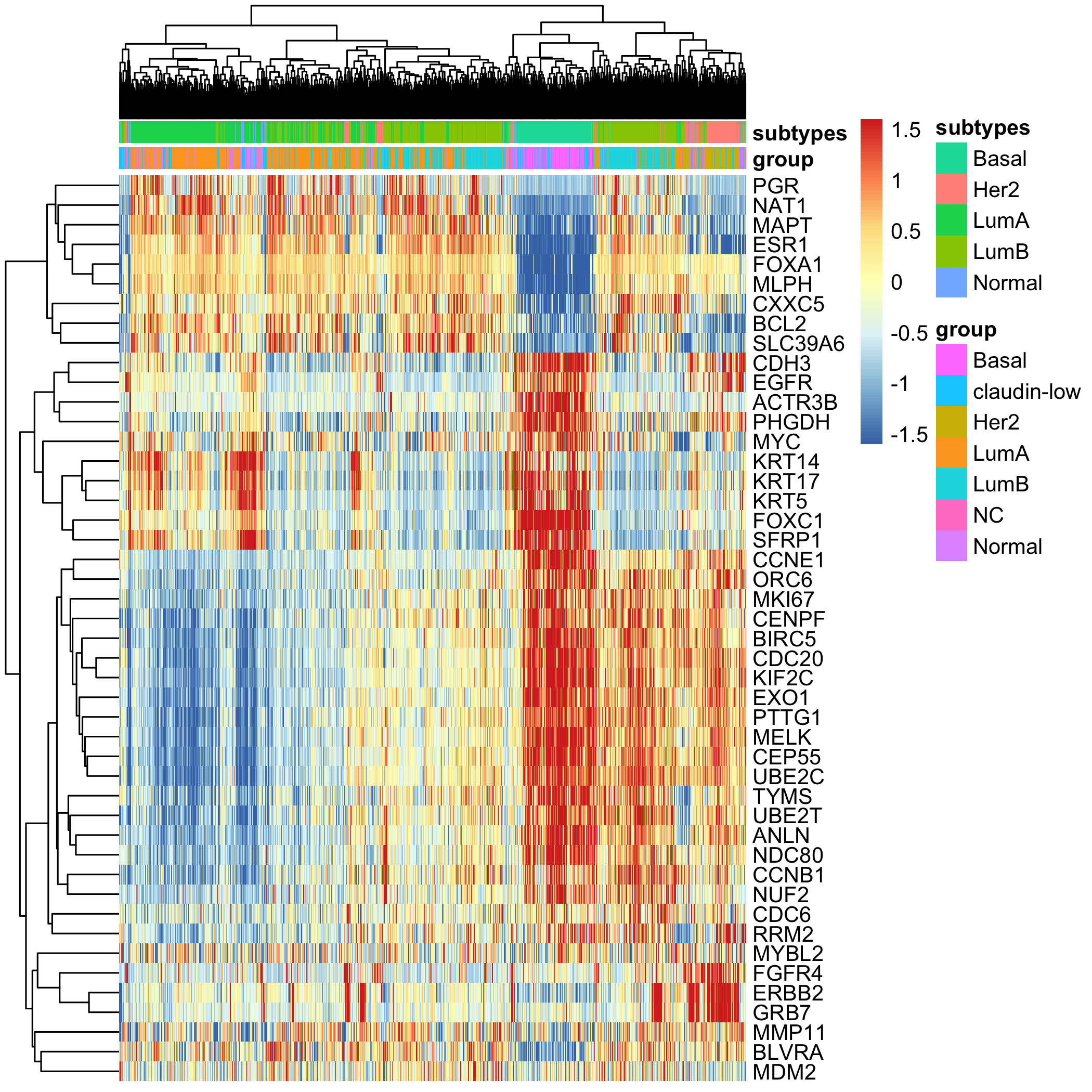
可以看到我们使用genefu算法对METABRIC的表达矩阵进行分类,和METABRIC自己记录的病人临床信息的一致性还蛮好的
如果把PAM50分类的那50个基因挑选出来,进行表达量的热图可视化,如下:
本研究仅仅是抛砖引玉,让大家熟悉这个乳腺癌领域多组学大队列,并不做细致探索,也没有科学假设,所以点到为止咯。
初期代码都在 scripts 文件夹, 感谢关注。
step0-install_packages.R
step1-check_clinical.R
step2-check-expression.R
step3-check-CNA.R
step4-check-mutations.R
step5-mutation-signatures.R
step6-maftools.R
step7-batch-survival.R
step8-pam50.R