Juan Emilio Zurita Macías 30 de May, 2022
- 1 Descripción del dataset.
- 2 Integración y selección de los datos de interés a analizar.
- 3 Limpieza de los datos.
- 4 Análisis de los datos
- 5 Resolución del problema.
if (!require('reshape')) install.packages('reshape')
if (!require('ggplot2')) install.packages('ggplot2')
if (!require('dplyr')) install.packages('dplyr')
if (!require('tidyverse')) install.packages('tidyverse')
if (!require('ggpubr')) install.packages('ggpubr')
if (!require('corrplot')) install.packages('corrplot')
if (!require('RColorBrewer')) install.packages('RColorBrewer')
if (!require('ResourceSelection')) install.packages('ResourceSelection')
if (!require('pROC')) install.packages('pROC')El dataset está relacionado a la variante “Vinho Verde” portugués y
contiene una serie de componentes fisioquimicos que pueden ser tratados
como variables de entrada además de una variable quality que define
con número entero del 0 al 10 la calidad de cada observación.
| Variable | Descripción | Unidades |
|---|---|---|
fixed.acidity (acidez fija) |
refiere al conjunto de los ácidos naturales procedentes de la uva (tartárico, málico, cítrico y succínico) o formados en la fermentación maloláctica (láctico) | g(tartaric acid)/dm3 |
volatile.acidity (acidez volátil) |
refiere al conjunto de ácidos formados durante la fermentación o como consecuencia de alteraciones microbianas. | g(tartaric acid)/dm3 |
citric.acid (ácido cítrico) |
es un acidificante para corregir la acidez y además posee una acción estabilizante | g/dm3 |
residual.sugar (azúcar residual) |
es la cantidad total de azúcar que queda en el vino que no ha sido fermentada por las levaduras | g/dm3 |
chlorides (clorurlos) |
son aniones derivados del cloruro de hidrógeno | g(tartaric acid)/dm3 |
free.sulfur.dioxide (dióxido de azufre libre) |
se utiliza en enología principalmente como conservante, pero también para otros fines (por ejemplo, para funciones antisépticas, antioxidantes, antioxidasicas, solubilizantes, combinadas y clarificantes) | mg/dm3 |
total.sulfur.dioxide (dióxido de azufre total) |
se utiliza en enología principalmente como conservante, pero también para otros fines (por ejemplo, para funciones antisépticas, antioxidantes, antioxidasicas, solubilizantes, combinadas y clarificantes) | mg/dm3 |
density (densidad) |
es una magnitud escalar referida a la cantidad de masa en un determinado volumen de una sustancia o un objeto sólido | g/cm3 |
pH |
es una medida de acidez o alcalinidad de una disolución acuosa | - |
sulphates (sulfitos) |
se encargan de neutralizar las levaduras propias de la viña, así como bacterias acéticas y lácticas que pueden provocar que el vino se avinagre | g(tartaric acid)/dm3 |
alcohol |
Compuesto de carbono, hidrógeno y oxígeno que deriva de los hidrocarburos y lleva en su molécula uno o varios hidroxilos (OH) | % vol. |
quality (calidad) |
- | - |
¿Por qué es importante y qué pregunta/problema pretende responder?
Este conjunto de datos pretende responder preguntas tales como, que componentes fisioquímicos afectan en mayor medida a la calidad del vino y con ellos incluso llegar a construir un modelo que permita predecir la calidad de un vino en función de éstos.
Puede ser el resultado de adicionar diferentes datasets o una subselección útil de los datos originales, en base al objetivo que se quiera conseguir.
Procedemos a la lectura de los datos que se encuentran en formato CSV
winequality <- read.csv("winequality-red.csv")Comprobamos que tipo de datos tiene y las primeras entradas del dataset
str(winequality)## 'data.frame': 1599 obs. of 12 variables:
## $ fixed.acidity : num 7.4 7.8 7.8 11.2 7.4 7.4 7.9 7.3 7.8 7.5 ...
## $ volatile.acidity : num 0.7 0.88 0.76 0.28 0.7 0.66 0.6 0.65 0.58 0.5 ...
## $ citric.acid : num 0 0 0.04 0.56 0 0 0.06 0 0.02 0.36 ...
## $ residual.sugar : num 1.9 2.6 2.3 1.9 1.9 1.8 1.6 1.2 2 6.1 ...
## $ chlorides : num 0.076 0.098 0.092 0.075 0.076 0.075 0.069 0.065 0.073 0.071 ...
## $ free.sulfur.dioxide : num 11 25 15 17 11 13 15 15 9 17 ...
## $ total.sulfur.dioxide: num 34 67 54 60 34 40 59 21 18 102 ...
## $ density : num 0.998 0.997 0.997 0.998 0.998 ...
## $ pH : num 3.51 3.2 3.26 3.16 3.51 3.51 3.3 3.39 3.36 3.35 ...
## $ sulphates : num 0.56 0.68 0.65 0.58 0.56 0.56 0.46 0.47 0.57 0.8 ...
## $ alcohol : num 9.4 9.8 9.8 9.8 9.4 9.4 9.4 10 9.5 10.5 ...
## $ quality : int 5 5 5 6 5 5 5 7 7 5 ...
Realizamos un summary para extraer estadísticos de cada variable
summary(winequality)## fixed.acidity volatile.acidity citric.acid residual.sugar
## Min. : 4.60 Min. :0.1200 Min. :0.000 Min. : 0.900
## 1st Qu.: 7.10 1st Qu.:0.3900 1st Qu.:0.090 1st Qu.: 1.900
## Median : 7.90 Median :0.5200 Median :0.260 Median : 2.200
## Mean : 8.32 Mean :0.5278 Mean :0.271 Mean : 2.539
## 3rd Qu.: 9.20 3rd Qu.:0.6400 3rd Qu.:0.420 3rd Qu.: 2.600
## Max. :15.90 Max. :1.5800 Max. :1.000 Max. :15.500
## chlorides free.sulfur.dioxide total.sulfur.dioxide density
## Min. :0.01200 Min. : 1.00 Min. : 6.00 Min. :0.9901
## 1st Qu.:0.07000 1st Qu.: 7.00 1st Qu.: 22.00 1st Qu.:0.9956
## Median :0.07900 Median :14.00 Median : 38.00 Median :0.9968
## Mean :0.08747 Mean :15.87 Mean : 46.47 Mean :0.9967
## 3rd Qu.:0.09000 3rd Qu.:21.00 3rd Qu.: 62.00 3rd Qu.:0.9978
## Max. :0.61100 Max. :72.00 Max. :289.00 Max. :1.0037
## pH sulphates alcohol quality
## Min. :2.740 Min. :0.3300 Min. : 8.40 Min. :3.000
## 1st Qu.:3.210 1st Qu.:0.5500 1st Qu.: 9.50 1st Qu.:5.000
## Median :3.310 Median :0.6200 Median :10.20 Median :6.000
## Mean :3.311 Mean :0.6581 Mean :10.42 Mean :5.636
## 3rd Qu.:3.400 3rd Qu.:0.7300 3rd Qu.:11.10 3rd Qu.:6.000
## Max. :4.010 Max. :2.0000 Max. :14.90 Max. :8.000
Como no sabemos que elementos son más relevantes, no vamos a descartar ninguna variable, por lo que podemos proceder a su limpieza con el dataset completo.
No parece haber elementos vacíos en este conjunto de datos, y solo hay una única variable que contiene valores cero (ácido cítrico) y parece corresponder con un valor válido.
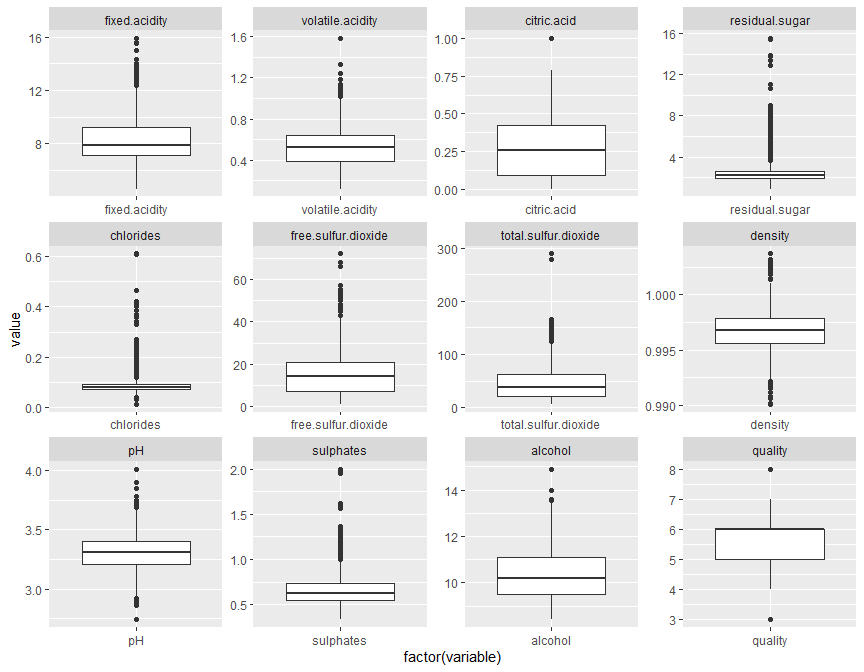
Para comprobar la posible presencia de outliers, representamos el mediante boxplots la distribución de valores de cada variable.
library(reshape)
library(ggplot2)
ggplot(melt(winequality), aes(factor(variable), value)) +
geom_boxplot() + facet_wrap(~variable, scale="free")Se puede observar una gran presencia de outliers en variables como
total.sulfur.dioxide, chlorides o sulphates. Se procederá a mirar
de cerca cada uno de ellos y descartar valores que estén muy por encima
de su mediana.
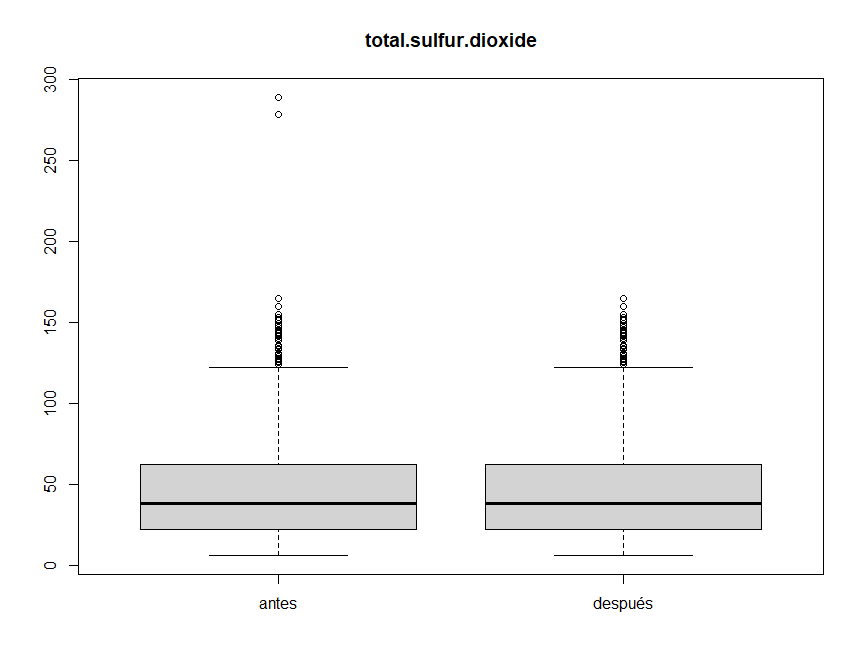
winequality.clean <- winequalityEn el caso de total.sulfur.dioxide, podemos considerar outliers ese
par de valores que sobresalen por encima de 200.
winequality.clean$total.sulfur.dioxide[winequality$total.sulfur.dioxide > 200] <- NA
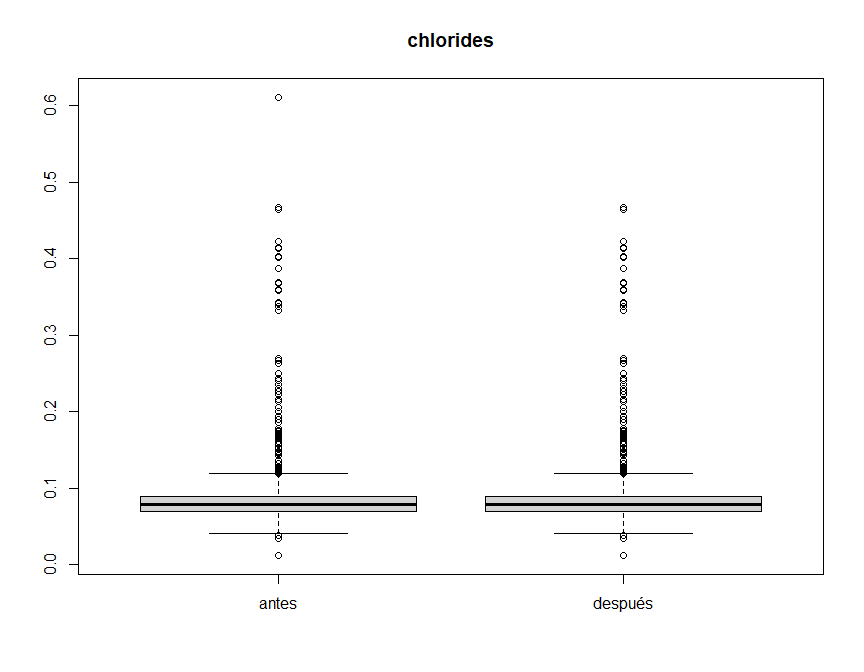
boxplot(winequality$total.sulfur.dioxide, winequality.clean$total.sulfur.dioxide, main="total.sulfur.dioxide", names = c("antes", "después"))En el caso de chlorides, podemos considerar outliers ese valor que
sobresale por encima de 0.5.
winequality.clean$chlorides[winequality$chlorides > 0.5] <- NA
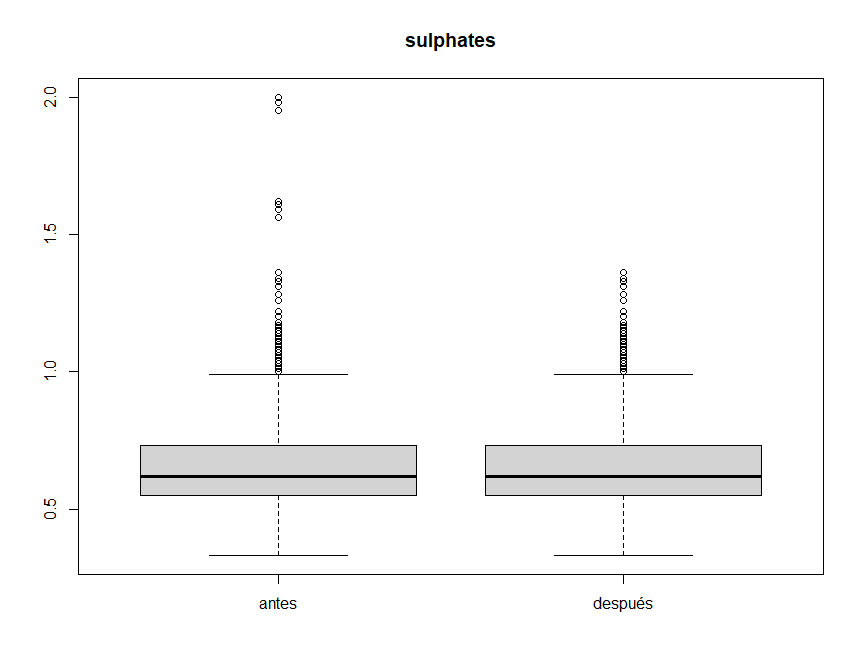
boxplot(winequality$chlorides, winequality.clean$chlorides, main="chlorides", names = c("antes", "después"))En el caso de sulphates, podemos considerar outliers ese par de grupos
de valores que sobresalen por encima de 1.5.
winequality.clean$sulphates[winequality$sulphates > 1.5] <- NA
boxplot(winequality$sulphates, winequality.clean$sulphates, main="sulphates", names = c("antes", "después"))library(dplyr)
library(tidyverse)
winequality.clean <- winequality.clean %>%
drop_na() %>%
unique()Comprobamos cuanto se ha reducido el dataset después de realizar la limpieza
nrow(winequality.clean)/nrow(winequality) * 100## [1] 84.36523
Se han descartado en torno al 16% de las observaciones del dataset original tras eliminar valores duplicados o outliers.
summary(winequality.clean)## fixed.acidity volatile.acidity citric.acid residual.sugar
## Min. : 4.600 Min. :0.1200 Min. :0.0000 Min. : 0.900
## 1st Qu.: 7.100 1st Qu.:0.3900 1st Qu.:0.0900 1st Qu.: 1.900
## Median : 7.900 Median :0.5200 Median :0.2600 Median : 2.200
## Mean : 8.312 Mean :0.5299 Mean :0.2706 Mean : 2.517
## 3rd Qu.: 9.200 3rd Qu.:0.6400 3rd Qu.:0.4300 3rd Qu.: 2.600
## Max. :15.900 Max. :1.5800 Max. :0.7900 Max. :15.500
## chlorides free.sulfur.dioxide total.sulfur.dioxide density
## Min. :0.01200 Min. : 1.00 Min. : 6.00 Min. :0.9901
## 1st Qu.:0.07000 1st Qu.: 7.00 1st Qu.: 22.00 1st Qu.:0.9956
## Median :0.07900 Median :14.00 Median : 38.00 Median :0.9967
## Mean :0.08699 Mean :15.83 Mean : 46.25 Mean :0.9967
## 3rd Qu.:0.09000 3rd Qu.:21.00 3rd Qu.: 62.00 3rd Qu.:0.9978
## Max. :0.46700 Max. :72.00 Max. :165.00 Max. :1.0037
## pH sulphates alcohol quality
## Min. :2.860 Min. :0.3300 Min. : 8.40 Min. :3.000
## 1st Qu.:3.210 1st Qu.:0.5500 1st Qu.: 9.50 1st Qu.:5.000
## Median :3.310 Median :0.6200 Median :10.20 Median :6.000
## Mean :3.312 Mean :0.6528 Mean :10.44 Mean :5.624
## 3rd Qu.:3.400 3rd Qu.:0.7200 3rd Qu.:11.10 3rd Qu.:6.000
## Max. :4.010 Max. :1.3600 Max. :14.90 Max. :8.000
Por ejemplo, si se van a comparar grupos de datos, ¿cuáles son estos grupos y qué tipo de análisis se van a aplicar?.
Para nuestro análisis, vamos a crear una nueva variable binaria
basándonos en la variable quality. Esta variable determinará si se
trata de un buen vino (bajo el criterio de una nota de corte mayor o
igual a 6) o de un vino mediocre.
winequality.clean$buen.vino <- ifelse(winequality.clean$quality >= 6, TRUE, FALSE)Como en este punto aún no tenemos claro que variables vamos a analizar (dependerá de análisis posteriores como la correlación), no vamos a realizar ninguna selección de momento.
Si consideramos la aplicación del teorema del límite central (TLC), podemos concluir que para muestras suficientemente grandes de la población, aunque la población original no siga una distribución normal, la media se aproxima a una distribución normal. Esto será útil por si queremos hacer algún contraste de medias en nuestro análisis.
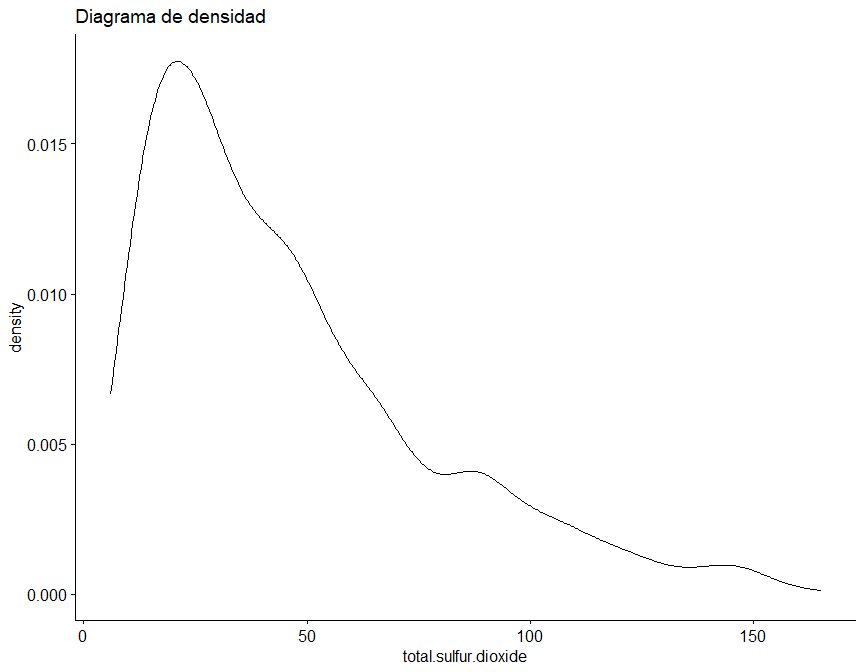
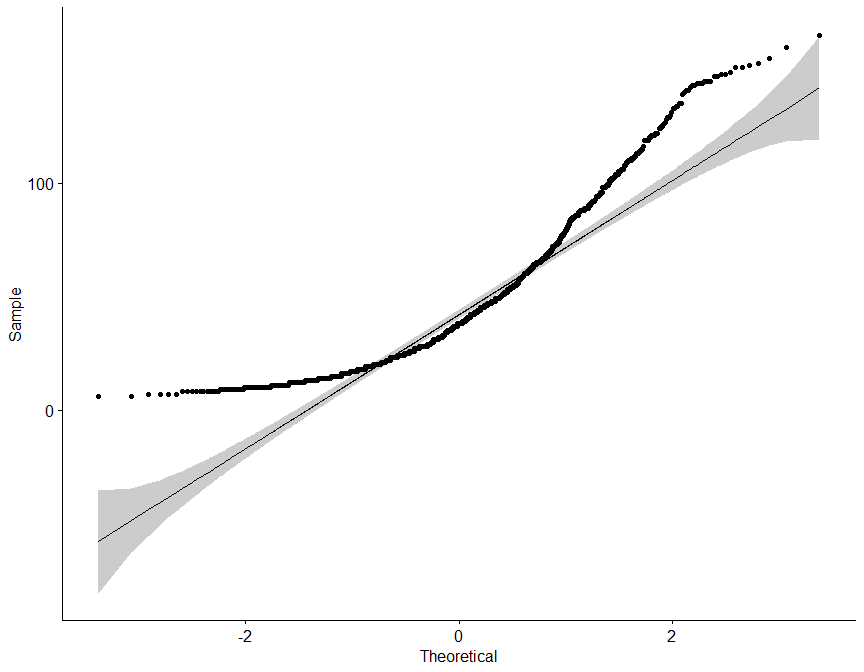
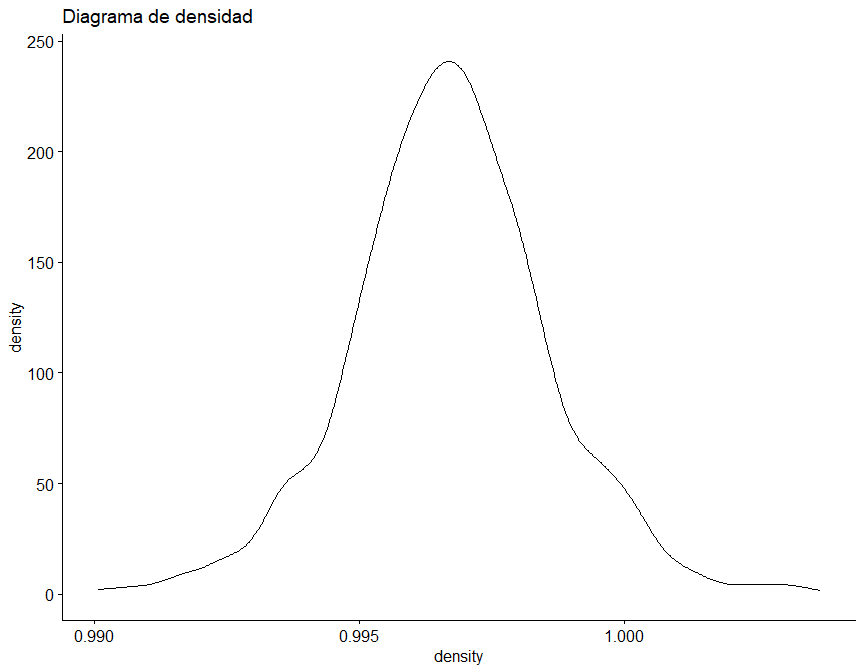
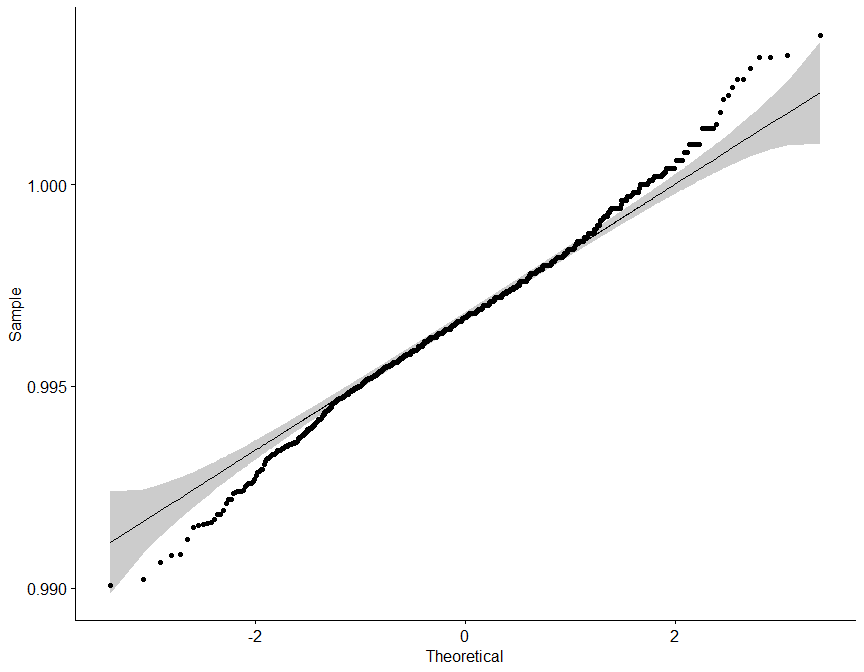
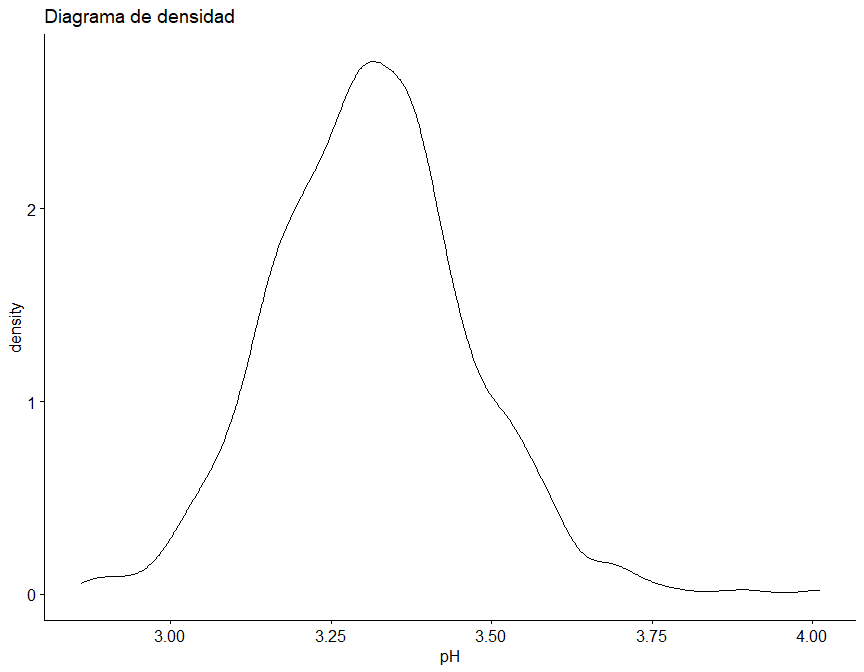
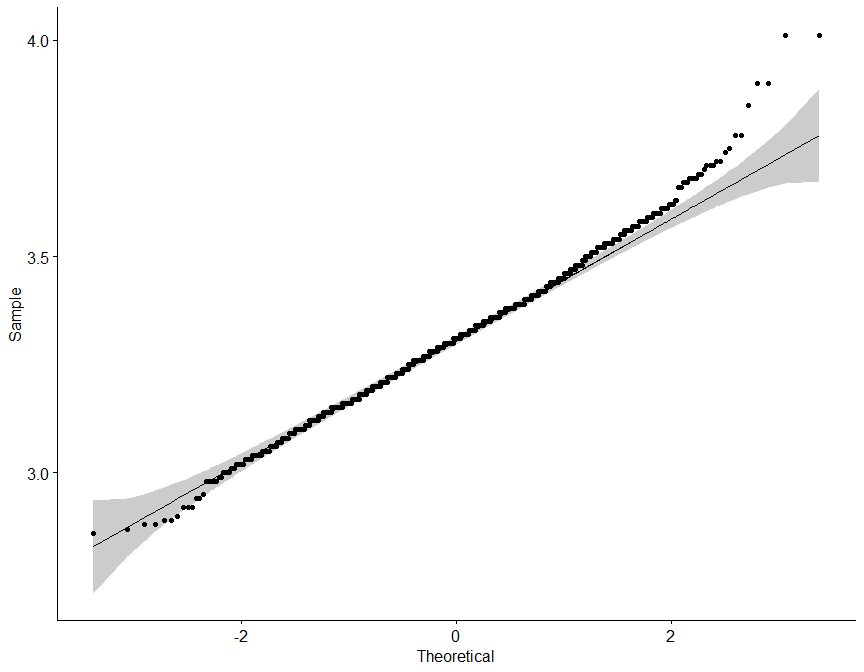
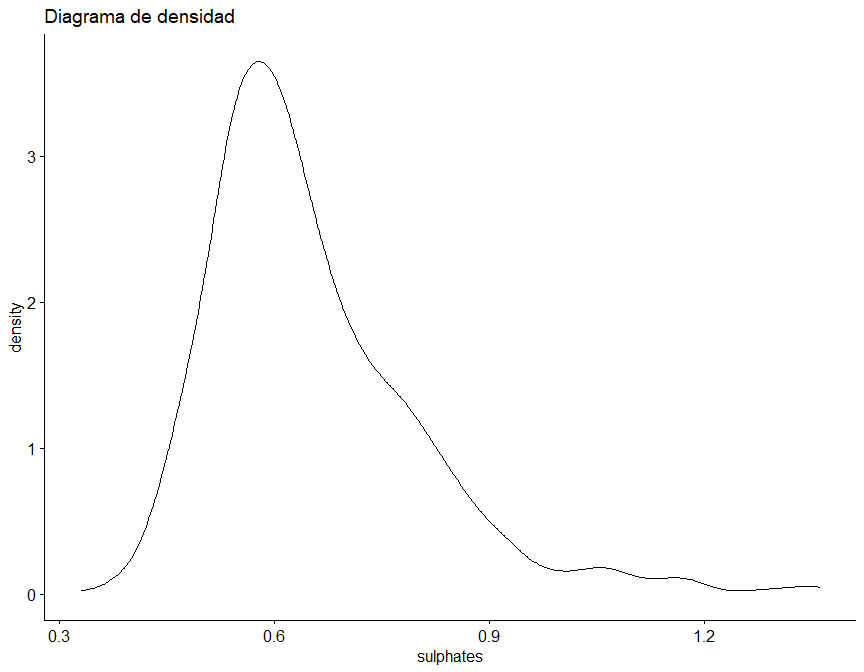
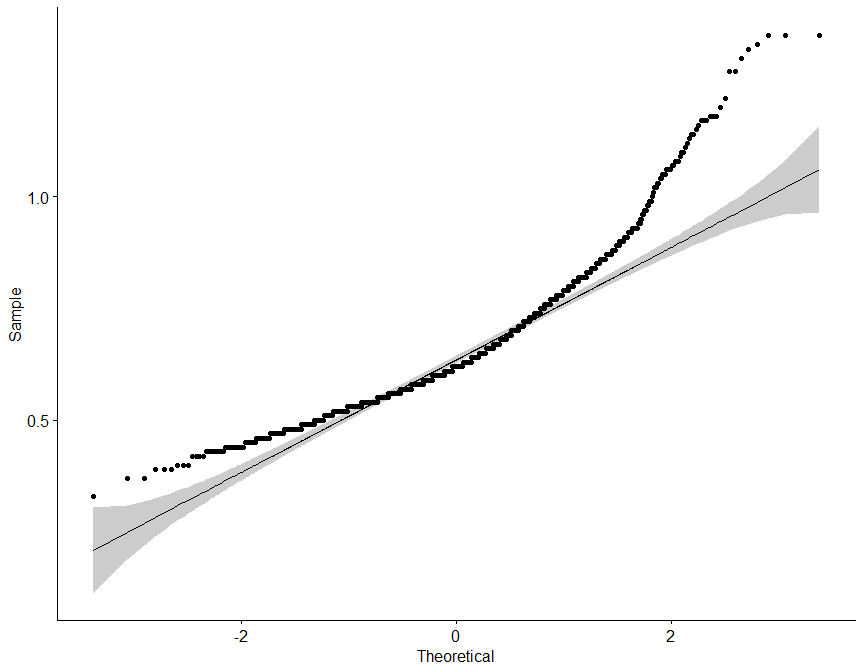
Sin embargo, se ha diseñado la siguiente función, que para un dataframe dado, realiza tanto un diagrama de densidad como Q-Q, además de el test de Saphiro-Wilk para una submuestra aleatoria de 50 elementos de cada variable.
library(ggpubr)
test.normalidad <- function(dataframe, NC){
alpha <- 1 - NC
for(var in colnames(dataframe)) {
if(is.numeric(dataframe[, var])) {
# Diagrama de densidad
print(ggdensity(dataframe[, var],
main = "Diagrama de densidad",
xlab = var))
# Diagrama Q-Q
print(ggqqplot(dataframe[, var]))
# Test de Saphiro-Wilk
set.seed(1)
pvalue <- shapiro.test(sample(dataframe[, var], 50))$p.value
if(pvalue > alpha){
print(sprintf("Según el test de Saphiro-Wilk como el valor p (%s) es mayor a alfa (%s) no se rechaza la hipótesis nula (H0), por lo tanto, la variable %s presenta un comportamiento normal o paramétrico.", round(pvalue, 4), alpha, var))
}
else{
print(sprintf("Según el test de Saphiro-Wilk como el valor p (%s) es menor a alfa (%s) se rechaza la hipótesis nula (H0), por lo tanto, la variable %s presenta un comportamiento NO normal.", round(pvalue, 4), alpha, var))
}
}
else{
message(sprintf("%s - no es numérica.", var))
}
}
}test.normalidad(dataframe = winequality.clean, NC = 0.95)## [1] "Según el test de Saphiro-Wilk como el valor p (0.0038) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable fixed.acidity presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0.2425) es mayor a alfa (0.05) no se rechaza la hipótesis nula (H0), por lo tanto, la variable volatile.acidity presenta un comportamiento normal o paramétrico."
## [1] "Según el test de Saphiro-Wilk como el valor p (0.0058) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable citric.acid presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable residual.sugar presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable chlorides presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0.0019) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable free.sulfur.dioxide presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable total.sulfur.dioxide presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0.7618) es mayor a alfa (0.05) no se rechaza la hipótesis nula (H0), por lo tanto, la variable density presenta un comportamiento normal o paramétrico."
## [1] "Según el test de Saphiro-Wilk como el valor p (0.7853) es mayor a alfa (0.05) no se rechaza la hipótesis nula (H0), por lo tanto, la variable pH presenta un comportamiento normal o paramétrico."
## [1] "Según el test de Saphiro-Wilk como el valor p (1e-04) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable sulphates presenta un comportamiento NO normal."
## [1] "Según el test de Saphiro-Wilk como el valor p (0.07) es mayor a alfa (0.05) no se rechaza la hipótesis nula (H0), por lo tanto, la variable alcohol presenta un comportamiento normal o paramétrico."
## [1] "Según el test de Saphiro-Wilk como el valor p (0) es menor a alfa (0.05) se rechaza la hipótesis nula (H0), por lo tanto, la variable quality presenta un comportamiento NO normal."
## buen.vino - no es numérica.
El cálculo de homogeneidad de varianzas se realizará en el apartado correspondiente al análisis de contraste de hipótesis.
En función de los datos y el objetivo del estudio, aplicar pruebas de contraste de hipótesis, correlaciones, regresiones, etc. Aplicar al menos tres métodos de análisis diferentes.
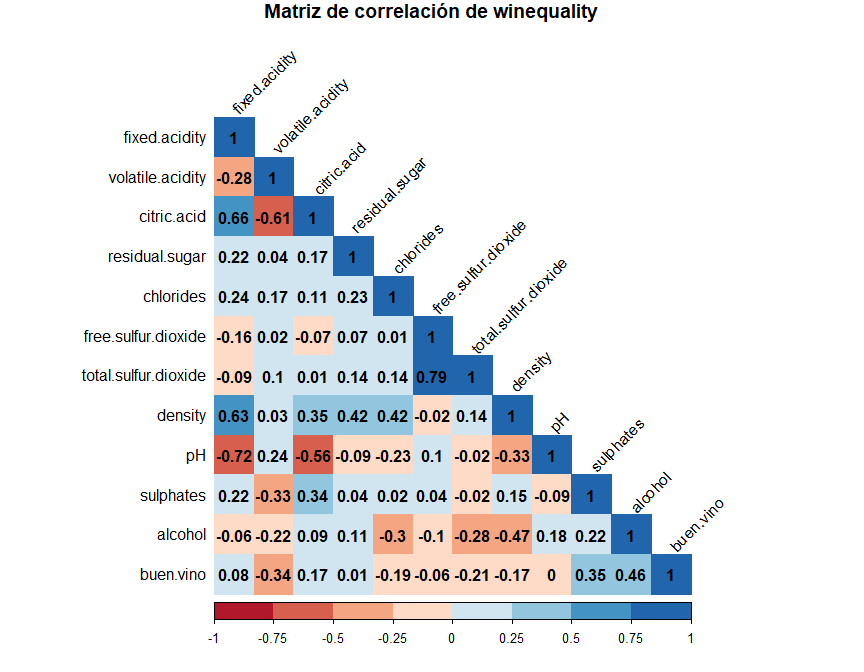
Para responder a la primera pregunta de nuestro análisis, vamos a hacer uso de la correlación por el método de Spearman
library(corrplot)
library(RColorBrewer)
corrplot(cor(winequality.clean[,-12], method = "spearman"),
title = "Matriz de correlación de winequality", mar = c(0,0,1,0),
method = "color", addCoef.col = "black",
tl.srt = 45, tl.col = "black",
col = brewer.pal(n = 8, name = "RdBu"),
type = "lower")Como el objetivo de este análisis es obtener los elementos que más
influyen en la calidad de un vino, vamos a extraer el vector de
correlación de la variable buen.vino y vamos a representar
gráficamente de forma ordenada sus valores absolutos.
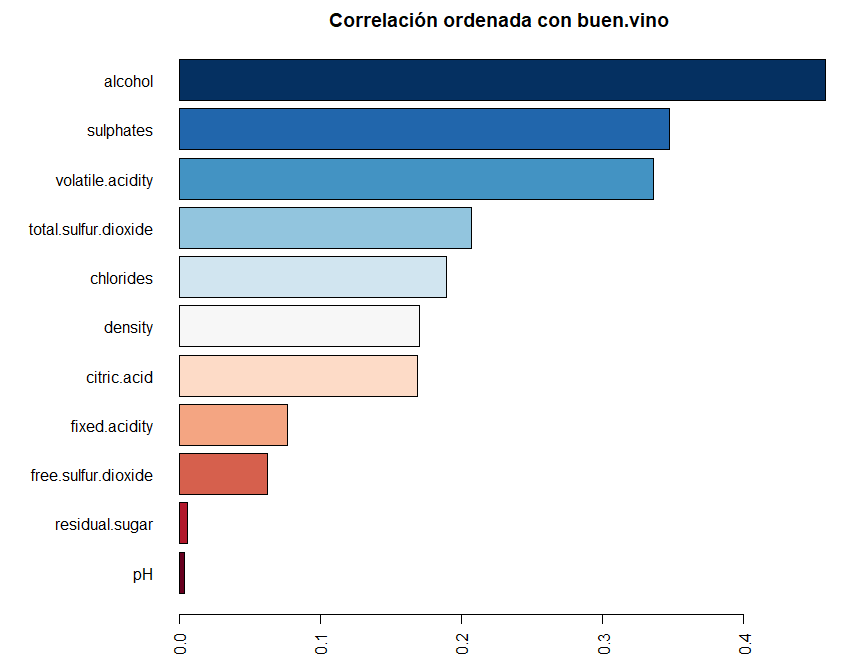
corr.buen.vino <- cor(winequality.clean[,-12], method = "spearman")[,"buen.vino"][1:11]
par(mar = c(3, 9, 2, 2))
barplot(sort(abs(corr.buen.vino)),
main = "Correlación ordenada con buen.vino",
horiz = TRUE,
las = 2,
col = brewer.pal(n = 11, name = "RdBu")
)De esta forma podemos ver claramente que los tres elementos que más
influyen en la calidad del vino son el alcohol, sulphates y
volatile.acidity.
4.3.2 ¿Es la media de alcohol de un buen vino μ1 superior a la media de alcohol de un vino mediocre μ2?
Deribado del análisis anterior, se ha obtenido que el elemento que más influye en la calidad del vino es el alcohol, y queremos saber si la media de alcohol de un buen vino es superior a la de un vino mediocre.
alcohol.buen.vino <- winequality.clean[winequality.clean$buen.vino == TRUE, ]$alcohol
alcohol.vino.mediocre <- winequality.clean[winequality.clean$buen.vino == FALSE, ]$alcoholDado que ambas muestras son lo suficientemente grandes como para asumir normalidad (por el Teorema Central del Límite), procedemos directamente a comprobar la homogeneidad de varianza.
var.test(alcohol.buen.vino, alcohol.vino.mediocre)##
## F test to compare two variances
##
## data: alcohol.buen.vino and alcohol.vino.mediocre
## F = 2.0594, num df = 714, denom df = 633, p-value < 2.2e-16
## alternative hypothesis: true ratio of variances is not equal to 1
## 95 percent confidence interval:
## 1.769335 2.395283
## sample estimates:
## ratio of variances
## 2.059374
Al obtener un valor p tan bajo, podemos concluir que las varianzas de ambas poblaciones son diferentes.
Vamos a realizar el cálculo del contraste de dos muestras independientes sobre la media con varianzas desconocidas diferentes.
t.test(alcohol.buen.vino, alcohol.vino.mediocre, alternative = "greater", var.equal = FALSE)##
## Welch Two Sample t-test
##
## data: alcohol.buen.vino and alcohol.vino.mediocre
## t = 18.576, df = 1277.9, p-value < 2.2e-16
## alternative hypothesis: true difference in means is greater than 0
## 95 percent confidence interval:
## 0.8765528 Inf
## sample estimates:
## mean of x mean of y
## 10.887016 9.925237
Podemos concluir, al tratarse de un test unilateral por la derecha, que el valor observado para un nivel de confianza del 95% es mayor que el valor crítico, y el valor p es menor que el nivel de significancia, por lo tanto podemos rechazar la hipótesis nula (H0) y aceptar la hipótesis alternativa (H1) de que la media de alcohol de un buen vino es mayor a la media de alcohol de un vino mediocre.
A continuación vamos a proceder a modelar la calidad de un vino en función del valor de solamente 3 de sus elementos fisioquímicos.
Para ello comenzamos con una simple regresión lineal, tomando como
variable dependiente quality y como variables explicativas alcohol,
sulphates y volatile.acidity.
regresion.multiple <- lm(quality ~ alcohol + sulphates + volatile.acidity, data = winequality.clean)
summary(regresion.multiple)##
## Call:
## lm(formula = quality ~ alcohol + sulphates + volatile.acidity,
## data = winequality.clean)
##
## Residuals:
## Min 1Q Median 3Q Max
## -2.77197 -0.37842 -0.04117 0.46167 2.14349
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 2.43662 0.21250 11.466 < 2e-16 ***
## alcohol 0.30420 0.01711 17.781 < 2e-16 ***
## sulphates 0.98370 0.12636 7.785 1.38e-14 ***
## volatile.acidity -1.18695 0.10449 -11.359 < 2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.6633 on 1345 degrees of freedom
## Multiple R-squared: 0.3515, Adjusted R-squared: 0.3501
## F-statistic: 243 on 3 and 1345 DF, p-value: < 2.2e-16
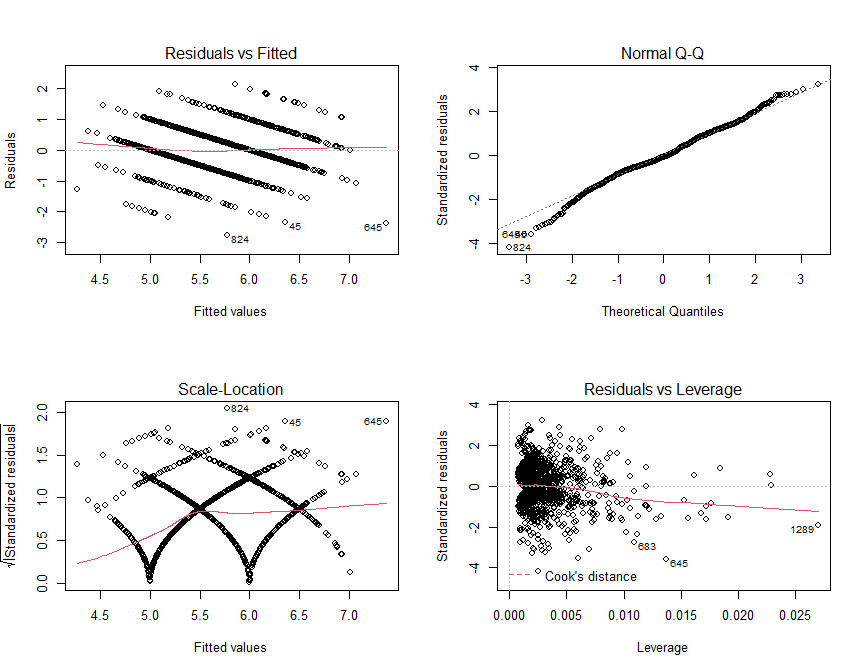
par(mfrow=c(2,2))
plot(regresion.multiple)Como podemos observar, los resultados obtenidos no son muy buenos, y
esto es debido a la distribución de la variable quality.
Para mejorar esto, vamos a proceder con una regresión logística usando
las mismas variables explicativas que para el modelo lineal, pero
tomando la variable binaria buen.vino en vez de quality.
regresion.logistica.multiple <- glm(buen.vino ~ alcohol + sulphates + volatile.acidity, data=winequality.clean, family=binomial(link=logit))
summary(regresion.logistica.multiple)##
## Call:
## glm(formula = buen.vino ~ alcohol + sulphates + volatile.acidity,
## family = binomial(link = logit), data = winequality.clean)
##
## Deviance Residuals:
## Min 1Q Median 3Q Max
## -3.3823 -0.8609 0.2973 0.8429 2.4266
##
## Coefficients:
## Estimate Std. Error z value Pr(>|z|)
## (Intercept) -10.27634 0.86338 -11.903 < 2e-16 ***
## alcohol 1.00245 0.07509 13.349 < 2e-16 ***
## sulphates 2.56287 0.45405 5.644 1.66e-08 ***
## volatile.acidity -3.04546 0.39206 -7.768 7.98e-15 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## (Dispersion parameter for binomial family taken to be 1)
##
## Null deviance: 1865.2 on 1348 degrees of freedom
## Residual deviance: 1427.8 on 1345 degrees of freedom
## AIC: 1435.8
##
## Number of Fisher Scoring iterations: 4
library(ResourceSelection)
hoslem.test(winequality.clean$buen.vino, fitted(regresion.logistica.multiple))##
## Hosmer and Lemeshow goodness of fit (GOF) test
##
## data: winequality.clean$buen.vino, fitted(regresion.logistica.multiple)
## X-squared = 3.902, df = 8, p-value = 0.8659
Un valor p alto sugiere una buena bondad de ajuste.
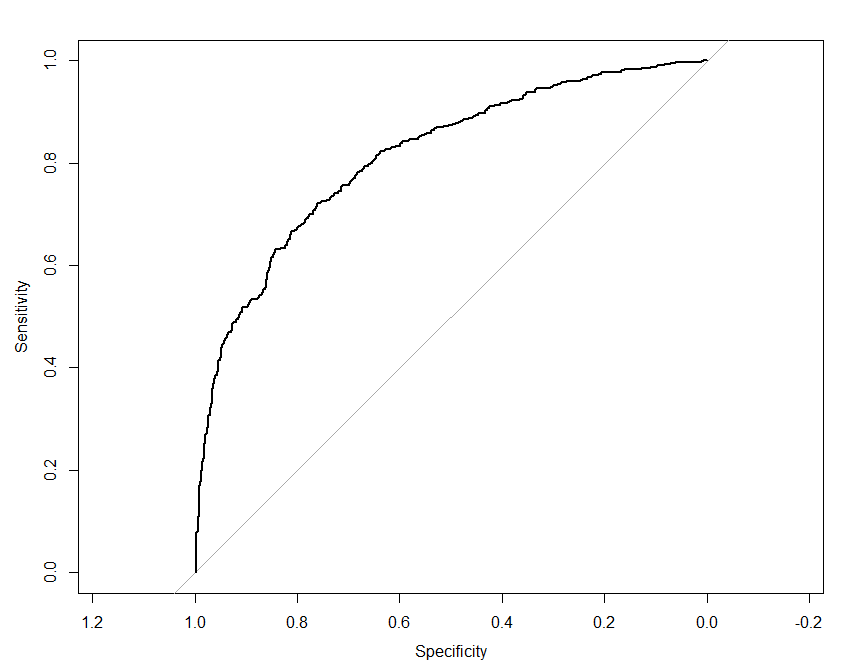
library(pROC)
prob <- regresion.logistica.multiple %>% predict(winequality.clean, type="response")
r <- roc(winequality.clean$buen.vino, prob, data = winequality.clean)
plot(r)auc(r)## Area under the curve: 0.812
El valor del área bajo la curva (AUROC) sugiere que en general el modelo discrimina de manera excelente.
Esta vez parece que el modelo es capaz de responder con una buena bondad
de ajuste y una discriminación excelente, si se trata de un buen vino o
no dados los valores de alcohol, sulphates y volatile.acidity.
Podemos incluso hacer una prediccion de los primeros 5 elementos del dataset para ver con que porcentaje nuestro modelo es capaz de predecir si se trata de un buen vino o no.
head(winequality.clean[c("alcohol", "sulphates", "volatile.acidity", "buen.vino")]) %>%
mutate(prediccion.buen.vino = predict(regresion.logistica.multiple,
data.frame(alcohol = alcohol,
sulphates = sulphates,
volatile.acidity = volatile.acidity),
type="response")
)## alcohol sulphates volatile.acidity buen.vino prediccion.buen.vino
## 1 9.4 0.56 0.70 FALSE 0.1750920
## 2 9.8 0.68 0.88 FALSE 0.1994684
## 3 9.8 0.65 0.76 FALSE 0.2495430
## 4 9.8 0.58 0.28 TRUE 0.5452182
## 6 9.4 0.56 0.66 FALSE 0.1933883
## 7 9.4 0.46 0.60 FALSE 0.1821718
Finalmente guardamos el dataset que se ha limpiado y usado para el análisis, en formato CSV.
write.csv(winequality.clean,"winequality-red-clean.csv", row.names = FALSE)A partir de los resultados obtenidos, ¿cuáles son las conclusiones? ¿Los resultados permiten responder al problema?
Dados los resultados del primer análisis de correlación pudimos extraer los 3 componentes que más afectan a la calidad del vino obteniendo como resultado el alcohol, sulfitos y acidez volátil.
Luego mediante un análisis de contraste de hipótesis pudimos responder a la pregunta de si un buen vino suele tener mayor cantidad de alcohol, y la respuesta fue afirmativa, el alcohol como elemento principal para definir la calidad de un vino, se suele encontrar en mayores niveles de éste en un buen vino que en un vino mediocre.
Y para finalizar, dados los resultados del análisis de regresión son que podemos determinar con un buen nivel de precisión si se trata de un buen vino o no, haciendo uso de solamente 3 componentes fisioquímicos.